Your new post is loading...
 Your new post is loading...
L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées. L'IPS2 est la résultante d’une importante restructuration et unification en 2015 de 3 des 4 instituts impliqués dans le Laboratoire d’Excellence / Ecole Universitaire de Recherche Sciences des Plantes de Saclay (SPS). La création de l’'IPS2 a permis de mieux structurer les activités de recherche, de formation et d’innovation en accord avec les objectifs de SPS. L’institut favorise aussi l'implication de ces équipes dans des projets innovants avec des partenaires industriels et/ou des instituts techniques, et accueille différentes start-ups, afin qu'IPS2 contribue à l’agriculture de demain. L'IPS2 héberge quatre plateformes regroupant ainsi différentes installations et différents savoir-faire dédiées aux plantes : la plateforme EPITRANS avec des activités de biologie translationnelle (grandes collections de TILLING en tomate, melon, concombre...) et d’épigénomique (analyses épigénomiques telles que DAP-seq, ATAC-seq et autres) sur les plantes cultivées; la plateforme POPS (transcriptomique) ; la plateforme Interactome (interactomique, interactions protéines-protéines) et la plateforme Métabolime-Métabolome. IPS2 : cliquer ICI Ces plateformes prennent régulièrement RDV avec vous au travers de FOCUS PLATEFORME. Profitez de cette brève pour découvrir à nouveau un éclairage sur leurs expertises, leurs équipements et leurs offres de collaborations ou de prestations ! Contacts : i) IPS2 / EPITRANS ii) IPS2 / Plateforme de transcriptOmique (POPS) iii) IPS2 / Plateforme Métabolisme Métabolome iv) IPS2 / Plateforme Interactomique (SPOmics-Interactome)
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
March 18, 2024 4:15 AM
|
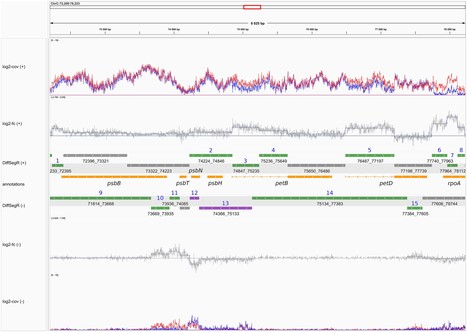
To fully understand gene regulation, it is necessary to have a thorough understanding of both the transcriptome and the enzymatic and RNA-binding activities that shape it. While many RNA-Seq-based tools have been developed to analyze the transcriptome, most of them only consider the abundance of sequencing reads along annotated patterns (such as genes). These annotations are typically incomplete, leading to errors in differential expression analyses. To address this question the OGE and GNET team from IPS2, with the support Phenoscope, the POPS platform and the LaMME, published in NAR Genomics and Bioinformatics DiffSegR, an R package that enables the discovery of transcriptome-wide expression differences between two biological conditions using RNA-Seq data. DiffSegR does not require prior annotations and uses a multiple changepoints detection algorithm to identify the boundaries of differentially expressed regions based on the per-base log2 fold change. In a few minutes of computation, DiffSegR could rightfully predict the role of chloroplast ribonuclease Mini-III in rRNA maturation and chloroplast ribonuclease PNPase in the 3′ or 5′ degradation of rRNA, mRNA, tRNA precursors, and spliced introns. We believe that DiffSegR will benefit biologists working on transcriptomics as it allows access to information from a layer of the transcriptome overlooked by classical differential expression analysis pipelines widely used today. DiffSegR is available at https://aliehrmann.github.io/DiffSegR/index.html. contact: guillem.rigaill@ips2.universite-paris-saclay.fr

|
Scooped by
Saclay Plant Sciences
March 10, 2024 4:40 PM
|
Métabolites spécialisés des plantes et agriculture"
24/04/2024 À 14H30
ANIMATEUR(S) : Loïc LEPINIECMichel DRON
Les plantes produisent de nombreux métabolites dits « spécialisés » (MS), généralement regroupés en trois grandes catégories : les terpénoïdes, les alcaloïdes et les composés phénoliques. Certains de ces composés jouent des rôles physiologiques et écologiques majeurs tout au long du cycle de vie des plantes. Ils peuvent les protéger contre des maladies, des ravageurs et des stress abiotiques ou, au contraire, faciliter leurs interactions avec des organismes bénéfiques ou symbiotiques.
Compte tenu de ces fonctions, les métabolites spécialisés suscitent un intérêt croissant pour des applications agroécologiques allant de la protection à la nutrition des plantes, en particulier dans le contexte des changements climatiques et de développement d’une agriculture plus durable utilisant moins d’intrants de synthèse. De plus, certains de ces métabolites présentent des effets positifs sur la nutrition et la santé animales et humaines, et constituent des sources de médicaments, de colorants, d'arômes ou de parfums pour l'industrie alimentaire, cosmétique ou de la santé.
Malgré leur importance, ces composés ont été caractérisés chez un nombre limité d'espèces, et la biosynthèse, le transport ou la régulation, ainsi que les fonctions biologiques de la plupart d’entre eux restent mal connus, en particulier pour des raisons techniques (ex. quantités faibles, structures chimiques complexes). De plus, il est aujourd’hui établi que la domestication puis l’amélioration des plantes ont conduit de façon intentionnelle (ex. élimination d’impacts négatifs au niveau organoleptique ou de transformation des produits) ou non (ex. sélection pour des germinations homogènes), à une diminution drastique de la quantité ou de la variabilité de ces métabolites dans les plantes et leurs produits.
Les progrès récents des connaissances de ces métabolites et le développement des nouvelles techniques d’amélioration des plantes permettent d’envisager de réintroduire ou d’optimiser les quantités de certains de ces composés de façon raisonnée dans les plantes cultivées. Ainsi, il doit être possible de bénéficier de leurs impacts positifs en agriculture et sur la qualité des produits végétaux pour des usages alimentaires ou industriels.
Introduction Loïc LEPINIEC
Exposé(s)
Métabolites spécialisés et manipulation comportementale des insectes : vers de nouvelles stratégies de contrôle des ravageurs ?
Anne-Marie CORTESERO, IGEPP, Rennes Diversité, plasticité et rôle des Métabolites spécialisés chez les graines
Massimiliano CORSO, IJPB, Versailles Les trichomes glandulaires, des usines à Métabolites spécialisés
Adnane BOUALEM, IPS2, Saclay Conclusion
Michel DRON Voir les CV des intervenants
Télécharger la présentation

|
Scooped by
Saclay Plant Sciences
March 9, 2024 1:53 PM
|
SPS Scholarships for master students Share Forward Print Academic year 2024-2025 The Saclay Plant Sciences network (SPS, www.saclayplantsciences.fr) offers scholarships for the best and most motivated students enrolled in the teaching programs in its perimeter.The teaching programs in the SPS...

|
Scooped by
Saclay Plant Sciences
February 19, 2024 5:05 AM
|
INRAE est partenaire de cet événement à portée internationale qui réunit pendant 2 jours, scientifiques, chercheurs, chefs d’entreprises, universitaires, responsables politiques, étudiants et représentants de la société civile autour de la connaissance scientifique au service du progrès et des grands enjeux environnementaux, économiques et sociétaux.

|
Scooped by
Saclay Plant Sciences
February 17, 2024 10:02 AM
|
Plant cell wall researchers were asked their view on what the major unanswered questions are in their field. This article summarises the feedback that was received from them in five questions. In this issue you can find equivalent syntheses for researchers working on bacterial, unicellular parasite and fungal systems.

|
Rescooped by
Saclay Plant Sciences
from Plant and Seed Biology
February 8, 2024 7:50 AM
|
Specialized metabolite (SM) modifications and/or decorations, corresponding to the addition or removal of functional groups (e.g. hydroxyl, methyl, glycosyl or acyl group) to SM structures, contribute to the huge diversity of structures, activities and functions of seed and plant SMs. This review summarizes available knowledge (up to 2023) on SM modifications in Brassicaceae and their contribution to SM plasticity. We give a comprehensive overview on enzymes involved in the addition or removal of these functional groups. Brassicaceae, including model (Arabidopsis thaliana) and crop (Brassica napus, Camelina sativa) plant species, present a large diversity of plant and seed SMs, which makes them valuable models to study SM modifications. In this review, particular attention is given to the environmental plasticity of SM and relative modification and/or decoration enzymes. Furthermore, a spotlight is given to SMs and related modification enzymes in seeds of Brassicaceae species. Seeds constitute a large reservoir of beneficial SMs and are one of the most important dietary sources, providing more than half of the world's intake of dietary proteins, oil and starch. The seed tissue- and stage-specific expressions of A. thaliana genes involved in SM modification are presented and discussed in the context of available literature. Given the major role in plant phytochemistry, biology and ecology, SM modifications constitute a subject of study contributing to the research and development in agroecology, pharmaceutical, cosmetics and food industrial sectors.
Via Loïc Lepiniec

|
Scooped by
Saclay Plant Sciences
January 12, 2024 4:29 AM
|
The primary objective of the 14th International Conference organized by the French Society of Plant Biology (SFBV) is to unite individuals with a keen interest in the biological characteristics of photosynthetic organisms, with a particular focus on plants. The conference will explore the practical applications of this knowledge in agriculture and bioenergy, addressing crucial issues such as global climate change and environmental preservation. At its core, the event aims to champion the comprehensive field of plant biology, encompassing both fundamental and applied aspects, while celebrating its diverse facets.
Throughout the conference, there will be a deliberate examination of plants within their environments and their intricate interactions with the surroundings. Recent advancements in: • Session introduction: Olivier Hamant « The 1972 Meadows report: A wake-up call for plant science » Round table animated by Olivier le Gall • Session 1 : Plant Imaging – Martine Pastuglia (IJPB, Versailles)
• Session 2 : Genome structure & expression – Frédy Barnèche (IBENS, Paris)
• Session 3 : Agricultural systems in the agroecological transition – To Be Announced
• Session 4 : Intra- and inter-cellular communications in plants – Fahrah Assaad (TUM, München)
• Session 5: Plant and pathogen interactions – Christine Faulkner (JIC, Norwich)
• Session 6 : Plant and beneficial microorganism interactions, microbiome – Sofie Goormachtig (VIB, Ghent)
• Session 7 : Plant development and nutrition – Bruno Guillotin (NYU, New York)
• Session 8 : Environmental stresses – Petra Marhava (UPSC, Umeå)
• Session 9 : Genomics, genetics and breeding – Alain Charcosset (GQE Le Moulon, Saclay)
• Session 10 : Plant and algae metabolism – Mark Stitt (MPI, Golm) Importantly, this gathering also seeks to initiate or uphold scientific collaborations, facilitate interactions among diverse stakeholders in the realm of plant biology research, and forge connections between public research institutions and private enterprises. Prioritizing the involvement of emerging scientists in such events is a key focus of the SFBV. In line with this commitment, scholarships will be extended to students for their participation in the 14th International Conference of the French Society of Plant Biology, with recognition through awards for outstanding oral presentations and posters. Opening subscription end of January 2024
Parution du livre international sur l'édition du génome des plantes 'A Roadmap for Plant Genome Editing' avec 34 chapitres en quatre parties (techniques, applications, réglementation, perception) chez Springer Nature ( en accès libre en pdf ou 52,74 euros pour format papier). Editors: Agnès Ricroch (editor-in-chief, Faculté Jean-Monnet, Laboratoire IDEST, UPSaclay, Sceaux), Dennis Eriksson, Dragana Miladinović, Jeremy Sweet, Katrijn Van Laere, Ewa Woźniak-Gientka. Genome Editing (GE) is a new genetic technique for improving plants so that they are more adapted to future challenges, such as increased demand for food production, climate change, use of less water, fertilizers and pesticides, and less land availability. GE involves targeted modification of genes within plants so that their expression is changed. It is a research and breeding method that directs and controls genetic mutations, and differs from transgenic genetic modification (GM) in that foreign genes are not present in the product. Using GE in plant breeding facilitates improved crop yields, enhancement of levels of beneficial nutrients, increased resistance to pathogens and pests, and higher tolerance to environmental stresses such as drought and heat. These tools can help increase the sustainability and resilience of the food systems and support the goals of the European Green Deal and the Farm to Fork Strategy. This book, written by 77 authors from 21 countries, gives an overview of the different methods of plant GE, such as Crispr-Cas and other site directed nucleases. Also, the application of GE to improve the yield, quality or resilience of different agricultural, horticultural and forestry crops is discussed. In addition, the book contains chapters describing the regulations of New Genomic Techniques (NGTs) in the European Union and many other countries and advocates appropriate science-based regulatory approaches for GE. Furthermore, societal and consumer attitudes are considered. At the book launch the chief editor Prof Agnès Ricroch says “This book will be of value to a wide audience including those wishing to develop a broader knowledge of Genome Editing as well as those wanting a deeper understanding of the science.” She also thanked the European Cooperation in Science and Technology (COST) programme for their support through the COST Action PlantEd (CA18111) and she acknowledged the contributions from numerous scientists in PlantEd and the Editorial Board. The COST Action PlantEd involved 620 scientists from organisations in 38 European and 13 non-European countries. The programme included consideration of recent advances in GE technology, novel and existing applications in crop plants, biosafety, socio-economic implications and communication issues. A more detailed description of the PlantEd COST action is available HERE.
Via Life Sciences UPSaclay
Merci à toutes et tous pour vos nombreuses contributions ! Excellente année 2024 avec toujours plus de belles découvertes dans le domaine des Sciences de la Vie !
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
December 19, 2023 9:02 AM
|
Mankind is facing an unprecedented challenge from anthropogenic climate change. This is already associated with altered rainfall patterns, extreme weather events and less predictable seasonal patterns, which are expected to increase in the future. Climate change is a major challenge to crop production, food security and therefore to human society. Agriculture is a victim of climate change but also a culprit since 20-25% of greenhouse gas (GHG) emissions are released through agricultural activities. Without a substantial diet change, the ever-growing world population will require a 70-100% increase of agricultural productivity by 2050 to produce enough food. This must be achieved in a sustainable manner without compromising nutritional quality and in a context of decreasing GHG emissions and increasing C-sequestration to help mitigate global climate change. This complex scenario will require resilient, higher yielding crops having a more efficient use of water and minerals, and suited to climate-change adapted cultivation schemes and practices. Moreover, laboratory/researcher practices will also have to be modified to accommodate low energy-use/low C-footprint research activities. In this context, the SPS-CEPLAS Summer School 2024 will focus on introducing and discussing strategies, techniques and practices to carry out next generation plant research aimed at improving sustainable climate-change friendly plant productivity. The summer school will bring together outstanding and enthusiastic young scientists (PhD students and young post-docs) and high-level researchers from all over the world in order to exchange knowledge and ideas. It is limited to a small group of participants (20 maximum) to privilege informal interactions and scientific discussions. Provisional program and speakers Download the provisional program of the Summer School https://www6.inrae.fr/saclay-plant-sciences_eng/Teaching-and-training/Summer-schools/Summer-School-2024

|
Scooped by
Saclay Plant Sciences
December 13, 2023 4:44 AM
|
CDD Ingénieur∙e d'études 12 mois (avec renouvellement potentiel)
Contexte et environnement du poste
Le réseau Sciences des Plantes de Saclay (SPS, www.saclayplantsciences.fr) représente plus de 700 personnes regroupées en une soixantaine d'équipes de recherche spécialisées dans les sciences du végétal, appartenant à cinq instituts de la région parisienne : l’Institut Jean-Pierre Bourgin (IJPB, Versailles), l’Institut de Sciences des Plantes Paris-Saclay (IPS2, Gif-sur-Yvette), l’UMR de Génétique Quantitative et Évolution - Le Moulon (GQE–Le Moulon, Gif-sur-Yvette), l’UMR Biologie et Gestion des Risques en Agriculture (BIOGER, Palaiseau) et l’Institut de Biologie Intégrative de la Cellule (I2BC, Gif-sur-Yvette). Les activités de recherche des équipes SPS portent sur la compréhension des mécanismes génétiques, moléculaires et cellulaires qui contrôlent le développement et la physiologie des plantes, et leurs interactions avec l’environnement biotique et abiotique.
Le réseau SPS s’est initialement constitué dans le cadre d’un projet de Laboratoire d’Excellence (LabEx, Programme Investissements d’avenir, PIA1), financé à hauteur de 12 M€ pour 9 ans (2011-2019). En 2017, le réseau a obtenu un nouveau financement de 16 M€ sur 10 ans (2018-2028) à travers le programme « Ecoles Universitaires de Recherche » (EUR, PIA3). Dans le cadre de ce nouveau projet, SPS a notamment pour but de renforcer les liens entre formation et recherche en sciences végétales dans l’environnement Paris-Saclay, à travers les formations de niveau master, les écoles doctorales et les équipes de recherche partenaires du projet.
Missions / Activités principales
Sous la responsabilité de la manager SPS et du Comité Directeur, et en relation avec le Comité de Pilotage, les différents groupes de travail, ainsi que les équipes scientifiques, administratives, financières et de communication des laboratoires et tutelles impliqués, vous viendrez en appui à la manager SPS pour la gestion opérationnelle de l’EUR SPS.
Date limite d’envoi des candidatures pour les premiers entretiens : 15 janvier 2024 (minuit)
Plus d'informations et modalités de candidatures : Page web de l'offre

|
Scooped by
Saclay Plant Sciences
November 25, 2023 5:23 AM
|
|

|
Scooped by
Saclay Plant Sciences
March 18, 2024 9:39 AM
|

|
Scooped by
Saclay Plant Sciences
March 15, 2024 12:22 PM
|
Plant Modeling: Opportunities and Challenges"
Early bird registration has been moved to March 29th!
The Jean-Pierre Bourgin Institute, a major laboratory for plant science research affiliated to both INRAE and AgroParisTech, is organising the second edition of its international symposium. Following on the first edition in 2018, this will take place from the 13th to 15th of May 2024 at the INRAE Centre in Versailles.

|
Scooped by
Saclay Plant Sciences
March 9, 2024 2:58 PM
|
La plateforme de cytologie et imagerie (Institut Jean Pierre Bourgin, centre INRAE de Versailles-Grignon), est une plateforme couvrant différents aspects de la microscopie, de l’observation en fond clair à l’analyse poussée de la fluorescence en microscopie confocale dont l’activité est centrée autour des échantillons végétaux. La plateforme offre un ensemble de microscopes, d’appareils de préparation des échantillons nécessaire pour l’observation et de compétences nécessaires. Elle est également partie intégrante d’un ensemble plus vaste, l’Observatoire du Végétal, qui regroupe des infrastructures allant de la culture à l’observation et l’analyse des plantes (phénotypage multiniveaux), ensemble que vous découvrirez aussi en un clic via son FOCUS PLATEFORME : IJPB / Observatoire du Végétal : 6 plateformes au service du végétal, mais pas que !. Quoi de neuf sur la plateforme ? La plateforme de cytologie et imagerie accueille depuis fin 2023 un tout nouveau microscope permettant l’imagerie super résolution de type dSTORM en 3D et multi-couleur. Obtenu dans le cadre de l’ERC STORMtheWALL (Kalina Haas, IJPB-INRAE, Versailles), cet équipement permettra le suivi de structure 3D de molécules uniques et de dynamiques cellulaires. La microscopie de fluorescence reste incapable de résoudre deux points distants de moins de 250 à 300 nm alors que la technologie STORM, basée sur la réitération de plusieurs étapes de photoactivation/photoblanchiment permet une résolution spatiale de 15 nm. Cette approche est utilisée pour étudier la nanostructure de la paroi végétale ainsi que les autres structures subcelullaires. Mais en fait l’équipement est bien plus complexe ! Il possède deux modules différents : le sFLIM et la photostimulation qui permettent de contrôler et d’observer simultanément des processus biochimiques et moléculaires dynamiques dans des plantes vivantes en utilisant en parallèle un activateur photoactivable (optogénétique) et plusieurs biocapteurs FRET. Couplé à des détecteurs très sensibles, le système permet l’imagerie spectrale “temps de vie” (sFLIM), en utilisant une technique de comptage de photons dans une gamme spectrale allant de 430 à 650 nm (16 canaux), même en faible luminosité. Cet équipement permet de dépasser les limites spatiales et temporelles habituelles et ouvre une nouvelle dimension pour la compréhension de mécanismes cellulaires à l'échelle nanométrique. Il fournira des données inédites pour la mise en place de modèles de processus biologiques. Ces modèles sont des briques essentielles des modèles multi-échelles prédisant la croissance et le rendement des cultures dans des environnements changeants. Contact : Bertrand DUBREUCQ (bertrand.dubreucq@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, le 15 février 2021 et le 6 décembre 2021, la plateforme de Cytologie et Imagerie publiait ses premiers Scoop-it® / FOCUS PLATEFORME ! Les relire à nouveau ? La microdissection assistée par laser, un apport « tranchant » dans l’analyse à très petite échelle des transcriptomes végétaux ; Vers de nouveaux fronts de science avec un microscope à super résolution à technologie STED dès 2022 ! La plateforme de Cytologie et Imagerie est une plateforme adossée à une très grosse unité (Institut Jean Pierre Bourgin, 350 personnes) et intégrée dans un ensemble plus vaste allant de la culture des plantes à l’observation finale ou l’analyse métabolique (Observatoire du Végétal). La plateforme a été pionnière dans le développement de nouvelles techniques d’étude du végétal notamment dans l’étude du matériel vivant (caractérisation du développement du méristème vivant, interaction entre protéines par la technique d’anisotropie de fluorescence chez les végétaux). La plateforme compte 8 permanents INRAE (5 ETP) et regroupe les activités et les équipements d'imagerie cellulaire nécessaires à l’étude des plantes : micro et macroscopie, microscopie confocale, vidéomicroscopie, cytométrie, microscopie électronique à balayage, microdissection laser, ainsi que toutes les ressources pour le traitement, l’inclusion et la coupe des échantillons (coupe semi fines, fines, ultrafines, cryosections). Elle est hébergée dans un bâtiment complètement rénové de 1000 m2 accueillant la trentaine de pièces optiques et les données produites par la plateforme sont hébergées sur un serveur institutionnel dont le contenu est sécurisé et sauvegardé. La plateforme est ouverte à toutes les demandes de l’unité et hors unité, institutionnelles ou du secteur privé. La structure est labellisée IBISA.
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
February 17, 2024 10:07 AM
|
Carbon (C) and nitrogen (N) metabolisms are tightly integrated to allow proper plant growth and development. Photosynthesis is dependent on N invested in chlorophylls, enzymes, and structural components of the photo- synthetic machinery, while N uptake and assimilation rely on ATP, reducing equivalents, and C-skeletons pro- vided by photosynthesis. The direct connection between N availability and photosynthetic efficiency allows the synthesis of precursors for all metabolites and building blocks in plants. Thus, the capacity to sense and respond to sudden changes in C and N availability is crucial for plant survival and is mediated by complex yet efficient signaling pathways such as TARGET OF RAPAMYCIN (TOR) and SUCROSE-NON-FERMENTING-1- RELATED PROTEIN KINASE 1 (SnRK1). In this review, we present recent advances in mechanisms involved in sensing C and N status as well as identifying current gaps in our understanding. We finally attempt to provide new perspectives and hypotheses on the interconnection of diverse signaling pathways that will allow us to understand the integration and orchestration of the major players governing the regulation of the CN balance.

|
Scooped by
Saclay Plant Sciences
February 9, 2024 1:45 PM
|
How defects in the Arabidopsis secretory pathway lead to autoimmunity (IPS2, SPS)
Figure legend: Representative pictures of 35-day-old plants showing the involvement of salicylic acid signalling in the snap33 mutant phenotype.

|
Rescooped by
Saclay Plant Sciences
from Plant and Seed Biology
February 7, 2024 8:59 AM
|
For a more sustainable and resilient food system, MEPs support a simpler process for NGT plants equivalent to conventional plants, while others must follow stricter rules.
Via Loïc Lepiniec
La Plateforme de Cytologie/Imagerie de l’Observatoire du Végétal de l’Institut Jean-Pierre Bourgin – IJPB (NRAE/AgroParisTech/UPSaclay, Versailles) vient de faire l’acquisition d’un microscope à super-résolution STORM. Ce microscope ouvre une nouvelle dimension pour la compréhension des mécanismes cellulaires et fournira des données inédites, essentielles à la mise en place de modèles multi-échelles visant à prédire la croissance et le rendement des cultures dans des environnements changeants. Plus d’informations
Via Life Sciences UPSaclay
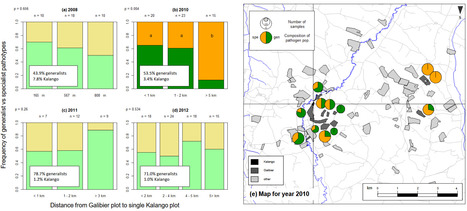
Dans une étude publiée récemment dans Scientific Reports, des scientifiques des unités BIOGER (INRAE/UPSaclay, Campus Agro Paris-Saclay, INRAE Palaiseau), BioSP (Avignon) et d’AgroParisTech (Palaiseau) ont étudié la composition génétique des populations de rouille brune présentes sur repousses de blé à l’échelle d’un paysage. Le déploiement de variétés résistantes est un moyen efficace pour gérer les maladies telles que la rouille brune, causée par Puccinia triticina. Après la récolte, ce champignon biotrophe peut survivre sur des repousses de blé jusqu’à la saison culturale suivante. Les auteurs ont caractérisé la diversité de populations de P. triticina collectées sur des repousses de blé durant six années (2007-2012) à l’échelle du paysage. Un total de 642 isolats de rouille brune, collectés dans une zone d’un rayon de 5 km, ont été classés selon 52 profils de virulence (pathotypes). Cette étude a révélé une diversité élevée des populations de P. triticina sur repousses en termes de composition en pathotypes (identité et abondance). Cette diversité a pu être mise en relation avec la variété d’origine des isolats, et analysée en fonction de la distance entre les parcelles échantillonnées. Les auteurs ont observé un impact de la variété d’origine sur la composition des populations pathogènes. Les niveaux de diversité de populations ont différé entre variétés et selon leur niveau de déploiement dans la zone d’étude. Ces résultats suggèrent que les repousses de blé pourraient contribuer significativement à la composition de l’inoculum primaire et à l’initiation des épidémies de rouille brune. Contact : tiphaine.vidal@inrae.fr
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
January 3, 2024 1:01 PM
|
The Saclay Plant Sciences network (SPS) wish you a Happy New Year 2024 !
The Saclay Plant Sciences network (SPS) wish you a Happy New Year 2024 !

|
Scooped by
Saclay Plant Sciences
December 16, 2023 4:22 AM
|
Le but de cette manifestation est de faire en sorte que le plus grand nombre de personnes autour du globe soient fascinées par les plantes et conscientes de l’importance des sciences végétales pour l’agriculture et la production durable de nourriture, ainsi que pour l’horticulture, la foresterie et des productions non alimentaires à base de plantes, telles que le papier, le bois de construction, l’énergie et des molécules utiles à l’industrie chimique et pharmaceutique. Le rôle des plantes dans la conservation de l’environnement est également un message clé.

|
Scooped by
Saclay Plant Sciences
November 25, 2023 3:26 PM
|
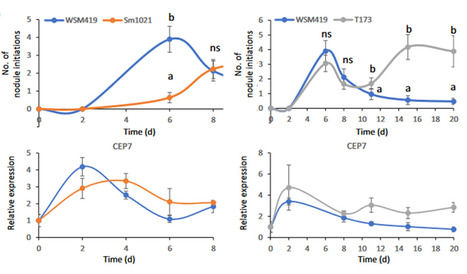
Rhizobium symbiotic efficiency meets CEP signaling peptides (IPS2, SPS) CEP (C-terminally Encoded Peptides) signaling peptides are drivers of systemic pathways regulating Nitrogen (N) acquisition in different plants, from Arabidopsis to legumes, depending on mineral N availability (e.g. nitrate) and on the whole plant N demand. Recent studies in the Medicago truncatula model legume revealed how root-produced CEP peptides control the root competence for endosymbiosis with N fixing rhizobia soil bacteria through the activity of the CRA2 (Compact Root Architecture) CEP receptor in shoots. Among CEP genes, MtCEP7 was shown to be tightly linked to nodulation. The team SILEG at IPS2 recently reported in New Phytologist that the dynamic temporal regulation of MtCEP7 expression reflects the plant ability to maintain a different symbiotic root competence window depending on the symbiotic efficiency of the rhizobium strain, as well as to reinitiate a new window of root competence for nodulation. Contact: florian.frugier@universite-paris-saclay.fr
|


 Your new post is loading...
Your new post is loading...