Your new post is loading...
 Your new post is loading...

|
Scooped by
Saclay Plant Sciences
September 13, 2024 8:25 AM
|
- L’AFBV organise son 13ème colloque le jeudi 10 octobre2024 à l’Académie d’Agriculture de France, 18 Rue de Bellechasse, 75007 Paris, sur le thème : La Domestication des plantes :des premiers agriculteurs aux biotechnologies Tarifs Programme

|
Rescooped by
Saclay Plant Sciences
from Plant and Seed Biology
September 8, 2024 4:39 AM
|
Environmental impacts of genetically modified crops
INTRODUCTION In recent decades, genetically modified (GM) crops have been widely adopted by some of the world’s leading agricultural nations, but the full extent of their environmental impacts remains largely unknown. Initial studies conducted in controlled environments provided valuable insights into the mechanisms that relate GM crops to the environment but offered an incomplete picture of their indirect environmental consequences. Adopting GM crops often leads to changes in other agricultural practices, such as pesticide use, cropping, and tillage patterns, with profound environmental implications (see figure). Furthermore, with widespread adoption, these changes become large in scale and can lead to substantial economic and ecological spillovers through markets and ecological interactions, influencing the environmental outcomes of nonadopting regions. These spillovers can also lead to opposing outcomes in adopting and nonadopting regions. For example, increased profits from GM crop adoption can incentivize agricultural expansion and intensification in adopting countries with potentially negative environmental impacts. However, the increased global supply from these changes in adopting countries can reduce expansion and intensification in nonadopting countries through market effects with potentially positive environmental implications. The goal of our review is to synthesize the recent understanding of the environmental implications of GM crop adoption, with a particular focus on indirect effects and spatial spillovers. Much of the existing literature focuses on the direct effects of GM crop adoption on agricultural outcomes and, to a lesser extent, its implications for the environment and human health. Recent studies employing causal inference methods have contributed to our understanding of the combined direct and indirect impacts of GM crop adoption through environmental management changes, including effects on yields, deforestation, biodiversity, and human health. Their findings paint a nuanced picture of GM crop adoption, with mostly positive impacts on yields and mixed effects on pesticide use, biodiversity, deforestation, and human health. These studies also find negative health effects from increased glyphosate use, possibly counteracted by reduced toxicity of insecticide applications due to the adoption of insect-resistant GM crops. However, the few studies that evaluate long-term consequences suggest that short-term benefits may decrease if pest resistance is not well managed. In addition, new results show that GM crop adoption increases deforestation locally, in contrast to previous studies that found a land-sparing global effect of GM crop adoption. Lastly, the evidence for the impacts of GM crop adoption on biodiversity is mixed. For example, a recent study found that although the overall impact of GM crops on bird diversity is small, the overall effect is composed of positive effects on insectivorous species and negative effects on plant and seed-eating species. While these studies greatly advance our understanding of the direct and indirect environmental effects of GM crops, they still do not fully assess the spillover effects on areas and regions that have not adopted GM crops. OUTLOOK To date, large-scale GM crop adoption has been limited to just two traits with different environmental and human health effects: herbicide tolerance and insect resistance. New GM crops and gene-edited versions with different traits, such as drought resistance, would likely have different environmental and human health impacts. Therefore, improving regulations and resistance management, providing incentives for the development and commercialization of new traits that align with social goals and human welfare, and gathering more comprehensive and detailed environmental data, especially on biodiversity, are critical for guiding agricultural innovations toward greater sustainability and allowing an accurate assessment of their impacts.
Via Loïc Lepiniec

|
Rescooped by
Saclay Plant Sciences
from Plant and Seed Biology
September 8, 2024 4:38 AM
|
Apply to organise a Concurrent Session or Workshop at ICAR2025 At ICAR2025 there are 18 Concurrent Sessions and 8 Workshop sessions (both 90minutes in length). The topics of these sessions will be decided by community applications. In general workshop sessions maybe be more 'interactive' than Concurrent sessions, although this distinction is somewhat blurred. Organisers can arrange sessions however they would like with regard career-stage and number of speakers. Importantly we strongly encourage organisers to consider all aspects of diversity when selecting speakers. In addition to suggesting invited speaker(s) the organisers will be provided with all submitted abstracts after the ICAR2025 abstract submission deadline in early 2025. Organisers then have the option of inviting other people to participant in their session. Please submit your application before October 21st. The Scientific Organising Committee will consider applications soon after the time and we will announce the selected sessions by December 1st. Concurrent session organisers will receive €1000 to support the session. The organisers have full control over how this finance is spent; whether it is on meeting-registrations or travel (either for organisers or participants). If you have any questions about the application process please contact Geraint Parry (geraint@aab.org,.uk). Submit your application here
Via Loïc Lepiniec
Les ressources biologiques et le concept "One Health" Du Mardi 24 septembre (14h00) au mercredi 25 septembre (12h00), à l’hôtel IBIS Style Paris-Bercy et en webinaire L’infrastructure de recherche RARe a le plaisir de vous inviter à son 4e séminaire scientifique. Le programme du séminaire a été conçu pour aborder les différentes dimensions du "One Health" avec des exemples pris dans tous les domaines couverts par RARe. Il est destiné à la communauté scientifique française, chercheurs et enseignants, partenaires socio-économiques, infrastructures de recherche, animateurs de programmes multidisciplinaires, décideurs. Ce séminaire visera d’une part à illustrer les relations complexes existant entre les composantes biotiques et abiotiques des écosystèmes, et d’autre part à montrer l’avantage de disposer d’une infrastructure telle que RARe pour susciter de nouveaux projets valorisant la diversité de ses collections. Thèmes : 1- Ecologie évolutive et prévention des zoonoses
2- Santé des écosystèmes et santé des populations
3- Diversité génétique et interactions hôtes-pathogènes
4- Relations entre production et contamination
5- Les bibliothèques du vivant et leur complémentarité avec la diversité sur le terrain
6- Restauration des écosystèmes dégradés à l’échelle du paysage Avec deux interventions de collègues de BIOGER (INRAE/UPSaclay, Campus Agro Paris-Saclay, Palaiseau) : - Tiphaine Vidal présentera "La résistance durable à la rouille du blé en France"
- Florence Carpentier présentera "Co-occurrence des maladies en systèmes Céréaliers : déterminants, rôle et gestion de la diversité cultivée et sauvage de la parcelle au paysage"
Plus d'informations et lien vers formulaire d'inscription
Via Life Sciences UPSaclay
Si nous commençons à comprendre les mécanismes de réponse des plantes aux contraintes de leur environnement, cela passe souvent par l'étude de stress individuels, sans nécessairement aborder les interactions majeures qui apparaissent dans la nature ou au champ, où les stress se combinent dans une dynamique complexe. Il est par exemple pertinent d’envisager la question de la disponibilité en eau lorsqu'on étudie un stress nutritionnel. Dans une étude publiée dans The Plant Cell, plusieurs scientifiques de l’Institut Jean-Pierre Bourgin – IJPB (INRAE/AgroParisTech/UPSaclay, Versailles), épaulés par la Plateforme de transcriptomique (POPS) à l’IPS2, ont collaboré pour révéler l'étendue de la diversité naturelle existant chez l'espèce Arabidopsis thaliana en réponse au stress hydrique et à la déficience en azote, seuls ou combinés. Ce travail de caractérisation multi-omique (phénomique, transcriptomique et métabolomique) montre à quel point il est difficile de définir une 'réponse universelle' chez cette espèce : les groupes de traits qui discriminent le mieux les réponses au stress sont différents d'une accession (variété) à l'autre. La principale interaction entre les stress a eu pour effet d'atténuer le syndrome lié au stress azoté lorsque combiné à un stress hydrique modéré. De multiples autres interactions eau x azote sont révélées et celles-ci sont en général spécifiques à chaque accession d'Arabidopsis étudiée. Lire aussi l’Actu IJPB Contact : olivier.loudet@inrae.fr
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
July 4, 2024 4:24 PM
|
The SPS-CEPLAS Summer School 2024, united young scientists and researchers to address climate change through plant science. The Global Plant Council led a workshop on science communication for emphasizing the importance of conveying complex research to broader audiences effectively.

|
Scooped by
Saclay Plant Sciences
July 4, 2024 2:23 AM
|
Photosystem I (PSI) is an essential protein complex for oxygenic photosynthesis and is also known to be an important source of reactive oxygen species (ROS) in the light. When ROS are generated within PSI, the photosystem can be damaged. The so-called PSI photoinhibition is a lethal event for oxygenic phototrophs, and it is prevented by keeping the reaction center chlorophyll (P700) oxidized in excess light conditions. Whereas regulatory mechanisms for controlling P700 oxidation have been discovered already, the molecular mechanism of PSI photoinhibition is still unclear. Here, we characterized the damage mechanism of PSI photoinhibition by in vitro transient absorption and electron paramagnetic resonance (EPR) spectroscopy in isolated PSI from cucumber leaves. Photodamage to PSI was induced by two different light treatments: 1. continuous illumination with high light at low (chilling) temperature (C/LT) and 2. repetitive flashes at room temperature (F/RT). These samples were compared to samples that had been illuminated with high light at room temperature (C/RT). The [FeS] clusters FX and (FA FB) were destructed in C/LT but not in F/RT. Transient absorption spectroscopy indicated that half of the charge separation was impaired in F/RT, however, low-temperature EPR revealed the light-induced FX signal at a similar size as in the case of C/RT. This indicates that the two branches of electron transfer in PSI were affected differently. Electron transfer at the A-branch was inhibited in F/RT and also partially in C/LT, while the B-branch remained active.

|
Scooped by
Saclay Plant Sciences
June 30, 2024 4:54 PM
|
Plants use a combination of sophisticated local and systemic pathways to optimize growth depending on heterogeneous nutrient availability in the soil. Legume plants can acquire mineral nitrogen (N) either through their roots or via a symbiotic interaction with N-fixing rhizobia bacteria housed in so-called root nodules. To identify shoot-to-root systemic signals acting in Medicago truncatula plants at N-deficit or N-satiety, plants were grown in a split-root experimental design, in which either high or low N was provided to a half of the root system, allowing the analysis of systemic pathways independently of any local N response. Among the plant hormone families analyzed, the cytokinin trans-Zeatin accumulated in plants at N-satiety. Cytokinin application by petiole feeding led to an inhibition of both root growth and nodulation. In addition, an exhaustive analysis of miRNAs revealed that miR2111 accumulates systemically under N-deficit in both shoots and non-treated distant roots, whereas a miRNA related to inorganic Phosphate (Pi)-acquisition, the miR399, does so in plants grown at N-satiety. These two accumulation patterns are dependent on CRA2 (Compact Root Architecture 2), a receptor required for CEP (C-terminally Encoded Peptide) signaling. Constitutive ectopic expression of the miR399 reduced nodule numbers and root biomass depending on Pi availability, suggesting that the miR399-dependent Pi-acquisition regulatory module controlled by N-availability affects the development of the whole legume plant root system.
L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. Cet institut héberge quatre plateformes (En savoir plus ?) regroupant ainsi différentes installations et différents savoir-faire dédiées aux plantes. Dans le présent FOCUS PLATEFORME … c’est au tour de la plateforme POPS (transcriptomique) de nous faire découvrir son expertise. Les plantes mixotrophes sont capables de produire leur énergie non seulement via la photosynthèse mais aussi en trompant leurs champignons mycorhiziens symbiotiques pour obtenir des nutriments. Elles peuvent ainsi ajuster leurs sources énergétiques en fonction des conditions environnementales. Cependant, malgré cet avantage écologique certain, les plantes mixotrophes restent relativement rares dans l’histoire de l’évolution. Pour mieux comprendre ce paradoxe, le MNHN de Paris et l’Université de Gdansk en Pologne ont mené une étude portant sur neuf espèces d’orchidées européennes. En étudiant le transcriptome et le métabolome de ces espèces in situ, ils ont révélé que la plasticité permise par la mixotrophie impose des contraintes spécifiques qui pourraient limiter leur adaptabilité. Ainsi, l’étude du contenu en gènes a révélé un ensemble de gènes spécifiques des orchidées mixotrophes suggérant que la transition de la mixotrophie depuis l’autotrophie impose l’acquisition de fonctions spécifiques. De plus, la physiologie des orchidées mixotrophes montre des contraintes telles qu’une capacité limitée à assimiler le tréhalose fongique ou à se passer totalement de photosynthèse. Enfin, l’étude des réseaux de métabolites suggère que les orchidées mixotrophes ont des capacités adaptatives plus limitées. Ces résultats sont importants pour comprendre la dynamique évolutive des réseaux mycorhiziens. En effet, les plantes parasitant ces réseaux (les espèces mixotrophes et mycohétérotrophes) constituent des menaces à leur stabilité et à leur résilience. Les contraintes liées à l’apparition de ces plantes limitent cette menace et permettent aux champignons mycorhiziens généralistes de se maintenir. Par ce travail qui se poursuit encore actuellement avec l’étude du développement de ces espèces, la plateforme POPS démontre sa capacité à accompagner ses collaborateurs dans des projets « hors du laboratoire » depuis le design expérimental jusqu’à l’interprétation des données produites. Contact : pops.ips2@universite-paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en février 2020 et juin 2023, POPS publiait ses premiers FOCUS PLATEFORME … Redécouvrez- les ! FOCUS PLATEFORME : L'analyse transcriptomique passe au naturel! ; FOCUS PLATEFORME : IPS2 / POPS : Au cœur de la défense des plantes contre les bactéries ! IPS2 / Plateforme de transcriptomique (POPS). La Plateforme de Transcriptomique des Plantes de Paris-Saclay (POPS) propose aux laboratoires académiques ou privés des outils d'analyse du transcriptome par séquençage à haut débit de l'ARN ou RNA-seq. Nous sommes spécialisés dans l'analyse des plantes qu'elles soient modèles ou de culture. Le service proposé inclue la génération des données de RNA-seq, les analyses bio-informatiques et statistiques ainsi que des conseils sur l'interprétation des résultats. Nous accompagnons donc nos collaborateurs depuis le design expérimental jusqu'à l'interprétation des données. La plateforme est certifiée pour l'ensemble de ses activités selon les exigences de la norme ISO 9001 depuis 2012. Afin d'offrir à chaque collaborateur l'analyse la plus adaptée à ses questions biologiques, nous disposons des équipements et des outils d'analyses bio-informatiques et statistiques permettant la construction de transcriptome de novo, l'analyse d'accumulation des ARNs poly-adenylés ou non, des petits ARNs (miRNAs, siRNAs, ribo-seq..) et des ARN poly-adénylés de très petite quantité de départ (jusqu'à 50pg). Pour les analyses d'expression tissu-spécifiques, nous offrons également des analyses de transcriptome après micro-dissection laser ou en cellule unique grâce à la technologie Chromium (10x Genomics). Par ailleurs, nous pouvons offrir à nos collaborateurs de sessions de formation sur une semaine à l'analyse statistique des données RNA-seq. A propos de l’IPS2. L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées.
Via Life Sciences UPSaclay
Cette première version actualisée en anglais de l’ouvrage « Chimie verte et industries agroalimentaires – Vers une bioéconomie durable » présente un kaléidoscope de visions sur la bioéconomie. Outre l'accent mis sur les innovations, il fournit de nombreuses définitions de termes et de concepts. L'objectif de ce livre est de montrer la complémentarité et l'intégration des chaînes de valeur alimentaires et non alimentaires pour le développement d'une bioéconomie durable. Un défi actuel auquel font face l'industrie et l'économie est de répondre aux besoins d'une population mondiale croissante tout en préservant l'environnement. L'utilisation des ressources énergétiques fossiles pendant plusieurs décennies a entraîné une diminution des réserves de ces ressources, ainsi qu'un phénomène de réchauffement climatique dû à l'émission de gaz à effet de serre dans l'atmosphère. De plus en plus de secteurs industriels, y compris l'industrie chimique, remplacent le carbone fossile par du carbone renouvelable. La bioéconomie consiste à utiliser des ressources biologiques renouvelables pour produire des aliments, des matériaux et de l'énergie. Une bioéconomie basée sur la chimie verte et les biotechnologies se développe dans le monde entier, comme levier pour réduire l'empreinte écologique des activités humaines. Le livre est articulé autour de six parties, chacune dédiée à une pierre angulaire de l'interface entre la chimie verte et l'industrie agroalimentaire. Table des matières Éditrice : Stéphanie Baumberger, contact, équipe "Biopolymères Lignocellulosiques : des assemblages pariétaux aux synthons pour la chimie verte" APSYNTH A propos de l'éditrice En relation avec une recherche développée à l’Institut Jean-Pierre Bourgin - Sciences du Végétal.
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
April 21, 2024 3:52 PM
|
Highlights - The nuclear proteostasis network is spatially organized, safeguarding nuclear functions during adverse conditions.
- Nuclear membrane-less organelles (MLOs) are crucibles for nuclear protein quality control (PQC), concentrating both aberrant proteins and PQC effectors upon proteotoxic stress.
- The properties of the nucleolus provide multiple PQC functions and mitigate the toxicity of aberrant proteins.
- Transient storage into MLOs preserves the nuclear proteostasis network and allows efficient recovery of nuclear functions when the stress subsides.
- The nucleus contains a number of non-canonical chaperones reflecting the specificity of this compartment.
Les modifications de la chromatine, en particulier l'acétylation des histones, jouent un rôle important dans la régulation de l'expression des gènes en réponse aux changements environnementaux. L'acétylation des histones est une modification covalente par l'histone acétyltransférase (HAT), avec l'acétyl-CoA comme substrat. Outre les histones, un grand nombre de protéines impliquées dans de différentes voies cellulaires subissent également des modifications par acétylation. L'acétyl-CoA utilisé pour l'acétylation des protéines est principalement dérivé du métabolisme des sucres. Des études antérieures ont montré que le niveau de métabolisme énergétique cellulaire affecte les modifications de la chromatine et l'expression des gènes associés. Par exemple, lorsque les niveaux d’énergie des cellules sont élevés, le degré d’acétylation de la chromatine augmente, stimulant ainsi l’expression des gènes liés à la prolifération cellulaire et favorisant la division cellulaire. En revanche, dans des environnements de stress où les niveaux d'énergie cellulaire diminuent, l’homéostasie de l'acétylation des histones est maintenue, même avec des niveaux d’acétylation augmentés sur des gènes liés au stress. Actuellement, le mécanisme sous-jacent reste flou. GCN5 est une HAT principale dans le noyau des cellules végétales. Cette enzyme peut former le complexe dit « SAGA » avec des protéines adaptatrices telle qu’ADA2. ADA2 peut renforcer la capture de l'acétyl-CoA par la HAT GCN5 pour acétyler les histones dans la chromatine. Dans une étude publiée dans Nature Plants dirigée par le Professeur Dao-Xiu Zhou à l’IPS2 (CNRS/INRAE/UPSaclay/UEVE/UParis, Gif-sur-Yvette), et réalisée en collaboration avec la Huazhong Agriculture University (Chine), les auteurs ont trouvé que GCN5 peut aussi acétyler plusieurs résidus lysines de la protéine ADA2. L'acétylation d’ADA2 affecte sa stabilité et son accumulation. En fait, ADA2 acétylée est ciblée spécifiquement par une ubiquitine ligase E3, conduisant à sa dégradation. Ensuite, les auteurs ont découvert qu’ADA2 est acétylé lorsque le niveau cellulaire de l’acétyl-CoA est élevé, donc moins accumulé ; mais en condition de stress ou le niveau du cofacteur est bas en raison de la limitation du métabolisme énergétique, ADA2 est moins acétylée mais plus accumulée dans la cellule. Il a été constaté que quand le niveau d'acétyl-CoA est élevé, l'activité de GCN5 pour acétyler les histones ne dépend pas d’ADA2 ; mais quand le niveau d’acétyl-CoA est faible notamment en condition de stress, ADA2 est nécessaire pour maintenir l’activité de GCN5. Cela a été observé in vitro et in vivo dans des plantes sauvage et mutantes, cultivées dans de différentes conditions. Ces résultats indiquent que l'acétylation d’ADA2 qui détecte les variations de l'acétyl-CoA cellulaire est un mécanisme pour réguler l’activité de GCN5 afin de maintenir homéostasie de l’acétylation des histones chez les plantes dont les conditions de croissance sont changeantes. Contact : dao-xiu.zhou@université.Paris-saclay.fr
Via Life Sciences UPSaclay
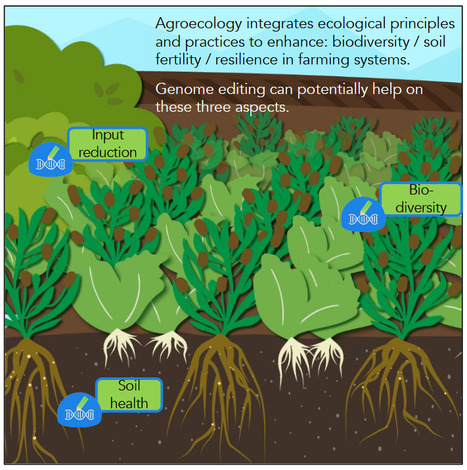
Dans une revue publiée dans iScience des chercheurs de différentes unités d'INRAE, dont l’Institut Jean-Pierre Bourgin – IJPB (INRAE/AgroParisTech/UPSaclay, Versailles), font le point sur l'intérêt potentiel des technologies d'édition du génome pour la transition agroécologique. Pour relever les défis de la transition agroécologique dans un contexte de changement climatique, il est nécessaire d'utiliser diverses stratégies telles que les régulations biologiques, les génotypes animaux et végétaux adaptés, les systèmes de production diversifiés et les technologies numériques. Les semences et les plantes, par le biais de la sélection végétale, jouent un rôle crucial dans la conduite de ces changements. L'émergence de l'édition du génome présente une nouvelle opportunité dans les pratiques de sélection végétale. Cependant, comme toute révolution technologique impliquant des organismes vivants, il est essentiel d'évaluer ses contributions potentielles, ses limites, ses risques, ses implications socio-économiques et les controverses associées. Cet article vise à fournir une revue complète des connaissances scientifiques sur l'édition du génome pour la transition agroécologique, en s'appuyant sur des approches multidisciplinaires englobant les sciences biologiques, agronomiques, économiques et sociales. Contact : fabien.nogue@inrae.fr
Via Life Sciences UPSaclay
|

|
Scooped by
Saclay Plant Sciences
September 11, 2024 2:05 AM
|
Voir la version en ligne Le Mensuel N°96 / Septembre 2024 À LA UNE Photographies de racines en laboratoire.

|
Rescooped by
Saclay Plant Sciences
from Plant and Seed Biology
September 8, 2024 4:39 AM
|
E-book Beyond GMO Welcome to SPPS' download site! The translation of the swedish book " Bortom GMO" has been sponsored by SPPS and Anke Carius (Society Secretary) has done the layout. Some images and graphics have been updated and modernized by DC Sci Art. Check out her work if you ever need any illustrations for your publications or presentations. In this book, you can read all about genetically modified plants, in an educative manner. You do not need to be a scientist to understand it but if you are, you will enjoy the chapters about their impact on economy, society and politics, how they are viewed internationally, and the comparison of the current international law. The e-book is free to download, but please respect the copyright is owned by the authors, so please cite properly and if you would like to use images or graphics from the book, ASK (you might even be provided with a version with better resolution )! If you would like to support initiatives like this and are a plant scientist, please check out our Journal "Physiologia Plantarum" when you choose where to publish your work next time. Everyone who would like to support SPPS can become a member. Click "Join" on the panel to the left. Thank you! Enjoy the read! Here you can download the free e-book by Stefan Jansson, Roland von Bothmer and Torbjörn Fagerström:
Via Loïc Lepiniec
L’université française gagne trois places par rapport à l’an dernier, dans un classement toujours largement dominé par les établissements anglo-saxons. Harvard conserve sa première place.
Via Life Sciences UPSaclay
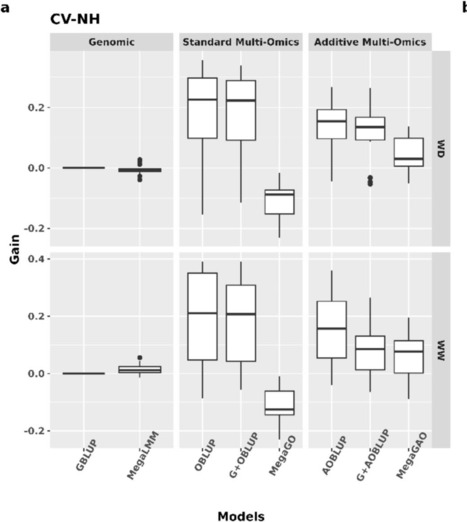
Dans une étude publiée dans Theoretical and Applied Genetics, les scientifiques de l’unité Génétique Quantitative et Evolution – GQE-Le Moulon (UPSaclay/CNRS/INRAE/AgroParisTech, Gif-sur-Yvette) ont développé des modèles permettant de prédire la productivité d’hybrides de maïs à partir de données moléculaires (transcrits et protéines). La prédiction de la valeur génétique est un enjeu important en amélioration des plantes car toutes les variétés candidates ne peuvent pas être évaluées dans tous les environnements. Des techniques de prédictions basées sur des polymorphismes au niveau de l’ADN ont été proposées il y a une vingtaine d’années, mais ces approches boites-noires ne sont pas très performantes pour prédire les effets non additifs (épistasie entre gènes, et interactions entre les gènes et les conditions de culture). De plus en plus d’études tentent de démontrer l’intérêt d’utiliser d’autres types de variations moléculaires pour modéliser les différents niveaux de régulations de l’expression des gènes au phénotype final dans un environnement donné. Les coauteurs de cette étude ont pu clairement démontrer que des données d’expression de gènes et d’abondances de protéines étaient très efficaces pour prédire les effets non-additifs quand elles étaient utilisées pour prédire dans des conditions de culture similaire à celles dans lesquelles les caractérisations moléculaires ont été réalisées. Contact : renaud.rincent@inrae.fr
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
July 11, 2024 4:45 AM
|
Plants use a combination of sophisticated local and systemic pathways to optimize growth depending on heterogeneous nutrient availability in the soil. Legume plants can acquire mineral nitrogen (N) either through their roots or via a symbiotic interaction with N-fixing rhizobia bacteria housed in so-called root nodules. In an article recently published in the Journal of Experimental Botany, in collaboration with Marc Lepetit (Montpellier) and Wouter Kohlen (Wageningen, The Netherlands), the team of Florian Frugier was interested to identify shoot-to-root systemic signals acting in Medicago truncatula plants at N-deficit or N-satiety. To this aim, plants were grown in a split-root experimental design, in which either high or low N was provided to a half of the root system, allowing the analysis of systemic pathways independently of any local N response. Among the plant hormone families analyzed, the cytokinin trans-Zeatin accumulated in plants at N-satiety. Cytokinin application by petiole feeding led to an inhibition of both root growth and nodulation. In addition, an exhaustive analysis of miRNAs revealed that miR2111 accumulates systemically under N-deficit in both shoots and non-treated distant roots, whereas a miRNA related to inorganic Phosphate (Pi)-acquisition, the miR399, does so in plants grown at N-satiety. These two accumulation patterns are dependent on CRA2 (Compact Root Architecture 2), a receptor required for CEP (C-terminally Encoded Peptide) signaling. Constitutive ectopic expression of the miR399 reduced nodule numbers and root biomass depending on Pi availability, suggesting that the miR399-dependent Pi-acquisition regulatory module controlled by N-availability affects the development of the whole legume plant root system. Contact: florian.frugier@universite-paris-saclay.fr

|
Rescooped by
Saclay Plant Sciences
from SEED-DREAM Lab info
July 4, 2024 12:14 PM
|
The European Plant Science Retreat 2024 will take place at the south of Paris in Gif-sur-Yvette. Organised by PhD students and for PhD students, the mood will be relaxed and friendly. We will have 3 days of talks, keynotes, poster sessions and various workshops. @EPSR_2024
Via Loïc Lepiniec

|
Scooped by
Saclay Plant Sciences
July 1, 2024 8:16 AM
|
Understanding plant responses to individual stresses does not mean that we understand real world situations, where stresses usually combine and interact. These interactions arise at different levels, from stress exposure to the molecular networks of the stress response. Here, we built an in-depth multi-omics description of plant responses to mild water (W) and nitrogen (N) limitations, either individually or combined, among five genetically different Arabidopsis (Arabidopsis thaliana) accessions. We highlight the different dynamics in stress response through integrative traits such as rosette growth and the physiological status of the plants. We also used transcriptomics and metabolomics profiling during a stage when the plant response was stabilized to determine the wide diversity in stress-induced changes among accessions, highlighting the limited reality of a ‘universal’ stress response. The main effect of the WxN interaction was an attenuation of the N-deficiency syndrome when combined with mild drought, but to a variable extent depending on the accession. Other traits subject to WxN interactions are often accession-specific. Multi-omics analyses identified a subset of transcript–metabolite clusters that are critical to stress responses but essentially variable according to the genotype factor. Including intra-specific diversity in our descriptions of plant stress response places our findings in perspective.
Cette année, 104 personnalités issues du monde de l’enseignement supérieur, de la recherche et de l’innovation, dont 56 femmes, ont été distinguées de l’ordre national du mérite par deux décrets parus au Journal officiel du 8 juin. Une directrice de recherche liée à l’Université Paris-Saclay a été promue au grade d’officier, ainsi que cinq scientifiques nommés au grade de chevalier. Au grade d’officier : Au grade de chevalier :
Via Life Sciences UPSaclay

|
Scooped by
Saclay Plant Sciences
May 27, 2024 6:26 AM
|
Epigenetic regulations, including chromatin accessibility, nucleosome positioning and DNA methylation intricately shape genome function. However, current chromatin profiling techniques relying on short-read sequencing technologies fail to characterise highly repetitive genomic regions and cannot detect multiple chromatin features simultaneously. Here, we performed Simultaneous Accessibility and DNA Methylation Sequencing (SAM-seq) of purified plant nuclei. Thanks to the use of long-read nanopore sequencing, SAM-seq enables high-resolution profiling of m6A-tagged chromatin accessibility together with endogenous cytosine methylation in plants. Analysis of naked genomic DNA revealed significant sequence preference biases of m6A-MTases, controllable through a normalisation step. By applying SAM-seq to Arabidopsis and maize nuclei we obtained fine-grained accessibility and DNA methylation landscapes genome-wide. We uncovered crosstalk between chromatin accessibility and DNA methylation within nucleosomes of genes, TEs, and centromeric repeats. SAM-seq also detects DNA footprints over cis-regulatory regions. Furthermore, using the single-molecule information provided by SAM-seq we identified extensive cellular heterogeneity at chromatin domains with antagonistic chromatin marks, suggesting that bivalency reflects cell-specific regulations. SAM-seq is a powerful approach to simultaneously study multiple epigenetic features over unique and repetitive sequences, opening new opportunities for the investigation of epigenetic mechanisms.
Le prochain Meeting de l’EUR Saclay Plant Sciences (SPS) aura lieu les 3 et 4 juin 2024 sur le Campus Agro Paris-Saclay (Palaiseau). Le 4 juin sera une journée scientifique ouverte à tous. L’inscription à cette journée scientifique est gratuite mais obligatoire La date limite pour s’inscrire est le 16 mai 2024 (minuit). Programme prévisionnel du 4 juin : 09:00 AM Welcome coffee
09:20 AM Introduction (Loïc Lepiniec)
09:30 AM Title to come - Claudia Jonak (Austrian Institute of Technology, Austria)
10:15 AM Title to come (SAB member)
11:00 AM Coffee break
11:30 AM The study of wild and domestic forms provides a dual perspective on the evolution of divergence with gene flow - Maud Tenaillon (UMR Quantitative Genetics and Evolution - GQE-Le Moulon) 12:15 PM Lunch (offered by SPS) 01:45 PM Unveiling the functional mechanism of Fatty Acid Photodecarboxylase: an algal photoenzyme with great promise for green fuel production - Pavel Müller (Institute of Integrative Biology of the Cell - I2BC)
02:30 PM The colorful genetic makeup of Whiteflies sheds light on plant-insect interactions - Florian Maumus (Unité de Recherche en Génomique-Info - URGI)
03:15 PM How plants deal with replication-induced DNA damage: signaling mechanisms and physiological relevance - Cécile Raynaud (Institut of Plante Sciences Paris-Saclay - IPS2)
04:00 PM Coffee break
04:30 PM Into outer space: fungal secretion of extracellular vesicles and their role in plant infection - Richard O'Connell (UMR BIOlogie et GEstion des Risques en agriculture - BIOGER)
05:15 PM Spatio-temporal regulation of plant cell division - David Bouchez (Institut Jean-Pierre Bourgin - IJPB) 06:00 PM End of the meeting
Via Life Sciences UPSaclay
Portrait Jeune Chercheur - Kévin Magne, chercheur en physiologie des interactions plantes-microorganismes
Kévin Magne est chargé de recherche INRAE depuis novembre 2023 dans l’équipe SYmbiotic Nitrogen Acquisition in Plant-microbe Systems (SYNAPS) dirigé par la Prof. A. Dellagi à l’Institut Jean-Pierre Bourgin (IJPB) de Versailles. L’équipe SYNAPS vise à mieux comprendre et à exploiter le microbiote des grandes cultures céréalières dans le but d’améliorer l’efficacité de l’utilisation de l’azote des plantes et de réduire l’utilisation des engrais azotés de synthèse. Kévin est issu du Master Biologie et Valorisation des Plantes de l’Université de Strasbourg et il a réalisé sa thèse dans le laboratoire du Dr. P. Ratet à l’Institut des Sciences des Plantes de Paris-Saclay (IPS2) de Gif-sur-Yvette. Durant sa thèse Kévin a étudié le rôle des gènes de type BLADE-ON-PETIOLE dans le développement et le maintien de l’identité des nodosités fixatrices d’azote chez les légumineuses ainsi que leurs implications dans le développement végétative et reproductif des dicotylédones et des monocotylédones. Il obtiendra son doctorat de biologie de l’Université Paris-Saclay en 2017. En 2018, Kévin rejoint le groupe du Dr. R. Geurts au laboratoire de biologie moléculaire de l’Université de Wageningen aux Pays-Bas. Il y étudie la régulation des gènes de type BLADE-ON-PETIOLE au cours de la symbiose Parasponia-rhizobium ainsi que leurs rôles dans la régulation de l’activité du cambium vasculaire et la xylogenèse chez Parasponia. En 2020, Kévin effectue un second post-doctorat sous la supervision du Prof. V. Gruber à l’IPS2 dans lequel il étudie la régulation de l’immunité des nodosités fixatrices d’azote. En 2021, Kévin réalise un troisième post-doctorat supervisé par le Dr. P. Ratet dans lequel il cherche à comprendre l’origine évolutive des relations mutualistes entre les légumineuses et les rhizobia par l’utilisation d’une souche bactérienne atypique du genre Ensifer capable d’adopter plusieurs comportements vis-à-vis d’un même hôte. Aujourd’hui, au sein de l’équipe SYNAPS, Kévin développe et étudie les effets bénéfiques du réseau mycorhizien commun dans un contexte de cultures associées impliquant céréales et légumineuses. Il s'intéresse notamment à la redistribution de l’azote fixé par le couple légumineuse/rhizobium au profit des cultures céréalières. « Le monde contient bien assez pour les besoins de chacun, mais pas assez pour la cupidité de tous » - Gandhi Contact : kevin.magne@inrae.fr
Via Life Sciences UPSaclay
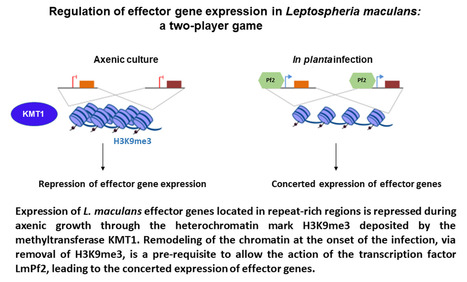
Une étude publiée dans The New Phytologist, réalisée dans le cadre de la thèse de Colin Clairet, sous la direction de Jessica Soyer et d’Isabelle Fudal au sein de l’unité BIOGER (INRAE/UPSaclay, Campus Agro Paris-Saclay, INRAE Palaiseau), a permis de caractériser la façon dont est contrôlée l’expression des effecteurs d’un champignon phytopathogène au cours de l’infection de sa plante hôte. Les effecteurs sont des molécules sécrétées par l’agent pathogène qui facilitent l’infection de sa plante hôte et l’établissement de la maladie. Ces effecteurs sont produits de façon concertée au cours de l’infection. Cependant, on sait peu de choses sur la manière dont l'expression des gènes codant ces effecteurs est régulée. Comme une partie de ces gènes est localisée dans des régions riches en éléments répétés, le rôle du remodelage de la chromatine dans leur régulation a été récemment étudié, établissant notamment que la marque hétérochromatinienne H3K9me3 (triméthylation de la lysine 9 de l’histone 3), mise en place par la méthyltransférase KMT1, était impliquée dans le contrôle de l’expression d’effecteurs chez plusieurs champignons phytopathogènes. Cependant, cette régulation chromatinienne ne permettait pas, à elle seule, d’expliquer l’induction d’expression des effecteurs au moment de l’infection, suggérant qu'un second niveau de régulation, impliquant probablement un facteur de transcription spécifique, était nécessaire. Chez Leptosphaeria maculans, un champignon de la famille des Dothideomycetes causant la nécrose du collet du colza, les chercheurs ont identifié l'orthologue de Pf2, un facteur de transcription appartenant à la famille Zn2Cys6 spécifique aux champignons, et décrit comme essentiel à la pathogénie et à l'expression des gènes codant des effecteurs. Les chercheurs ont étudié son rôle conjointement avec KMT1, en inactivant et en surexprimant LmPf2 dans une souche sauvage et dans un mutant inactivé pour KMT1. Les analyses fonctionnelles et transcriptomiques des transformants correspondants ont mis en évidence un rôle essentiel de LmPf2 dans l'établissement de la pathogénie et un effet majeur sur l'induction de l'expression des effecteurs une fois la répression exercée par KMT1 levée. Ces résultats montrent, pour la première fois, l’implication d’un double contrôle dans l'expression des effecteurs fongiques. Contact : jessica.soyer@inrae.fr ou isabelle.fudal@inrae.fr
Via Life Sciences UPSaclay
|
 Your new post is loading...
Your new post is loading...
 Your new post is loading...
Your new post is loading...