Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
September 29, 2024 5:35 PM
|
Depuis sa création, le Genoscope – Centre National de Séquençage participe à des projets collaboratifs à fort impact scientifique en fournissant à la communauté scientifique toute l’expertise et les capacités de production et d’analyse de données indispensables à ces projets. Depuis sa contribution majeure, il y a près de 20 ans, au projet Human Genome en publiant la séquence complète du chromosome 14 (Heilig et al., Nature 2003), la plateforme de séquençage située au cœur du biocluster Genopole installé à Évry-Courcouronnes a réalisé le séquençage de nombreuses plantes (vigne, riz, blé…), d’animaux (tétraodon, anophèle…), de champignons (truffe, champignons pathogènes du colza et de la vigne) et de plus d’une centaine de procaryotes. Grâce à sa collaboration avec le Très Grand Centre de Calculs du CEA, situé lui-aussi en Essonne, le Genoscope - CNS a permis de traiter de très grands ensembles de données en utilisant l’informatique haute performance de pointe. Ainsi, depuis sa création, le Genoscope-CNS a contribué à plus de 750 publications et ceci grâce à sa plateforme de séquençage. Nous l’évoquions il y a quelques mois déjà dans un FOCUS PLATEFORME, le Genoscope - CNS a choisi de s’aligner sur la stratégie et les engagements de l'Union Européenne en matière de biodiversité pour 2030 tels que la lutte contre le déclin des pollinisateurs, l'amélioration de l'état de conservation des espèces et des habitats clés, la promotion de la restauration écologique des systèmes forestiers et des rivières, et la minimisation des impacts des espèces invasives. Accompagné d’autres partenaires européens, le Genoscope – CNS participe activement à la recherche en génomique de la biodiversité en Europe visant à étayer la conservation de celle-ci et sa biosurveillance basée sur l'ADN par la génération de génomes de référence de haute qualité provenant de toutes les formes de vie afin de d’établir un Atlas européen des génomes de référence (ERGA). L’initiative ERGA est une réponse scientifique paneuropéenne aux menaces actuelles qui pèsent sur la biodiversité. Les génomes de référence fournissent l’aperçu le plus complet des bases génétiques qui constituent chaque espèce et représentent une ressource puissante pour comprendre le fonctionnement de la biodiversité. Alors qu’environ un cinquième des quelques 200 000 espèces européennes sont menacées d’extinction, nous devons agir rapidement et ensemble pour générer à grande échelle des ressources génomiques complètes de haute qualité. Les acteurs de l'Atlas Européen des Génomes de Référence (ERGA) célèbrent tous une réalisation majeure avec l'achèvement réussi du projet pilote. Cette initiative novatrice a réuni des chercheurs de 33 pays pour produire des génomes de référence de haute qualité pour 98 espèces européennes, faisant progresser la création d'une base de données génomique complète pour la biodiversité européenne. La France a été représentée par le Genoscope – CNS avec un financement du réseau France Génomique. Les accomplissements du projet pilote incluent les premiers assemblages de génomes au niveau chromosomique pour des espèces de France, telles que le Vison d'Europe (Mustella lutreola) et le Marmottier (Prunus brigantina) (Mc Cartney et al, Nature 2024). Ces génomes sont désormais accessibles au public, illustrant la puissance de la collaboration internationale dans l'avancement de la recherche sur la biodiversité. La plateforme de séquençage a également permis de séquencer les génomes des espèces Phaeosaccion multiseriatum, Aglaophenia octodonta, Acrocephalus paludicola, Niphargus dancaui, Niphargus schellenbergi, Buccinim undatum… Mais serez-vous assez curieux pour découvrir quelles espèces se cachent derrière ces noms ? Parmi elles se trouvent des algues, un coquillage présent dans les plateaux de fruits de mer ou un oiseau migrateur ! Le projet pilote d’ERGA a également mis en évidence l’importance croissante de la génomique de la biodiversité, avec des données destinées à orienter les efforts de conservation et les pratiques durables. Par exemple, le génome de la Grande Argentine, une espèce de poisson d'importance commerciale, aidera à prendre des décisions éclairées sur des pratiques de pêche responsables. Dans le cadre du projet mondial Earth BioGenome Project, ERGA a démontré qu’un modèle de production de génomes décentralisé et collaboratif est à la fois faisable et efficace, même sans financement centralisé. Ce succès établit une nouvelle norme pour les initiatives de génomique de la biodiversité à grande échelle dans le monde entier ! Pour obtenir des renseignements sur les modalités d’accès de la plateforme de séquençage, n’hésitez pas à contacter Pedro OLIVEIRA ! Contact : Pedro OLIVEIRA (Pedro.COUTOOLIVEIRA@genoscope.cns.fr) Plug In Labs Université Paris-Saclay : cliquer ICI A propos de GENOSCOPE / Plateforme de Séquençage - Genopole. Le Genoscope s’est orienté vers l’étude de la biodiversité, à travers le séquençage de novo de grands génomes complets, de métagénomes complexes et plus généralement de collections d’ADN nécessitant une approche de séquençage à très grande échelle. L’équipe du laboratoire de séquençage s’implique dans la prise en main des projets à partir de la réception des échantillons jusqu’à la validation des séquences brutes. Le séquençage est assuré par des séquenceurs très haut débit de deuxième et troisième génération. Ces équipements high-techs permettent d’atteindre un débit autorisant le séquençage de génomes entiers dans un intervalle de temps limité. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 66 entreprises de biotechnologies, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : soutenir les politiques nationales de réindustrialisation et de souveraineté sanitaire, créer et soutenir les entreprises de biotechnologies et le transfert de technologies vers le secteur industriel (notamment pour décarboner la production), favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
September 15, 2024 10:36 AM
|
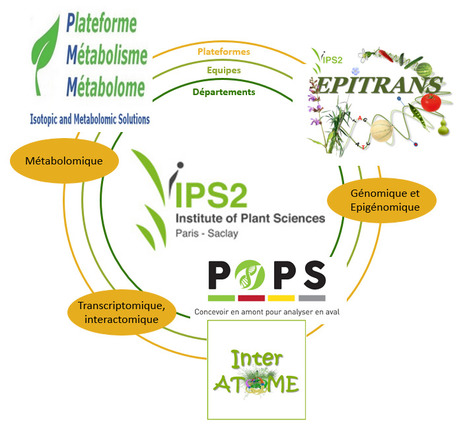
La plateforme SPOmics-Interactome (Institute of Plant Sciences Paris-Saclay (IPS2), Univ Evry, INRAE, CNRS, Université Paris-Saclay, Université Paris Cité) et l’équipe « Génomique et Epigénétique des Tumeurs Rares » (INSERM U1016 - CNRS UMR 8104, Institut Cochin) ont récemment établi une collaboration pour la mise en place d’un test fonctionnel à haut débit. Le but de ce projet est d’évaluer simultanément l’impact de tous les variants faux-sens du domaine exonucléase de l’ADN polymérase epsilon (POLE) par un test d’édition génomique à saturation au sein d’un modèle levure. La réplication de l’ADN est un processus complexe qui implique plusieurs ADN polymérases, dont l’ADN polymérase epsilon pour le brin direct et l’ADN polymérase delta pour les fragments d’Okazaki. L’ADN polymérase epsilon possède plusieurs domaines dont un domaine polymérase pour la synthèse du brin naissant et un domaine 3’5’ exonucléase corrigeant les erreurs réalisées par le domaine polymérase. Ce domaine exonucléase, qui est très conservé de la levure à l’Homme, permet l’excision et la correction des mismatchs naissants avant même leur sortie du complexe polymérase, augmentant d’un facteur 100 la fidélité de la réplication. Il y a maintenant 10 ans, une première étude mettait en évidence la causalité d’un variant faux-sens du domaine exonucléase de POLE au sein d’un syndrome de prédisposition aux cancers principalement digestifs, autrement nommé PPAP pour polymerase proofreading associated polyposis. Depuis, les descriptions se sont multipliées et le risque cumulé d’avoir développé un cancer à l’âge de 70 ans est de l’ordre de 70%. Grace à l’expertise présente sur la plateforme et aux connaissances de l’équipe Génomique et Epigénétique des Tumeurs Rares nous optimisons ce test qui s’appuie sur plusieurs étapes (mutagénèse, sélection fonctionnelle, séquençage et analyses bioinformatiques) et qui prévoit à plusieurs reprises du repiquage automatisé de plusieurs milliers de clones de levure depuis une gélose vers des plaques 96 puits. Contact : Dario Monachello (dario.monachello@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Envie de (re)lire leurs précédents FOCUS PLATEFORME ? (14 septembre 2020) ? FOCUS PLATEFORME : SPOmics-Interactome ou comment les plantes peuvent servir la découverte de nouveaux rôles de l'ubiquitination des protéines ; (18 septembre 2023) FOCUS PLATEFORME : SPOmics-Interactome ou comment les plantes peuvent servir la découverte de nouveaux rôles de l'ubiquitination des protéines Découvrir toutes les plateformes de l’IPS2 ? cliquer ICI ! IPS2 / SPOmics-Interactome. SPOmics-Interactome est la plateforme d'étude des interactions protéine-protéine de l'IPS2. Les technologies qu'elle maitrise sont basée sur la seule méthode permettant la détection de ces interactions à haut débit et in vivo - le système double-hybride chez la levure (Y2H) - aujourd'hui optimisé et automatisé. A ce titre, grâce à l'utilisation de plaques 384 puits et d'un système robotisé d'une part, mais aussi d'un protocole entièrement en milieu liquide, la plateforme est capable de cribler un pool de 50 protéines hybrides (DB- X) contre la banque de protéines hybrides AD-AIM (environ 12000 protéines d'Arabidopsis) en deux mois. Le protocole généralement utilisé est le suivant : Les plasmides portant les ORFs codant pour les protéines d'intérêt fournis par nos collaborateurs dans le vecteur pDEST - DB sont utilisés pour transformer des levures, puis les protéines DB-X hybrides sont testées pour éliminer celles capables d'auto-activation. Les levures exprimant les protéines DB-X hybrides sont alors cultivées individuellement dans des milieux sélectifs, puis combinées en pools de 50 clones et criblées contre la banque AD-AIM. Après identification des protéines candidates, une analyse matricielle en Y2H est effectuée par « déconvolution » des 50 DB-proies et par tests individuels contre les candidats AD-appâts. Une étape finale de séquençage des protéines AD- et DB- permet la validation de l'identité des protéines en interaction. Chaque nouvel ORF criblé est intégré dans la banque AD- permettant une constante implémentation du réseau. A propos de l’IPS2. L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées.

|
Scooped by
Life Sciences UPSaclay
August 26, 2024 5:36 PM
|
Qu’est-ce que le GIS IBiSA ? Quelles sont ses missions ? Quels sont les intérêts du label ? Et les conditions d’obtention ? Réponses dans un document synthétique, qui donne aussi des chiffres sur les structures labellisées, les appels d’offres Plateformes et CRB. Lire la communication. Vous souhaitez recevoir les news d’IBiSA chaque trimestre ? Inscrivez-vous à la newsletter ! Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique.

|
Scooped by
Life Sciences UPSaclay
July 7, 2024 11:18 AM
|
Qu’est-ce que la magnétoencéphalographie (MEG) ? La MEG est une technique d’imagerie cérébrale non invasive permettant de mesurer les champs magnétiques générés par les neurones du cerveau humain avec une haute résolution temporelle et une bonne résolution spatiale. Lorsqu’elle est combinée à l'IRM anatomique du participant, la MEG permet de reconstruire l'activité cérébrale dans le cortex et certaines zones sous-corticales avec une résolution spatiale de quelques millimètres. Elle constitue une méthode adaptée à l’étude des bases neurales des grandes fonctions cognitives humaines telles que le langage, l’attention, la perception et la prise de décision. La magnétoencéphalographie (MEG) est présente sur NeuroSpin (Institut des Sciences du Vivant Frédéric Joliot, Centre CEA Paris-Saclay, Gif-sur-Yvette) depuis 2008, mais depuis quelques mois, c’est un nouvel équipement (financé à hauteur de 2 M€ par la région Ile-de-France (CPER 2021-2027)) qui est progressivement mis en service sur la plateforme NEUROSPIN / Imagerie chez l'homme en MEG : un Triux Neo de MEGIN ! Quoi de neuf avec cet équipement (Triux Neo, MEGIN) ? La nouvelle électronique de ses capteurs permet un gain considérable de qualité d’enregistrement par rapport à l’ancienne machine (Neuromag 306, Elekta) : son rapport signal-à-bruit augmente de 20% et la plage dynamique est multipliée par 3 par rapport à l’ancien système, permettant ainsi des acquisitions chez les patients avec une stimulation cérébrale profonde, des implants cochléaires ou électrodes implantées. Son nouveau siège modulaire et ajustable permet de simplifier les expériences chez l’enfant et le bébé. En effet, ces petits participants seront installés plus facilement dans ce dispositif entièrement adapté. Enfin, elle possède un système de recyclage d’hélium : un liquéfacteur reliquéfie 100% de l’hélium vaporisé durant la nuit. Cela représente un gain économique et écologique très important sans impacter les plages d’acquisition de données ! Au programme des prochains mois, plusieurs projets de recherche auront l’opportunité de bénéficier de ce nouvel instrument notamment pour étudier comment le cerveau humain calcule l’incertitude lorsqu’il prend une décision, comment il mémorise des séquences d’évènements lorsque celles-ci sont régulières et enfin, si la pratique de la musique permet aux enfants de meilleurs apprentissages scolaires. Contacts : Leila Azizi (leila.azizi@cea.fr); Fosca Al Roumi (fosca.alroumi@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, en octobre 2020, la plateforme publiait son premier FOCUS PLATEFORME… Redécouvrez- le aujourd’hui : FOCUS PLATEFORME : La MEG révèle les mécanismes utilisés par le cerveau humain pour compresser l’information en mémoire ! A propos de NeuroSpin. Neurospin est une infrastructure de recherche sur le cerveau exploitant des grands instruments d'imagerie. NeuroSpin offre à la communauté scientifique publique et privée la possibilité de faire progresser la connaissance du cerveau, et particulièrement du cerveau humain, en proposant un accès à des méthodologies de pointes en imagerie cérébrale et en neuro-informatique. NeuroSpin développe et met à la disposition de la communauté des instruments uniques, notamment en imagerie très hauts champs et dans le domaine des big data. Cette offre s'inscrit dans le cadre des missions spécifiques de NeuroSpin qui sont : i) analyser les fonctions du cerveau humain, leur développement dans l'enfance, et l'impact de la culture et de l'éducation ; ii) identifier les marqueurs et les mécanismes de maladies neurologiques, psychiatriques et neurodéveloppementales ; iii) comparer le cerveau humain et celui d'autres espèces animales ; développer et tester des méthodes d'imagerie à toutes les échelles d'observation : par résonance magnétique (IRM), par électro- et magnéto-encéphalographie (EEG et MEG), et par électrophysiologie massivement parallèle ou l'imagerie photonique et v) développer des logiciels spécialisés dans le traitement et la modélisation des grands jeux de données en neuroimagerie. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
June 22, 2024 6:31 PM
|
Les grandes évolutions de la cryo-microscopie électronique. En l’espace d’une décennie, cette méthode d’observation ultraprécise n’a eu de cesse d’être perfectionnée. Co-responsable de la plateforme Biostruct@UPSAY, Stéphane Bressanelli explique le bénéfice de ces améliorations dans le domaine de la biologie structurale. Lire l’article. IBiSA est dans le Microscopoly ! A l’occasion de ses 20 ans, le réseau technologique de microscopie photonique de fluorescence multidimensionnelle (RTmfm) lance la version microscopie du Monopoly ! IBiSA se trouve sur le plateau, aux côtés de nombreuses plateformes labellisées. L’empreinte carbone, c’est aussi une affaire de plateformes. Les plateformes sont-elles obligées de réaliser un bilan carbone ? Que faut-il prendre en compte ? Tête-à-tête avec Floriant Bellvert, ingénieur sur la plateforme MetaToul et intervenant sur les ateliers de formation IBiSA axés empreinte environnementale de la recherche. Lire l'article. Quels outils pour la gestion de projets sur les plateformes ? La gestion de projets est facilitée par une multitude de logiciels. Comment choisir et adapter les outils aux plateformes ? Exemples de solutions originales présentées par des ingénieurs de plateformes au cours des ateliers de formation IBiSA. Lire l'article. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
June 9, 2024 5:35 PM
|
Le cerveau humain contient des dizaines de milliards de neurones, connectés entre eux par un formidable réseau de synapses. La synapse est le lieu d’échanges moléculaires complexes, et constitue l’entité fonctionnelle de la neurotransmission. Des atteintes synaptiques sont retrouvées dans la plupart des maladies neurologiques (maladie d'Alzheimer, maladie de Parkinson, épilepsie, etc.), mais aussi dans certaines maladies psychiatriques (dépression, schizophrénie, etc.), où l’on suspecte certaines anomalies de la plasticité synaptique. Traditionnellement, la densité synaptique est estimée par des méthodes invasives ex vivo, ne permettant pas d’apprécier la dynamique d’évolution de la densité synaptique de manière longitudinale. Depuis quelques années, l'imagerie par tomographie par émission de positons (TEP) de la glycoprotéine 2A de la vésicule synaptique (SV2A) a été développée comme méthode quantitative et non-invasive pour mesurer la densité synaptique et étudier les phénomènes de synaptogénèse ou de plasticité synaptique in vivo. SV2A est exprimée de façon ubiquitaire dans pratiquement toutes les vésicules présynaptiques, indépendamment du système de neurotransmission impliqué (synapse glutamatergique, dopaminergique, serotoninergique, etc.). Les chercheurs de BioMaps (UMR CEA/CNRS/Inserm/Université Paris-Saclay, Service Hospitalier Frédéric Joliot (SHFJ), Institut Joliot, Orsay) produisent et utilisent le [11C]UCB-J, un radiopharmaceutique spécifique de SV2A, pour étudier la densité synaptique dans les différentes régions du cerveau vivant. L’imagerie TEP au [11C]UCB-J permet d’apprécier l’atteinte synaptique chez l’Homme, ou dans différents modèles animaux de pathologies cérébrales. Par rapport aux approches conventionnelles d’imagerie fonctionnelle (IRMf ou TEP au [18F]FDG), qui étudient principalement la fonction neuronale, l’imagerie TEP SV2A renseigne plus spécifiquement sur la densité synaptique et permet ainsi de réaliser des mesures répétées dans le temps afin de mettre en évidence les changements dynamiques de la densité synaptique. La figure ci-dessus met en avant la structure chimique du traceur (UCB-J), le positionnement de son marquage isotopique par le carbone-11 (T1/2 : 20,4 minutes seulement), et une image choisie (coupe axiale) de sa distribution cérébrale chez le rat (gradient de couleur proportionnel à la concentration, rouge = forte, bleu = faible). Le Service Hospitalier Frédéric Joliot (SHFJ) opère plusieurs plateaux techniques d’imagerie moléculaire par TEP, dont l’un d’entre eux, notamment équipé d’imageurs microTEP et microTEP-CT est dédié à l’imagerie du petit animal (SHFJ / Imagerie préclinique, in vivo, MicroTEP). Des études longitudinales sont actuellement réalisées dans différents modèles de pathologies cérébrales, en collaboration avec les équipes de chercheurs en neurologie, psychiatrie et neuroscience. Contacts : Nicolas Tournier (nicolas.tournier@cea.fr), Plug In Labs Université Paris-Saclay : cliquer ICI SHFJ / Imagerie préclinique, in vivo, MicroTEP. La plateforme d'imagerie TEP préclinique du SHFJ offre les équipements nécessaires pour préparer les animaux et réaliser de l'imagerie corps entier par tomographie par émission de positons (TEP) seule ou couplée à la tomodensitométrie (CT) chez le rongeur, au moyen de radiotraceurs provenant de l’extérieur ou produits sur site. Les services associés (hébergement des animaux, autoradiographie, immunohistochimie) peuvent compléter les prestations. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
May 26, 2024 5:06 PM
|
Créée en 2017, la plateforme d’histologie – anatomocytopathologie de Genopole propose les équipements, le savoir-faire et l’expertise nécessaire pour répondre aux besoins croissants des entités du biocluster Genopole dont l’activité est liée au domaine de la Santé. Les prestations de service proposées par cette plateforme sont tout naturellement la préparation des tissus (cryoconservation, inclusion en paraffine ou en résine), leur section (cryosection, microtomie ou ultra-microtomie), leurs colorations histologiques dites classiques ou bien la réalisation de marquages immunohistochimiques (DAB ou fluorescents) via des automates et enfin leur exploration en microscopie en vue d’une analyse morphométrique précise des tissus observés. Pour y arriver, des technologies et des compétences complémentaires sont requises à chacune de ces étapes. D’autre part, l’expertise sur lames par les médecins pathologistes du service anatomie et cytologie pathologiques (dit Service ACP) du Centre Hospitalier Sud Francilien (CHSF, Corbeil-Essonnes) peut, dans certains cas, constituer l’étape finale de cette chaine de préparation des tissus, étape qui nécessite un savoir-faire et une technicité certaine afin de préserver la structure des tissus parfois très fragiles et précieux. L’intégralité des étapes de préparation des tissus sont réalisées par les membres de la plateforme d’histologie, certifiée ISO 9001:2015 et dirigée par Nathalie DANIELE en sa qualité de Responsable du département d’évaluation préclinique de Généthon qui compte 4 équipes dont celle dédiée à l’histologie-immunomarquage. Nathalie DANIELE nous indique qu’en 2023 « l’équipe a développé trois nouvelles colorations qui viennent s’ajouter au catalogue des colorations dites de routine que nous proposons déjà : la coloration au trichrome de Gomori pour les cryosections de muscle, le trichrome de Masson pour les tissus inclus en paraffine, et le bleu au luxol rapide ou LFB pour Luxol fast blue pour une mise en évidence de la myéline présente dans le système nerveux » avant d’ajouter que « en 2024, il y aura un double défi à relever pour toute l’équipe de la plateforme d’histologie à savoir, le développement de la transcriptomique spatiale déjà initiée en 2023 d’une part et, d’autre part, la mise en place d’une formation exhaustive en histologie couvrant les bases histologiques des tissus et s’étendant jusqu’à l’analyse et l’interprétation des lames qui sera ouverte à toute personne souhaitant se former à ces techniques qui requièrent un réel savoir-faire et beaucoup de patience ! ». Vous l’aurez également compris, la singularité de la plateforme d’histologie – anatomocytopathologie est qu’elle s’appuie sur l’expertise des membres du service ACP du CHSF dont l’effectif a augmenté significativement au moment du regroupement avec le service du même nom du Groupe Hospitalier Nord Essonne (Juvisy-sur-Orge/Longjumeau/Orsay) comme nous l’indique le Dr. Virginie CAHN, Cheffe du Service ACP du CHSF : « En effet, trois médecins pathologistes, cinq techniciens, une aide de laboratoire et une secrétaire médicale sont venus renforcer l’équipe déjà présente. Nous sommes tous ravis de cette nouvelle dynamique de groupe mais également de pouvoir collaborer avec les entités génopolitaines qui nous en font la demande », avant d’ajouter « c’est un réel plaisir pour nous de pouvoir diversifier notre activité et de mettre à disposition notre savoir-faire et nos compétences au service de projets aussi intéressants qu’ambitieux ». Si vous avez des besoins en préparation de tissus, en coloration ou en expertise sur lames, n’hésitez pas, contactez-nous ! Contact : Julien Picot (julien.picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, le 3 mai 2021, la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? GENOPOLE / Plateforme d'histologie-anatomocytopathologie. La plateforme d'histologie-anatomocytopathologie offre des équipements et le savoir-faire associé nécessaires à la préparation des tissus, leurs colorations histologiques et/ou immunohistochimiques en vue de leur exploration par imagerie photonique ou électronique. Cette plateforme s'appuie sur l'expertise de médecins anatomocytopathologistes du Centre Hospitalier Sud-Francilien pour la bonne réalisation de projets précliniques et cliniques. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à la bioéconomie, Genopole réunit 65 entreprises, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : favoriser l’émergence et la croissance de sociétés de biotechnologie, le transfert d’innovations vers le secteur industriel, le développement de la recherche et l’enseignement supérieur en sciences de la vie. Dirigé par Gilles Trystram, Genopole est un Groupement d’intérêt public principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes technologiques labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, contactez Julien Picot (julien.picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
May 19, 2024 5:18 PM
|
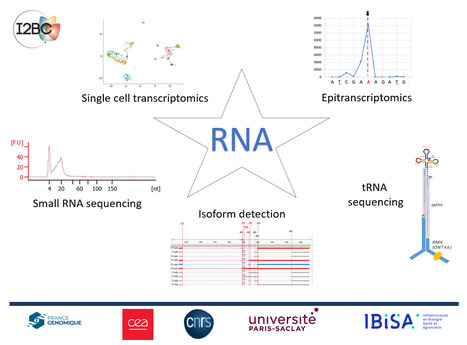
Des technologies variées au service des ARNs à l’I2BC ! Qu’ils soient messagers, petits, d’expression variée entre cellules, modifiés, ou très structurés, les ARN offrent une vision précieuse du fonctionnement cellulaire et de l’expression des gènes. Mais accéder à toute leur diversité nécessite des technologies de pointe et une connaissance avancée de leurs spécificités. Ces technologies sont accessibles aux équipes de recherche du territoire Paris-Saclay et au-delà grâce à l’équipement et à l’expertise des ingénieurs de la plateforme de séquençage à haut débit de l’I2BC. A l’inverse des méthodes classiques de séquençage d’ARN qui ne fournissent qu’une moyenne des profils transcriptomiques d’une population de cellules, le séquençage d’ARN en cellule unique a pour avantage de donner accès à la diversité des profils transcriptomiques entre cellules, ainsi qu’aux profils de coexpression au sein de chaque cellule. S’appuyant sur la technologie 10X Genomics, la plateforme de séquençage à haut débit de l’I2BC a ainsi collaboré avec l’équipe Levraud/Joly (NeuroPSI/TEFOR Paris Saclay) afin de déterminer les mécanismes de réparation à l’œuvre dans certaines cellules du cerveau (Banerjee et al., 2022), ou avec l’équipe Noordermeer (I2BC) pour étudier l’impact de l’organisation de la chromatine sur l’activité des gènes (Moniot-Perron et al., 2023). Autre technologie disponible, le séquençage Nanopore a pris une importance de premier plan en biologie. Cette technologie de séquençage en lecture longue permet d’obtenir des lectures directes de fragments d’ADN ou de molécules d’ARN (van Dijk et al., 2023). Pour ces dernières, elle permet ainsi l’identification des isoformes d’ARN messagers (épissage alternatif) ainsi que la détection directe des bases modifiées. Cette technologie est applicable par exemple à la détection des modifications présentes sur les anti-codons des ARNt. La plateforme est une des seules en France proposant un service de détection directe des modifications d’ARN avec la technologie Oxford Nanopore. Enfin, la plateforme de séquençage à haut débit a une expertise forte en séquençage des petits ARN (lecture courte). Les technologies de séquençage de nouvelle génération ont révolutionné l’étude des petits ARN (small RNA : sRNA). Cependant, les méthodes classiques de préparation de banques à partir de sRNA introduisent de nombreux biais, principalement lors des étapes de ligation de l’adaptateur. Plusieurs types de sRNA contiennent ainsi un 2'-O-méthyle (2'-OMe) au niveau de leur terminaison 3', inhibant la ligation de l’adaptateur et rendant la préparation de la banque particulièrement difficile. Dans ce contexte, la plateforme a comparé les protocoles existants et développé un protocole original réduisant les biais (van Dijk et al., 2021). A noter dans vos agendas : en juin 2024 aura lieu à l’I2BC une formation spécifiquement dédiée à la préparation des banques NGS (Illumina) à partir des petits ARNs. N’hésitez pas à vous y inscrire : https://cnrsformation.cnrs.fr/preparer-des-banques-ngs-a-partir-des-petits-arn-pour-la-technologie-illumina (Attention : il vous faut copier ce lien dans votre navigateur favori … il ne fonctionne pas en cliquant directement dessus !). Contacts : I2BC-sequencage@i2bc.paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI I2BC / Plateforme de séquençage à haut débit. La plateforme de séquençage à haut débit de l’Institut de Biologie Intégrative de la Cellule (I2BC, campus CNRS de Gif-sur-Yvette) collabore étroitement avec un grand nombre d’équipes de recherche du territoire Paris-Saclay et au-delà. Accessible à tous les utilisateurs académiques et industriels, son accompagnement couvre non seulement la mise en place de protocoles et technologies avancées en collaboration avec les équipes, mais aussi la réalisation de prestations de services de séquençage. Son expertise porte sur une grande variété de domaines en transcriptomique ou génomique avec les technologies Oxford Nanopore (lectures longues), Illumina (lectures courtes), ou 10X Genomics (cellule unique). Ses services, outre le conseil et l’accompagnement, couvrent toutes les étapes d’un projet de séquençage, de la préparation de librairies au séquençage mais aussi à l’analyse bioinformatique des données. Enfin, elle est membre de l’infrastructure nationale France Génomique, certifiée ISO:9001/NFX:50-900, labellisée IBiSA, et participe au Réseau des plateformes de génomique de Paris-Saclay (GENOPS). A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 61 équipes de recherches et 16 plateformes technologiques réparties en 6 pôles. Principalement localisé sur le campus CNRS de Gif-sur-Yvette, l’institut est spécialisé dans les approches transverses en biologie cellulaire et moléculaire.

|
Scooped by
Life Sciences UPSaclay
April 25, 2024 6:21 PM
|
Le développement de nouveaux médicaments est un processus long et complexe qui nécessite de comprendre en détail comment le principe actif est distribué dans l'organisme et sa manière d’interagir avec lui. Pour ce faire, le marquage (radioactif) est un outil de choix, permettant de suivre le trajet d’une molécule et de ses métabolites in vivo. En particulier, les isotopes du carbone, carbone-11 (11C, émetteur beta+, T1/2 = 20 min) et carbone-14 (14C, émetteur beta-, T1/2 = 5730 ans), sont des radioisotopes de choix, car ils peuvent être incorporés dans une molécule sans en modifier les propriétés biologiques. On parle alors de marquage isotopique ou comme le disent bien mieux les anglo-saxons, « true labeling ». En revanche, l'un des principaux défis du radiomarquage au carbone (11C, 14C) réside dans la synthèse même des composés visés. En effet le dioxyde de carbone (CO2), qui s’avère être la source la plus courante de carbone radioactif, est un gaz, de plus très stable, et donc, très peu réactif. C’est dans ce contexte que s’inscrivent les recherches menées au sein du Service de Chimie Bioorganique et de Marquage (SCBM, Département Médicaments et technologies pour la Santé, Institut Joliot, CEA, Centre de Paris-Saclay) et tout particulièrement sa plateforme de marquage isotopique. Cette dernière a récemment mis au point deux méthodes de marquage novatrices qui permettent d'intégrer des isotopes du carbone provenant du dioxyde de carbone (CO2). Elles reposent sur des réductions à un ou deux électrons de ce précurseur primaire, réalisées par photo catalyse à l'aide de lumière bleue. D’une part, nous avons identifié un accès rapide au radical anion du CO2, décrit pour la première fois avec des isotopes du carbone, qui a permis l’insertion de ces isotopes dans une large librairie de molécules d’intérêt, dont des composés « médicaments » modèles (Malandain et al., JACS 2023). D’autre part, en collaboration avec l’équipe de Zakaria Halime / Institut de Chimie Moléculaire et des Matériaux d'Orsay (ICMMO, Université Paris-Saclay / CNRS), nous avons développé une méthode permettant la réduction du dioxyde de carbone (CO2) en monoxyde de carbone (CO). Cet excellent « réactif » chimique, a ensuite été utilisé dans une panoplie de réactions pour conduire à des dérivés de type esters, amides, cétones, aldéhydes … Un point fort de cette nouvelle méthode est, notamment, la possibilité d’atteindre la conversion totale du CO2 en CO après seulement 10 minutes d’irradiation du photocatalyseur dans le bleu (Monticelli et al., Nat. Comm. 2023). Applicables sans difficultés particulières avec le carbone-14, ces nouvelles méthodes de marquage ont aussi démontré leur efficacité avec le carbone-11, isotope radioactif à demi-vie extrêmement courte (20 minutes !) dont la manipulation implique la mise en place de conditions nettement plus restrictives. Une première application en radiomarquage (Dr Fabien Caillé) et imagerie in vivo par Tomographie par Emission de Positons (TEP) chez la souris (Dr Nicolas Tournier) a été faite en collaboration via un autre département de Joliot (SHFJ, BioMaps, Orsay) avec l’[11C]oxaprozin, médicament de type anti-inflammatoire non stéroïdien (AINS). Ces méthodes de radiochimie, faciles d’utilisation et relativement universelles, constituent d’ores et déjà de nouveaux outils de marquage dans l’arsenal de nos plateformes ! Contacts: Davide Audisio (davide.audisio@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme de marquage isotopique du CEA / Paris-Saclay (Institut Joliot, Département Médicaments et Technologies pour la Santé, Service de Chimie Bioorganique et de Marquage, Gif-sur-Yvette) est unique sur le territoire Paris-Saclay. Forte de son expertise dans la préparation (synthèse, contrôle de qualité) et formulation de molécules marqués, elle assure régulièrement des prestations et collaborations, académiques comme industrielles, dans le domaine du (radio)marquage moléculaire. Elle offre également à la demande, son expertise et environnement unique de travail (laboratoires « chauds », équipements dédiés) pour l’analyse et la caractérisation d’échantillons radioactifs : mesure de puretés chimique et radiochimique par HPLC, détermination d'enrichissements isotopiques et d'activités spécifiques par SM, analyse et détermination structurale par RMN liquide comme solide, mesure d’activités radioactives par comptage à scintillation. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
April 14, 2024 5:47 PM
|
IQuaRe, le plein de qualité, d'entraide et de bonne humeur ! IQuaRe, c’est LE réseau qualité du GIS IBiSA. Mais aussi – pour ne pas dire surtout – une grande famille où naturellement, les anciens prennent les plus jeunes sous leur aile. Chaque année, tous se réunissent aux Journées IQuaRe pour faire le point sur les évolutions et les difficultés de leur métier. Sans oublier le plein d’énergie, dans l’ambiance et la convivialité. Lire l’article. IQuaRe, visages et témoignages. « Le réseau IQuaRe et la démarche qualité ont fortement inspiré le fonctionnement global et le quotidien de notre plateforme. Le soutien constant du réseau et sa communauté vivante ont été essentiels à notre succès dans ce domaine. » - Céline Hernandez, Plateforme de séquençage de l’I2BC (PSI2BC). Plus de témoignages. L'appel d’offres Plateformes IBiSA est ouvert jusqu'au 3 avril 2024. Cette année, les modalités privilégient les plateformes engagées dans une démarche 3R ou travaillant sur certains thèmes, comme les PDX, les iPSC et les arthropodes vecteurs. L'édition 2024 est néanmoins ouverte à tous les domaines, et jusqu'au 3 avril. Rendez-vous sur le site d'IBiSA pour candidater. En savoir plus. IBiSA délocalise ses ateliers de formation sur le PGD. A la demande des plateformes lyonnaises, le GIS IBiSA, en partenariat avec l'IFB, organisera bientôt son premier atelier hors Paris. La formation apportera des connaissances et des outils pour bien construire un plan de gestion des données (PGD) et favoriser l'adoption de bonnes pratiques à toutes les étapes d'un projet. En savoir plus. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
March 31, 2024 10:14 AM
|
La Région Ile-de-France contribue régulièrement, et au travers de différents dispositifs, à renforcer les compétences scientifiques et technologiques de son écosystème. A ce titre, elle réédite cette année son AAP SESAME - SESAME 2024 - AAP visant à donner aux laboratoires de recherche de son territoire les moyens nécessaires pour développer de nouveaux projets et pour mettre en œuvre des dispositifs expérimentaux originaux de recherche (comprenez « équipements »). Comme lors des précédentes éditions, ne sont éligibles à SESAME 2024, que des équipements dont le coût total est compris entre 0,2 M€ et 3 M€ HT ; D’autre part, pour rappel, l’aide prendra la forme d’une subvention dont le taux maximum d’intervention régionale pour l’acquisition de ces équipements s’élève à 66% du montant HT du coût total du projet. Ce taux d’intervention régionale pour les projets d’équipements en sciences humaines et sociales pourra néanmoins atteindre 100% du montant HT du coût total, à condition que 3 laboratoires franciliens distincts ou plus participent au projet. Les critères d'éligibilité et de sélection sont disponibles dans le texte de l'appel à projet SESAME 2024. D’autre part, le dossier de candidature est à compléter en ligne sur mesdemarches.iledefrance.fr, par le porteur du projet directement, après sélection par l’Université Paris-Saclay en ce qui nous concerne. Cette sélection se fera naturellement en prenant en compte, outre les critères de la Région mentionnés dans l'AAP, la plus-value collective pour l'Université Paris-Saclay. Attention également de bien prendre les critères d'exclusion mentionnées dans l’AAP, et si cela s'applique à votre projet, de tenir compte des remarques faites par le jury de la Région en cas de soumission les années précédentes. Ci-dessous, le rappel du calendrier, intégrant les échéances propres à l’Université Paris-Saclay : - le mardi 19 mars 2024 : Lancement de l’appel à projets SESAME 2024 ; Les porteurs de projets sont invités à prévenir le plus tôt possible leur Gradiate School/Institut (GS/I) de leur intention de préparer un dossier.
- le jeudi 2 mai 2024 au plus tard: les porteurs de projet transmettent aux GS/I concernés leur projet dans l'état le plus avancé possible, sans oublier la fiche résumé complétée ; Egalement, copie de l’ensemble est envoyé par mail à l'adresse vp.recherche@universite-paris-saclay.fr avec mention de l'institution qui porterait le projet.
- du jeudi 2 mai au vendredi 10 mai 2024 : les GS/I évaluent les dossiers qui leur sont remontés et produisent un avis (classement en trois catégories A, B, C) et le cas échéant les priorisent au sein d'une catégorie s'il y a beaucoup de projets. Le retour des évaluations des GS/I au CoDiReV est attendu pour lemardi 21 mai 2024 au plus tard. Aussi, comme les années précédentes, une journée d’audition des porteurs des projets rattachés au domaine Sciences de la Vie de l’Université Paris-Saclay et à ses interfaces est programmée ; la date pressentie est le mardi 14 mai 2024 (à confirmer).
- autour du vendredi 24 mai 2024 (et au plus tard le vendredi 31 mai 2024), le CoDiRev se réunira, sélectionnera les huit projets que l'UPSaclay est autorisée à faire remonter dans le cadre de cet AAP, et fera parvenir aux porteurs de projets la lettre de soutien de l’Université Paris-Saclay.
- les porteurs de projets auront ensuite jusqu’au lundi 3 juin 2024 (17h00) – date/heure limite de dépôt – pour finaliser leur dossier et le déposer auprès de la Région IdF.

|
Scooped by
Life Sciences UPSaclay
March 17, 2024 5:51 PM
|
L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées. L'IPS2 est la résultante d’une importante restructuration et unification en 2015 de 3 des 4 instituts impliqués dans le Laboratoire d’Excellence / Ecole Universitaire de Recherche Sciences des Plantes de Saclay (SPS). La création de l’'IPS2 a permis de mieux structurer les activités de recherche, de formation et d’innovation en accord avec les objectifs de SPS. L’institut favorise aussi l'implication de ces équipes dans des projets innovants avec des partenaires industriels et/ou des instituts techniques, et accueille différentes start-ups, afin qu'IPS2 contribue à l’agriculture de demain. L'IPS2 héberge quatre plateformes regroupant ainsi différentes installations et différents savoir-faire dédiées aux plantes : la plateforme EPITRANS avec des activités de biologie translationnelle (grandes collections de TILLING en tomate, melon, concombre...) et d’épigénomique (analyses épigénomiques telles que DAP-seq, ATAC-seq et autres) sur les plantes cultivées; la plateforme POPS (transcriptomique) ; la plateforme Interactome (interactomique, interactions protéines-protéines) et la plateforme Métabolime-Métabolome. IPS2 : cliquer ICI Ces plateformes prennent régulièrement RDV avec vous au travers de FOCUS PLATEFORME. Profitez de cette brève pour découvrir à nouveau un éclairage sur leurs expertises, leurs équipements et leurs offres de collaborations ou de prestations ! Contacts : i) IPS2 / EPITRANS ii) IPS2 / Plateforme de transcriptOmique (POPS) iii) IPS2 / Plateforme Métabolisme Métabolome iv) IPS2 / Plateforme Interactomique (SPOmics-Interactome)

|
Scooped by
Life Sciences UPSaclay
March 1, 2024 11:48 AM
|
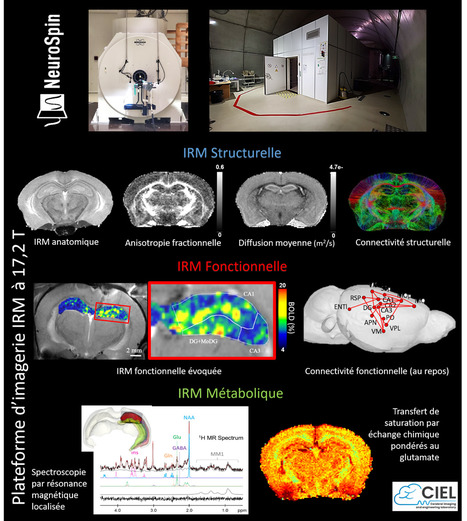
L’utilisation de champs et de systèmes de gradients extrêmes (i.e. champ magnétique supérieur à 7 Tesla pour l’aimant et amplitude supérieure à 500 mT/m pour le système de gradients de champ magnétique) en IRM offre de nombreux avantages, en particulier l’atteinte des résolutions spatio-temporelles d’un ordre de grandeur plus élevées qu’avec un IRM conventionnel, mais aussi l’exploitation de nouveaux mécanismes biophysiques inaccessibles à plus bas champ permettant d’explorer le fonctionnement et le métabolisme cérébral avec une précision inégalée. Soumis à l’appel à projets régional SESAME 2023 - Équipements et plateformes scientifiques et technologiques, pour un budget total de 1 136 k€, TopBrainx17, porté par la plateforme NEUROSPIN / Imagerie in vivo chez le petit animal, ex vivo et in vitro, IRM 17,2 T, fait aujourd’hui partie des 3 projets UPSaclay sélectionnés par la région Île-de-France, et recevra à ce titre une subvention de 750 k€ (le complément étant assuré par le CEA). TopBrainx17 est le seul projet « cœur de métier » Sciences de la Vie (SdV) financé par la région cette année, aux côtés de GEODIA (interface SdV et Sciences & Ingénierie (S&I)) et CRYOVAP (S&I). TopBrain17 permettra de réaliser une jouvence complète de l’électronique de son IRM à 17,2 Tesla dédiée à l’imagerie du petit animal, permettant ainsi à cet instrument unique au monde de se doter des toutes dernières innovations technologiques mises à disposition par le constructeur Bruker BioSpin. Cette jouvence permettra en particulier la mise en œuvre de nouveaux protocoles de neuro-imagerie de pointe, combinant l'imagerie par résonance magnétique fonctionnelle (IRMf) basée sur l’observation du phénomène BOLD (Blood Oxygenation Level Dependent) et ASL (Arterial Spin Labeling), la spectroscopie par résonance magnétique localisée (SRM), le transfert de saturation par échange chimique (CEST) et l'imagerie/spectroscopie du deutérium (2H) appliquées aux neurosciences cliniques et fondamentales. En particulier, il s’agit de mettre à disposition de ses utilisateurs de nouveaux moyens de caractérisation des altérations cellulaires du tissu cérébral (neurones, astrocytes, microvascularisation) au cours du développement et du vieillissement à partir de modèles animaux, en conditions normales ou pathologiques, et également dans le cadre de l’évaluation de nouveaux médicaments. L’utilisation conjointe du champ magnétique extrême à 17,2 Tesla et de la toute nouvelle électronique issue du projet TopBrainx17 fournira des images du cerveau avec des résolutions spatiales et temporelles, un niveau de sensibilité, et une qualité d’image sans précédent. Les protocoles CEST permettront de cartographier les principaux métabolites cérébraux (glucose, glutamate, créatine) et de suivre leur évolution au cours du développement, du vieillissement ou de la neurodégénérescence. En outre, en tirant parti de la sensibilité accrue offerte par la sonde au deutérium refroidie par cryogénie, la plateforme IRM à 17,2 T permettra de développer des stratégies d'acquisition spécifiques pour le suivi in vivo des métabolites marqués au deutérium (lactate, glutamate, glucose ou glycogène), ce qui permettra d'étudier l'équilibre entre le métabolisme énergétique aérobie et anaérobie, ainsi que le cycle de la neurotransmission et le stockage de l'énergie. Tous ces développements contribueront à une meilleure compréhension du fonctionnement et du métabolisme cérébral normal ou pathologique à travers les échelles spatiales et temporelles. Contact : Luisa Ciobanu (luisa.ciobanu@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI NEUROSPIN / Imagerie in vivo chez le petit animal, ex vivo et in vitro, IRM 17,2 T. NeuroSpin dispose d’un imageur 17,2 Tesla de la société Bruker BioSpin. L'IRM 17,2 T est le système d’imagerie préclinique horizontal le plus puissant au monde. L’espace disponible à l’intérieur des gradients (85 mm) permet l’imagerie de petits animaux (souris, rats). En dehors d’un ultra-haut champ magnétique, une autre particularité de ce système est que son gradient de champ magnétique est capable de produire 1000 mT/m (approximativement 15 fois plus puissant que ce qu’il est possible de faire avec des scanners cliniques). Grâce à ce système, nous sommes capables d’acquérir des images avec une résolution isotrope de 25 µm. Nous disposons d’une variété d’antennes radiofréquences adaptées à des applications spécifiques : antennes de surface et antennes volumique pour les cerveaux de rats et de souris et des micro-antennes d’un diamètre allant de 700 µm à 2 mm pour les échantillons et enfin des antennes de surface à noyau-X (13C, 31P) pour la spectroscopie. A propos de NeuroSpin. Neurospin est une infrastructure de recherche sur le cerveau exploitant des grands instruments d'imagerie. NeuroSpin offre à la communauté scientifique publique et privée la possibilité de faire progresser la connaissance du cerveau, et particulièrement du cerveau humain, en proposant un accès à des méthodologies de pointes en imagerie cérébrale et en neuro-informatique. NeuroSpin développe et met à la disposition de la communauté des instruments uniques, notamment en imagerie très hauts champs et dans le domaine des big data. Cette offre s'inscrit dans le cadre des missions spécifiques de NeuroSpin qui sont : i) analyser les fonctions du cerveau humain, leur développement dans l'enfance, et l'impact de la culture et de l'éducation ; ii) identifier les marqueurs et les mécanismes de maladies neurologiques, psychiatriques et neurodéveloppementales ; iii) comparer le cerveau humain et celui d'autres espèces animales ; développer et tester des méthodes d'imagerie à toutes les échelles d'observation : par résonance magnétique (IRM), par électro- et magnéto-encéphalographie (EEG et MEG), et par électrophysiologie massivement parallèle ou l'imagerie photonique et v) développer des logiciels spécialisés dans le traitement et la modélisation des grands jeux de données en neuroimagerie. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.
|

|
Scooped by
Life Sciences UPSaclay
September 18, 2024 10:33 AM
|
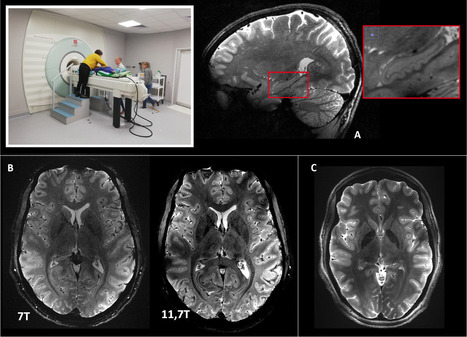
L'IRM dit Iseult, installé à Neurospin (CEA/DRF/JOLIOT, plateforme NEUROSPIN / Imagerie in vivo chez l’homme, ex vivo et in vitro, IRM 11,7T) a livré ses premières images in vivo de cerveau humain. Avec un niveau de champ magnétique de 11.7T, c'est une première mondiale qui vient d'être réalisée. Les images obtenues ont un contraste exceptionnel et un niveau de signal accru permettant d'atteindre des résolutions difficilement envisageables jusqu'ici : [A] Acquisition cerveau entier en pondération T2 à une résolution de 550 microns isotrope. Zoom sur l’hippocampe, structure cérébrale qui joue un rôle central dans la cognition, la mémoire, l'apprentissage et le repérage dans l'espace ; [B] Comparaison d’une acquisition en pondération T2* d’une coupe cérébrale axiale acquise à 7T et sur l’IRM Iseult à 11.7T. La résolution est de 200 microns dans le plan ; [C] Acquisition d’une coupe axiale en pondération T2 à une résolution de 300 microns dans le plan. L'autorisation de mener ces premières expériences avait été obtenue en février 2023 auprès des autorités compétentes : l'agence nationale de sécurité du médicament et des produits de santé (ANSM), et le comité de protection des personnes (CPP). L'objectif de l'étude, appelée "PREMS", était double. D'une part, il s'agissait de vérifier l'innocuité du champ magnétique de 11.7T sur volontaires sains et, d'autre part, de réaliser les premières images in vivo de cerveau humain à 11.7T. 40 volontaires ont été recrutés entre mai 2023 et février 2024. Parmi eux, 20 volontaires ont été soumis à un premier examen fictif, réalisé sans champ magnétique, et 20 autres volontaires ont été exposés au champ magnétique de 11.7T pendant 1h30 pour réaliser l'examen d'imagerie. Les deux groupes de volontaires ont entrepris une série de tests dont des mesures physiologiques, des tests d'équilibre, des tests de l'attention, et ceux avant, pendant et après l'examen. Aucun effet biologique du champ magnétique de 11.7T n'a été observé entre les deux groupes. Une étude de génotoxicité a également pu être menée sur le groupe exposé à 11.7T pour n’identifier aucun effet significatif non plus. Concernant le second objectif de l'étude PREMS, des images IRM anatomiques ont été acquises à l'aide de méthodologies développées au sein du laboratoire sur de nombreuses années utilisant l'IRM 7T déjà installé à Neurospin depuis 2010. Ces méthodologies, pour certaines brevetées par le laboratoire ou pour d'autres publiées dans la littérature scientifique, ont notamment permis d'obtenir des images tridimensionnelles d'une grande qualité couvrant entièrement le cerveau. Plusieurs types de contrastes ont été acquis comme le montre la figure ci-jointe. Parmi ces images, certaines atteignent une résolution spatiale à l'échelle mésoscopique avec une taille de 200 microns dans le plan. Dès lors, une nouvelle étape dans l'exploitation de l'IRM Iseult 11.7T est ouverte. Les équipes sont maintenant en marche pour préparer les premières acquisitions en imagerie fonctionnelle pour voir le cerveau à l'œuvre lors de tâches cognitives. En parallèle, de nouvelles méthodologies vont être implémentées, notamment pour corriger les effets délétères des mouvements involontaires, permettant d'atteindre des résolutions encore plus fines à l'avenir. L’instrument Iseult bénéficie également depuis 2021 d’une contribution gouvernementale importante dans le cadre du Programme d’Investissement d’Avenir France 2030 au travers du projet PRESENCE (ANR-21-ESRE-006) financé via l’action ESR/EquipEx+ et géré par l’Agence Nationale de la Recherche. Ce projet vise d’une part à réaliser la jouvence de certains éléments de l’IRM pour les remplacer par de nouveaux équipements plus performants, et d’autre part à permettre son exploitation et son ouverture la plus large à la communauté scientifique dans une approche de science ouverte. Contacts : Nicolas Boulant (nicolas.boulant@cea.fr); Frank Mauconduit (frank.mauconduit@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, en septembre 2019, la plateforme publiait son premier FOCUS PLATEFORME … Redécouvrez- le aujourd’hui : FOCUS PLATEFORME : 11,7 teslas, record mondial de champ magnétique pour un aimant d'IRM du corps humain ! NEUROSPIN / Imagerie in vivo chez l'homme, ex vivo et in vitro, IRM 11.7T. Reposant sur un design innovant proposé par le CEA/IRFU et sur une électronique de dernière génération fournie par la société Siemens Healthineers, la plateforme d’IRM à 11,7 teslas est dotée d’un aimant de grande ouverture (90 cm) à 11.7 Teslas, d’un système de gradients capables de délivrer 80 mT/m, d’une électronique d’acquisition à 8 canaux en émission et 32 canaux en réception, ainsi que d’une panoplie d’antennes à haute densité en éléments dédiées à l’exploration cérébrale chez l’homme. Cette plateforme permet la réalisation de protocoles d’imagerie anatomique, fonctionnelle, métabolique et de spectroscopie tirant partie du champ magnétique extrême pour atteindre des résolutions spatiales (~100 micromètres), temporelles (~ seconde) ou spectrales inaccessibles à plus bas champ. Cet instrument unique au monde permet également d’exploiter de nouveaux mécanismes de contraste observés à 11.7 teslas et d’explorer les possibilités de l’imagerie moléculaire à haute sensibilité. A propos de NeuroSpin. Neurospin est une infrastructure de recherche sur le cerveau exploitant des grands instruments d'imagerie. NeuroSpin offre à la communauté scientifique publique et privée la possibilité de faire progresser la connaissance du cerveau, et particulièrement du cerveau humain, en proposant un accès à des méthodologies de pointes en imagerie cérébrale et en neuro-informatique. NeuroSpin développe et met à la disposition de la communauté des instruments uniques, notamment en imagerie très hauts champs et dans le domaine des big data. Cette offre s'inscrit dans le cadre des missions spécifiques de NeuroSpin qui sont : i) analyser les fonctions du cerveau humain, leur développement dans l'enfance, et l'impact de la culture et de l'éducation ; ii) identifier les marqueurs et les mécanismes de maladies neurologiques, psychiatriques et neurodéveloppementales ; iii) comparer le cerveau humain et celui d'autres espèces animales ; développer et tester des méthodes d'imagerie à toutes les échelles d'observation : par résonance magnétique (IRM), par électro- et magnéto-encéphalographie (EEG et MEG), et par électrophysiologie massivement parallèle ou l'imagerie photonique et v) développer des logiciels spécialisés dans le traitement et la modélisation des grands jeux de données en neuroimagerie. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
September 8, 2024 11:31 AM
|
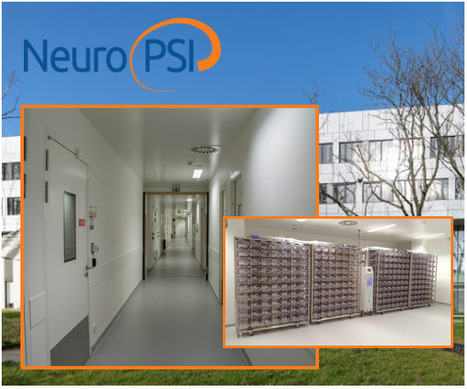
NeuroPSI / Animalerie, aussi connue sous le nom de code « zootechnie 151 », est la toute nouvelle plateforme d’hébergement en place sur le plateau de Saclay (NeuroPSI, RD306 entrée Sud, Allée des Neurosciences, 91191 Gif-sur-Yvette) ! Cette toute nouvelle installation regroupe maintenant sur un site unique, l’ensemble des prestations autrefois dispersées sur Gif-sur-Yvette et Orsay. Hier … env. 1200 m2 d’espace, sur deux sites géographiques distincts, pour des espèces « rongeurs » et des espèces « aquatiques » : i) Campus CNRS de Gif-sur-Yvette, bâtiment 32-33, site connu sous l’appellation « Animalerie Centrale » ; ii) Campus de la Faculté des Sciences d’Orsay, bâtiment 440-447. L’animalerie située à Gif-sur-Yvette (env. 800 m2) se trouvait excentrée de la partie laboratoire, sur le côté du bâtiment 32, et datait de 1998. Le site disposait de 12 pièces d’hébergement (3 dites aquatiques et 9 dites rongeurs). L’animalerie était de plein pied, avec le stockage de nourriture et de litière dans le même bâtiment. Les zones à statut sanitaire étaient bien définies, avec notamment pour la zone EOPS, un SAS pour l’entrée des zootechniciens et un autoclave pour l’entrée du matériel. L’animalerie située à Orsay (env. 400 m2), était dispersée sur plusieurs étages du bâtiment dit 440-447, disposait de 14 pièces d’hébergements (3 dites aquatiques et 11 dites rongeurs), toutes de petites tailles, et pour la plupart, démunies de SAS. Les zones à statut sanitaire n’étaient que partiellement définies, mais le site disposait d’un autoclave pour l’entrée du matériel en EOPS. Le site était aussi équipé de 2 laveries : une au 3ème étage et une au 1er étage. Le stock de litière et nourriture se trouvait au RDC, loin des pièces d’animalerie. Aujourd’hui … env. 2400 m2 d’espace, de plus sur un site unique, et au service exclusivement de rongeurs (souris, rats) et certaines espèces d’oiseaux (canari, diamant mandarin, calopsitte, coq de Bankiva) ; plus de poissons et de xénopes, ces espèces aquatiques sont aujourd’hui gérées par la plateforme TPS-AQUA (ou zootechnie aquatique de TEFOR Paris-Saclay), à quelques mètres seulement puisque également hébergés sur NeuroPSI. Pour la souris, un total de 3 000 cages sont aujourd’hui en service, permettant un hébergement de plus de 10 000 individus (1150 dites conventionnelles ou A1, 1800 réservées en EOPS, une centaine en A2, et près de 250 pour la zone de quarantaine). Pour le rat, ce sont 330 cages avec à nouveau une distribution sous différents statuts sanitaires, permettant l’accueil de près de 900 individus. Enfin, pour les oiseaux, ce sont 7 volières disponibles d’une capacité de 30 individus chacune, et près de 75 cages à taille variable, pour leur reproduction. Le site opère aussi une laverie ultramoderne et de nombreuses annexes (stockage aliments, laboratoires expérimentaux). La laverie dispose de 150 m², avec un zonage en place pour le côté propre et le côté sale. Une pièce donnant sur cette laverie est dédiée au remplissage des cages. La pièce des autoclaves quant à elle, pour l’entrée du matériel en EOPS, donne sur la laverie côté propre. Le stockage de la litière et matériel est située juste à côté de la laverie. Toutes les zones ont deux pièces de stockage dédiées à la zone. La plateforme NeuroPSI / PSI-CO (avec ses pièces dédiées au comportement et chirurgies) est directement reliée par un couloir à la zone conventionnelle où sont hébergés les animaux. Cette plateforme comporte 50 pièces, dont 11 pièces communes qui peuvent être réservées via un logiciel. Contacts : Valérie Lavallée (valerie.lavallee@cnrs.fr) Plug In Labs Université Paris-Saclay : cliquer ICI NeuroPSI / Animalerie (zootechnie 151). Le Service de Zootechnie de NeuroPSI fournit une expertise de haute qualité en technologie des animaux de laboratoire, en particulier pour la recherche en neurosciences. Il est engagé à œuvrer pour faire progresser l’excellence dans la science des soins et du bien-être des animaux de laboratoire, de sorte que ses activités sont dédiées à : i) Assister les chercheurs dans leur mission de recherche de qualité sur l’utilisation des animaux de laboratoire ; ii) Agir en tant que centre de ressources pour les différentes composantes de l’institut sur toutes les questions relatives aux animaux de laboratoire ; iii) Aider les équipes de recherche à atteindre leur objectif de traitement des animaux de laboratoire de manière éthique dans le strict respect des règles ; iv) Fournir une formation et une expertise vétérinaires et du personnel en technologie des animaux de laboratoire.

|
Scooped by
Life Sciences UPSaclay
July 11, 2024 5:43 AM
|
Depuis sa création, le Genoscope – Centre National de Séquençage participe à des projets collaboratifs à fort impact scientifique en fournissant à la communauté scientifique toute l’expertise et les capacités de production et d’analyse de données indispensables à ces projets. Depuis sa contribution majeure, il y a près de 20 ans, au projet Human Genome en publiant la séquence complète du chromosome 14 (Heilig et al., Nature 2003), la plateforme de séquençage située au cœur du biocluster Genopole installé à Évry-Courcouronnes a réalisé le séquençage de nombreuses plantes (vigne, riz, blé…), d’animaux (tétraodon, anophèle…), de champignons (truffe, champignons pathogènes du colza et de la vigne) et de plus d’une centaine de procaryotes. Grâce à sa collaboration avec le Très Grand Centre de Calculs du CEA, situé lui-aussi en Essonne, le Genoscope - CNS a permis de traiter de très grands ensembles de données en utilisant l’informatique haute performance de pointe. Ainsi, depuis sa création, le Genoscope-CNS a contribué à plus de 750 publications et ceci grâce à sa plateforme de séquençage. Très récemment, le Genoscope - CNS a choisi de s’aligner sur la stratégie et les engagements de l'Union Européenne en matière de biodiversité pour 2030 tels que la lutte contre le déclin des pollinisateurs, l'amélioration de l'état de conservation des espèces et des habitats clés, la promotion de la restauration écologique des systèmes forestiers et des rivières, et la minimisation des impacts des espèces invasives. Accompagné d’autres partenaires européens, le Genoscope – CNS participe activement à la recherche en génomique de la biodiversité en Europe visant à étayer la conservation de celle-ci et sa biosurveillance basée sur l'ADN par la génération de génomes de référence de haute qualité provenant de toutes les formes de vie afin d’établir un Atlas européen des génomes de référence (ERGA). L’initiative ERGA est une réponse scientifique paneuropéenne aux menaces actuelles qui pèsent sur la biodiversité. Les génomes de référence fournissent l’aperçu le plus complet des bases génétiques qui constituent chaque espèce et représentent une ressource puissante pour comprendre le fonctionnement de la biodiversité. Alors qu’environ un cinquième des quelques 200 000 espèces européennes sont menacées d’extinction, nous devons agir rapidement et ensemble pour générer à grande échelle des ressources génomiques complètes de haute qualité. Parallèlement à cet important programme européen, le Genoscope - CNS a récemment lancé ATLASea, l’un des projets et équipements prioritaires de recherche (PEPR) exploratoires sélectionnés dans le cadre de France 2030. Copiloté par le CNRS et le CEA et financé à hauteur de 41,3 millions d’euros sur 8 ans, le programme national ATLASea a pour objectif de séquencer le génome de 4500 espèces marines contenant, par conséquent, des millions de gènes encore inconnus à ce jour en visant spécifiquement l’analyse des génomes d'un large éventail d'espèces marines dans tous les groupes eucaryotes présents au sein du littoral français, plus précisément dans sa zone économique exclusive qui s’étend sur plus de 11 millions de Km2, et qui en fait la plus grande du monde, devant celle des Etats-Unis et de l’Australie. Autre actualité importante de la plateforme de séquençage : l’arrivée, courant 2023, d’un séquenceur Revio (PacBio) (en photo) dédié au séquençage des longs fragments et capable de séquencer 1300 génomes humains par an en profondeur 30X, venu renforcer le parc de séquenceurs de la plateforme déjà composé d’un NovaSeq 6000 (Illumina) acquis en 2018, deux MiSeq (Illumina), un PromethION (Oxford Nanopore Technologies), un GridION (Oxford Nanopore Technologies), un MinION Mk1C (Oxford Nanopore Technologies) et un DNBSEQ-G400 (MGI Tech). Pour obtenir des renseignements sur les modalités d’accès de la plateforme de séquençage, n’hésitez pas à contacter Pedro Oliveira ! Aussi, le 24 mai 2021 (déjà !), la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? Contact : Pedro OLIVEIRA (pcoutool@genoscope.cns.fr) Plug In Labs Université Paris-Saclay : cliquer ICI GENOSCOPE / Plateforme de Séquençage - Genopole. Le Genoscope s’est orienté vers l’étude de la biodiversité, à travers le séquençage de novo de grands génomes complets, de métagénomes complexes et plus généralement de collections d’ADN nécessitant une approche de séquençage à très grande échelle. L’équipe du laboratoire de séquençage s’implique dans la prise en main des projets à partir de la réception des échantillons jusqu’à la validation des séquences brutes. Le séquençage est assuré par des séquenceurs très haut débit de deuxième et troisième génération. Ces équipements high-techs permettent d’atteindre un débit autorisant le séquençage de génomes entiers dans un intervalle de temps limité. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 66 entreprises de biotechnologies, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : soutenir les politiques nationales de réindustrialisation et de souveraineté sanitaire, créer et soutenir les entreprises de biotechnologies et le transfert de technologies vers le secteur industriel (notamment pour décarboner la production), favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
June 28, 2024 6:04 PM
|
L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. Cet institut héberge quatre plateformes (En savoir plus ?) regroupant ainsi différentes installations et différents savoir-faire dédiées aux plantes. Dans le présent FOCUS PLATEFORME … c’est au tour de la plateforme POPS (transcriptomique) de nous faire découvrir son expertise. Les plantes mixotrophes sont capables de produire leur énergie non seulement via la photosynthèse mais aussi en trompant leurs champignons mycorhiziens symbiotiques pour obtenir des nutriments. Elles peuvent ainsi ajuster leurs sources énergétiques en fonction des conditions environnementales. Cependant, malgré cet avantage écologique certain, les plantes mixotrophes restent relativement rares dans l’histoire de l’évolution. Pour mieux comprendre ce paradoxe, le MNHN de Paris et l’Université de Gdansk en Pologne ont mené une étude portant sur neuf espèces d’orchidées européennes. En étudiant le transcriptome et le métabolome de ces espèces in situ, ils ont révélé que la plasticité permise par la mixotrophie impose des contraintes spécifiques qui pourraient limiter leur adaptabilité. Ainsi, l’étude du contenu en gènes a révélé un ensemble de gènes spécifiques des orchidées mixotrophes suggérant que la transition de la mixotrophie depuis l’autotrophie impose l’acquisition de fonctions spécifiques. De plus, la physiologie des orchidées mixotrophes montre des contraintes telles qu’une capacité limitée à assimiler le tréhalose fongique ou à se passer totalement de photosynthèse. Enfin, l’étude des réseaux de métabolites suggère que les orchidées mixotrophes ont des capacités adaptatives plus limitées. Ces résultats sont importants pour comprendre la dynamique évolutive des réseaux mycorhiziens. En effet, les plantes parasitant ces réseaux (les espèces mixotrophes et mycohétérotrophes) constituent des menaces à leur stabilité et à leur résilience. Les contraintes liées à l’apparition de ces plantes limitent cette menace et permettent aux champignons mycorhiziens généralistes de se maintenir. Par ce travail qui se poursuit encore actuellement avec l’étude du développement de ces espèces, la plateforme POPS démontre sa capacité à accompagner ses collaborateurs dans des projets « hors du laboratoire » depuis le design expérimental jusqu’à l’interprétation des données produites. Contact : pops.ips2@universite-paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en février 2020 et juin 2023, POPS publiait ses premiers FOCUS PLATEFORME … Redécouvrez- les ! FOCUS PLATEFORME : L'analyse transcriptomique passe au naturel! ; FOCUS PLATEFORME : IPS2 / POPS : Au cœur de la défense des plantes contre les bactéries ! IPS2 / Plateforme de transcriptomique (POPS). La Plateforme de Transcriptomique des Plantes de Paris-Saclay (POPS) propose aux laboratoires académiques ou privés des outils d'analyse du transcriptome par séquençage à haut débit de l'ARN ou RNA-seq. Nous sommes spécialisés dans l'analyse des plantes qu'elles soient modèles ou de culture. Le service proposé inclue la génération des données de RNA-seq, les analyses bio-informatiques et statistiques ainsi que des conseils sur l'interprétation des résultats. Nous accompagnons donc nos collaborateurs depuis le design expérimental jusqu'à l'interprétation des données. La plateforme est certifiée pour l'ensemble de ses activités selon les exigences de la norme ISO 9001 depuis 2012. Afin d'offrir à chaque collaborateur l'analyse la plus adaptée à ses questions biologiques, nous disposons des équipements et des outils d'analyses bio-informatiques et statistiques permettant la construction de transcriptome de novo, l'analyse d'accumulation des ARNs poly-adenylés ou non, des petits ARNs (miRNAs, siRNAs, ribo-seq..) et des ARN poly-adénylés de très petite quantité de départ (jusqu'à 50pg). Pour les analyses d'expression tissu-spécifiques, nous offrons également des analyses de transcriptome après micro-dissection laser ou en cellule unique grâce à la technologie Chromium (10x Genomics). Par ailleurs, nous pouvons offrir à nos collaborateurs de sessions de formation sur une semaine à l'analyse statistique des données RNA-seq. A propos de l’IPS2. L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées.

|
Scooped by
Life Sciences UPSaclay
June 15, 2024 6:18 PM
|
La plateforme de spectrométrie de masse présente au laboratoire LAMBE (Laboratoire Analyse, Modélisation, Matériaux pour la Biologie et l'Environnement, Université d’Évry Paris-Saclay / CNRS / Cergy Paris Université), labellisée par Genopole, se dote d’un photomètre de masse TwoMP (Refeyn), une technologie très récente, pour la caractérisation (bio)moléculaire. Cet équipement, financé par Genopole et la Graduate School de Chimie de l'Université Paris-Saclay, a été installé en novembre dernier sur la plateforme. Le photomètre de masse associé à l’expertise des membres du LAMBE en chimie analytique, analyse structurale et biophysique élargit considérablement les possibilités de caractérisation (bio)moléculaire de notre écosystème, offrant des informations bio-structurales complémentaires de celles obtenues par les autres équipements de caractérisation disponibles sur la plateforme de spectrométrie de masse. Qu’est-ce que la photométrie de masse ? Cette technologie très récente s'appuie sur les principes de la microscopie à réflexion interférentielle et de la microscopie à diffusion interférométrique. C’est une méthode sans marquage qui mesure, en solution, la masse d’objets à l’échelle de la molécule unique. Lorsqu’une molécule entre en contact avec une surface, elle interfère avec le faisceau de lumière réfléchi par cette surface. Cette interférence est ensuite exploitée pour déterminer la masse moléculaire. Régis Daniel, directeur du LAMBE et co-responsable de la plateforme de spectrométrie de masse, indique que « le photomètre de masse TwoMP (Refeyn) permet une mesure rapide (inférieure à 5 min) sur une large gamme de masse (30 kDa à 5 MDa), avec une précision de mesure de ± 2% et une erreur en masse de ± 5%. En outre, cette mesure ne nécessite qu’un faible volume d’échantillon (de 0,1 à 20 µL) et une concentration comprise généralement entre 100 pM et 100 nM, offrant ainsi des caractéristiques inégalées de sensibilité (<< 1 ng pour des protéines), de rapidité de mesure et de consommation d'échantillon. Enfin, c’est une technique non destructive, compatible avec une grande diversité de tampons, permettant ainsi l’étude, dans des conditions natives, d’un large éventail d’objets moléculaires complexes naturels ou synthétiques ». Cet équipement mutualisé répond lui aussi aux enjeux actuels de recherche et de l’innovation biotech des laboratoires et entreprises du biocluster Genopole. Les applications sont très variées, parmi lesquelles celles mises en œuvre au LAMBE et avec des équipes partenaires du biocluster Genopole et de la communauté scientifique sud-francilienne et au-delà, dont : i) La caractérisation de la pureté et de l’homogénéité d’échantillons (suivi de productions d’anticorps thérapeutiques, dégradations de préparations, etc.) ; ii) La mise en évidence des états d’oligomérisation de protéines ou de structures quaternaires spécifiques présentes même minoritaires (enzymes, anticorps, etc.) ; iii) L’analyse d’acides nucléiques (ADN et ARN) sous différentes formes (ribosomiques, plasmides, etc.) et de virus ; iv) La quantification des interactions biomoléculaires de haute affinité (stœchiométrie, KD, etc.) pour différents complexes non covalent (anticorps/antigène, histones/ADN, etc.) ; v) L’étude d’assemblage de complexes (supra-)macromoléculaires (nanocages, nanostructures, ribosomes, virus adéno-associé AAV, etc.). Une palette technologique remarquablement complète sur la plateforme ! « Nouvellement équipés de cette technologie très récente, les membres de la plateforme de spectrométrie de masse de Genopole démontrent une nouvelle fois leur capacité à offrir des services novateurs aux chercheurs et entrepreneurs du biocluster Genopole ainsi qu’à toute la communauté scientifique sud-francilienne étudiant des molécules biologiques complexes pour une meilleure compréhension du vivant, cherchant des composés à visée thérapeutique ou environnementale, ou encore développant des biomatériaux innovants » souligne Julien Picot, Directeur adjoint délégué aux infrastructures et plateformes de Genopole. Pour connaitre les modalités d’accès à ce photomètre de masse, n’hésitez pas, contactez-nous ! Contacts : regis.daniel@univ-evry.fr / Julien.Picot@genopole.fr Plug In Labs Université Paris-Saclay : cliquer ICI GENOPOLE / LAMBE / Plateforme de spectrométrie de masse. La plateforme de spectrométrie de masse offre l'expertise nécessaire au développement de méthodes d'analyses par spectrométrie de masse et chromatographie liquide couplée à la spectrométrie de masse dédiées aux macromolécules biologiques et aux polymères synthétiques. Des analyses protéomiques et glycomiques sont réalisées en routine ainsi que des analyses et dosages de petites molécules. Les domaines d'activités de la plateforme portent principalement sur la caractérisation de complexes protéiques immunopurifiés et l'identification des partenaires d'interaction, ainsi que sur l'étude d'interactions non-covalentes (protéine-protéine, polysaccharides-protéines, ADN-ligand, protéines-peptides, biomolécules-cations métalliques). A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 66 entreprises de biotechnologies, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
June 2, 2024 5:10 PM
|
La plateforme « épigénomique et recherche translationnelle » de l’IPS2 (EPITRANS) est une infrastructure scientifique collective (ISC) de l’INRAE, labellisée IBISA et certifiée Iso9001. Elle a pour mission de développer et de mettre à disposition de la communauté scientifique des outils de génétique haut-débit chez les espèces cultivées et propose d’identifier les gènes et les régulations épigénomiques contrôlant des caractères d’intérêts agronomiques. Elle est l’une des quatre plateformes (En savoir plus ?) que pilote l’IPS2. Les progrès récents dans les approches microfluidiques ont permis ces dernières années le développement du RNA-seq ou de l’ATACseq en cellules uniques (scRNA-seq ; scATACseq). Ces approches offrent l'opportunité unique d'étudier les changements transcriptionnels à l'échelle de la cellule. Dans le cadre du développement de ces approches génomiques, notre plateforme avait la nécessité de s’équiper d’un trieur de cellules pour noyaux et protoplastes, permettant un tri préservant l’intégrité d’échantillons fragiles, d’éviter leur contamination et de minimiser l’impact des tampons de tri sur les cellules d’intérêts. Acheté conjointement avec la plateforme de transcriptomique (IPS2 / POPS) et à l’aide de financements INRAE, du Labex « Saclay Plant Science » (SPS), de l’Université d’Evry Val d’Essonne (UEVE) et d’IBISA, le trieur MACSQuant Tyto de Miltenyi Biotec est le seul cytomètre trieur à proposer un système simple d’utilisation répondant à nos exigences. Il fonctionne en circuit stérile et utilise des cartouches facilement transportables. Cela permet aux utilisateurs de venir avec une cartouche dans laquelle ils auront préalablement injecté leurs échantillons, de réaliser leur tri, et de repartir avec, dans leur cartouche, d’un côté les cellules triées dans le tampon choisi par l’utilisateur, et de l’autre le reste des populations, et le tout dans un environnement stérile. L’utilisation de cartouches offre la possibilité de travailler sur des cellules particulièrement sensibles à l’environnement. Les tris sur le MACSQuant Tyto sont réalisés à très basse pression (environ 3PSI), assurant une meilleure viabilité post-tri et préservant la fonctionnalité des cellules et noyaux triés. Ceci permet d’obtenir des analyses génomiques plus performantes, avec une meilleure représentativité des échantillons de départ. Enfin, compact et sans fluidique, l’instrument permet un gain de place et est complétement automatisé pour une prise en main rapide par les utilisateurs. Enfin, en janvier 2020 et mars 2023, EPITRANS publiait ses premiers FOCUS PLATEFORME… Redécouvrez- les ! Contacts: fabien.marcel@inrae.fr ou marion.dalmais@inrae.fr Plug In Labs Université Paris-Saclay : cliquer ICI IPS2 / EPITRANS. La plateforme EPIgénomique et recherche TRANSlationnelle (EPITRANS), Infrastructure Scientifique Collective (ISC) de l'INRAE, a pour missions de valoriser la recherche fondamentale en transformant les idées en produits et de faire le lien entre le chercheur et le secteur économique. Elle s'intéresse aux allèles et épi-allèles qui améliorent la performance des plantes dans un environnement de plus en plus contraint et plus particulièrement chez les espèces d'intérêt agronomique. Son activité consiste à développer et à mettre à disposition de la communauté scientifique des outils de génétique haut-débit, forward et reverse, chez les végétaux, notamment lorsque les études traditionnelles de génétiques ne peuvent s'appliquer (transformation génétique). Cinq outils sont ainsi proposés : le clonage positionnel par NGS, le TILLING (« targeted Induced Local Lesion IN Genome ») et l'ECO-TILLING, l'Epigénomique et le CRISPR. Le premier permet de cloner des gènes d'intérêts agronomiques pour ensuite étudier leur(s) fonction(s) par TILLING, par recherche de modifications épigénomiques, ou encore par édition du génome via le système CRISPR (selon les espèces). Le TILLING s'appuie sur la production de larges collections (<5000 lignées) de plantes mutées ou de collections de germoplasmes (ECOTILLING) combinée à une identification rapide et systématique des mutations dans les séquences cibles. Ces allèles, non génétiquement modifiés, sont proposés aux sélectionneurs pour améliorer leurs lignées élites en leur offrant une alternative aux collections limitées de germoplasmes. C'est pourquoi, la plateforme a des liens étroits et durables avec le secteur industriel dans différents domaines d'applications (Limagrain, Gautier Semence, Rijk Zwaan, Symrise). EPITRANS est leader en Europe dans le domaine de la recherche translationnelle de par la richesse de ses collections de mutants (260 000 lignées) et par la diversité des espèces cultivées disponibles (13 au total). La plateforme est labellisée par le GIS-IBISA (Infrastructures en Biologie Santé et Agronomie) et est certifiée Iso9001. A propos de l’IPS2. L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées.

|
Scooped by
Life Sciences UPSaclay
May 23, 2024 8:53 AM
|
Dans une étude publiée dans Nucleic Acids Research, les scientifiques de l’Institut des Sciences des Plantes de Paris-Saclay – IPS2 (CNRS/INRAE/UEVE/UPCité/UPSaclay, Gif-sur-Yvette) et de l’unité Génétique Quantitative et Evolution – GQE-Le Moulon (UPSaclay/CNRS/INRAE/AgroParisTech, Gif-sur-Yvette) ont étudié plusieurs caractéristiques épigénétiques sur des séquences uniques et répétitives dans la chromatine des plantes. Les régulations épigénétiques, telles que l'accessibilité de la chromatine, le positionnement des nucléosomes et la méthylation de l'ADN, jouent des rôles cruciaux dans la détermination de la fonction des gènes. Cependant, les méthodes actuelles d'étude de la chromatine, qui reposent sur des technologies de séquençage à courte lecture, peinent à analyser les régions répétitives du génome et ne peuvent pas examiner plusieurs caractéristiques de la chromatine à la fois. Les auteurs ont utilisé le séquençage par nanopores à longue lecture pour obtenir un profilage haute résolution de l'accessibilité de la chromatine marquée par m6A ainsi que de la méthylation endogène des cytosines dans Arabidopsis et le maïs. Grâce à cette approche, ils ont révélé des interactions entre l'accessibilité et la méthylation de l'ADN au sein des gènes, des éléments transposables et des régions centromériques. Ils ont également montré que cette méthode peut être utilisée pour détecter les empreintes de l'ADN dans les régions régulatrices, indiquant où les facteurs de transcription se lient à la chromatine pour contrôler l'activité des gènes. Dans l'ensemble, ce travail fournit une approche puissante pour étudier simultanément plusieurs caractéristiques épigénétiques, offrant ainsi de nouvelles perspectives sur les mécanismes complexes de la régulation des gènes chez les plantes. Contact : leandro.quadrana@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
May 12, 2024 5:27 PM
|
Suite à son installation à Henri Moissan en juillet 2022, la plateforme de cytométrie en flux PLAIMMO de l'Unité Mixte de Service "Ingénierie et Plateformes au Service de l'Innovation Thérapeutique"(UMS-IPSIT) est devenue CYM (Cytométrie Henri Moissan). Le personnel de la plateforme, accompagné par sa référente scientifique, le Pr Géraldine Schlecht, a relancé ses activités pour vous accompagner dans la réalisation de projets de recherche fondamentale ou préclinique, ainsi que dans la mise en place de protocoles de recherche clinique nécessitant l'utilisation d'un cytomètre. Après l’installation des laboratoires, l’objectif premier a été de dynamiser et faire découvrir les possibilités de la cytométrie au sein de l’Université Paris-Saclay. Marie-Laure Aknin, responsable de la plateforme CYM, a sollicité l’ensemble des plateformes de l’écosystème de l’Université Paris-Saclay dans l’objectif de créer un réseau de cytométrie de proximité. La dynamique très positive, apparue dès les premières réunions, a permis de choisir collégialement le nom de ce réseau d’experts, baptisé CyPSay (Cytométrie Paris-Saclay). Ce réseau, créé tout début 2023, regroupe à ce jour 8 plateformes : CYM (ex PLAIMMO, IPSIT / Plateforme de Cytométrie H. Moissan), SpICy (ICP / SpectroImageries et Cytométrie), FlowCyTech (IDMIT, Institut JACOB) et I2BC / Plateforme de cytométrie (IMAGERIE-GIF) d’une part, ensemble qui vient compléter le réseau des plateformes présent sur le biocluster GENOPOLE nommé OCCIGEN. Ce réseau génopolitain regroupe les équipements complémentaires présents sur ImCy (Imagerie-Cytométrie, GENETHON), sur la plateforme de bioproduction (cellulaire, automatisée) de l’I-Stem, au LGRK (Laboratoire de Génomique et Radiobiologie de la Kératinopoïèse, Institut JACOB) et sur la plateforme de tri et clonage de levures opérée par Hybrigenics Services. Le réseau recense aujourd’hui une vingtaine d’équipements dont une quinzaine d’analyseurs : Fortessa (BD Biosciences) ; cytoFLEX S et cytoFLEX LX (Beckman Coulter) ; Partec (Sysmex) ; NovoCyte 3000 (Agilent), MACS Quant 10 et MACS Quant X (Miltenyi) ; SP6800 (Sony), très souvent situés à proximité de trieurs : SH800 (Sony) ; ARIA IIu et ARIA Fusion (BD Biosciences) ; MoFlo AstriosEQ et cytoFLEX SRT (Beckman Coulter). L’objectif du réseau est de partager les expertises respectives des partenaires à travers des réunions régulières, de discuter des évolutions des technologies pour acquérir et analyser des données de cytométrie, d’identifier les besoins en investissements et de se fédérer pour des demandes de financement d’instruments et/ou de logiciels spécifiques. A travers ce Focus Plateforme, nous souhaitons rendre encore plus visible ce réseau proposant un accompagnement optimal de tous vos projets en cytométrie et promouvoir la découverte de cette technologie à travers différentes journées de formation ou séminaires ouvertes à tous. Le réseau a organisé une première journée de formation de cytométrie en flux le 26 octobre dernier à Henri Moissan. Cette journée a été l’occasion de présenter les différentes plateformes du réseau puis de proposer une formation théorique sur les bases de la cytométrie, réalisée par Mr Jean-Baptiste Guillerme de la société Biolegend. Cette journée fut une belle réussite avec environ 40 participants. L’enquête de satisfaction a démontré un bon niveau de satisfaction. Le réseau CyPSay travaille déjà à l’organisation d’une deuxième journée, à laquelle vous serez prochainement invités. Nous espérons vous y retrouver très nombreux ! Pour tout renseignement complémentaire, n'hésitez pas à contacter la plateforme du réseau CyPSay la plus proche. Contact : Marie-Laure Aknin (marie-laure.aknin@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, la plateforme a déjà publié trois FOCUS PLATEFORME, n’hésitez-pas à les relire … IPSIT / Plateforme de Cytométrie H.Moissan (CYM). La plateforme de cytométrie en flux (CYM) de l'Unité Mixte de Service, Ingénierie et Plateformes au Service de l'Innovation Thérapeutique (UMS-IPSIT), située au rez-de-chaussée du bâtiment Recherche Henri Moissan (HM1) offre régulièrement ses services aux équipes de recherche académiques du territoire Paris-Saclay ainsi qu'aux industriels. Le personnel de la plateforme est à votre disposition pour vous aider à la réalisation de projets de recherche fondamentale, préclinique sur des modèles expérimentaux ainsi que pour des protocoles de recherche clinique. Son personnel est aussi à votre service pour la mise au point de nouvelles techniques utilisant la cytométrie en flux. Les équipements de cytométrie en flux de la plateforme permettent le phénotypage des cellules par la détection de molécules membranaires et intracellulaires (biomarqueurs) mais aussi des études fonctionnelles telles que la détection de phosphorylation des protéines, la prolifération cellulaire, la quantification de cytokines ou chimiokines excrétées ou la détection d'ARN. Enfin, des tris cellulaires à haut débit sont aussi proposés par la plateforme. Nos activités qui peuvent être en relations avec celle d'autre plateforme, permettent l'identification de nouveaux biomarqueurs pouvant être des cibles thérapeutiques. A propos d’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 11 plateformes techniques, organisées en trois pôles technologiques (IMCELLF, OMICS et INTERACTIONS) et trois plateformes transverses. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS-et 25 équipes de recherche. Enfin, IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
April 20, 2024 6:28 AM
|
FOCUS PLATEFORME : Appel d’offre interdisciplinaire pour un accès aux plateformes de biologie structurale, d'imagerie biologique et de protéomique
Les trois infrastructures nationales ProFi, FranceBioImaging et FRISBI ainsi que le GIS IBiSA ont le plaisir d'annoncer un 3e appel pour un accès financé aux installations labellisées IBiSA. Notre objectif est de promouvoir la mise en réseau des installations IBiSA par le biais de projets de recherche interdisciplinaires. Les candidatures doivent demander l'accès à au moins deux installations IBiSA différentes dans deux disciplines (biologie structurale, imagerie biologique et protéomique). L'appel est ouvert à tout laboratoire académique. Le projet doit être considéré comme le point de départ d'un projet plus vaste ou comme une étude de validation de concept. Le montant de l'aide financière sera de 5000 € maximum par demande pour couvrir les frais d'installation ; les frais de voyage et d'hébergement ne sont pas éligibles. Les candidatures doivent être soumises à Call-IBISA-FBI-FRISBI-PROFI@i2bc.paris-saclay.fr en utilisant le modèle de document à télécharger ICI. La date limite pour cet appel d'accès inter-infrastructures est le 31 mai 2024. Le texte complet de l’AO peut être téléchargé ICI.

|
Scooped by
Life Sciences UPSaclay
April 6, 2024 5:33 PM
|
La plateforme de Spectroscopie IRTF (Institut de Biologie Intégrative de la Cellule (I2BC), Gif-sur-Yvette, Institut Joliot, CEA, Saclay) met à disposition des utilisateurs un parc de spectromètres Infrarouge à Transformée de Fourier (IRTF) de pointe, équipés de plusieurs accessoires (cellule à transmission, ATR, cellule électrochimique, thermostat, cryostat). L’équipe d’adossement, Photobiologie Photosynthèse Photocatalyse (PPP), a une longue expertise des études mécanistiques de processus biologiques photo-induits, et de photocatalyse. Elle a contribué, en collaboration avec une équipe de l’ICMMO (Institut de chimie moléculaire et des matériaux d’Orsay, CNRS/Université Paris Saclay, Orsay), à décrypter le cycle catalytique d’une porphyrine à fer capable de réduire, de façon efficace et sélective, le CO2 en CO. La réduction du CO2 représente, en effet, un défi sociétal majeur, et on assiste à des progrès notables dans la conception de divers catalyseurs moléculaires pour la réduction électro- et/ou photo-catalytique du CO2. En particulier, les porphyrines à fer sont classées parmi les systèmes les plus actifs pour la formation de CO ou d’acide formique (HCOOH). Bien que le CO ne soit qu'une forme réduite à deux électrons du CO2, il constitue le point de départ du processus de production du syngas et peut donc contribuer de différentes manières à atténuer la production anthropique de CO2. Dans notre système, en utilisant les spectroscopies IR, UV-Visible, EPR et Raman de résonance, nous avons pu montrer que dans la porphyrine de fer étudiée, l'étape d'activation du CO2 est déplacée de l'état redox Fe(0), nécessaire dans d'autres porphyrines à fer, à l'état Fe(I), plus facile à atteindre. Cet effet résulte de l’activation du substrat CO2 par des liaisons hydrogène fournies par les groupes urée présents dans la seconde sphère de coordination du fer, ce qui permet de diminuer l’énergie nécessaire à la catalyse. La spectro-électrochimie infrarouge a donc permis de détecter l’intermédiaire catalytique Fe(II)CO, et, en complémentarité avec les spectroscopies Raman et EPR, également présentes dans le pôle de Biophysique de l’I2BC, a permis la caractérisation complète du processus catalytique. En savoir plus ? Amanullah et al., Angew. Chemie, Int. Ed 2023 Contact : Annamaria Quaranta (annamaria.quaranta@cea.fr); Philipp Gotico (Philipp.Gotico@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Envie de (re)lire leur précédent FOCUS PLATEFORME (13 Juillet 2020) ? FOCUS PLATEFORME : La spectroscopie IRTF différentielle, un outil déterminant pour l'étude des mécanismes mis en jeu dans les réactions de photocatalyse. I2BC / Plateforme de spectroscopie IRTF. La plateforme de spectroscopie IRTF est située au Laboratoire des Mécanismes Fondamentaux en Bioénergétique (UMR 9198). Elle met à disposition des utilisateurs des spectromètres IRTF avancés et elle est équipée pour répondre à la plus grande partie des besoins des analyses IRTF. La plateforme comprend 4 spectromètres avec plusieurs accessoires : cellule à transmission, accessoires ATR, cellule électrochimique, thermostats, cryostats pour expériences à basse température... Elle permet l'étude d'échantillons sous différentes formes (liquide, solide, poudre...). Le laboratoire est spécialisé dans la spectroscopie IRTF différentielle, résolue dans le temps, à basse température, et possède une bonne expertise dans l'étude de réactions biochimiques et photo-induites. La plateforme de spectroscopie IRTF fait partie du pôle des plateformes de Biophysiques de l'I2BC qui comprend les plateformes de RPE, FTIR, Résonance Raman, Spectroscopies Electroniques et Microscopie de fluorescence à super-résolution. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 60 équipes de recherches et 15 plateformes technologiques, provenant de 8 unités de recherches (CGM, IBBMC, IGM, ISV, LEBS, VMS, SB2SM, SBiGeM). L’institut est réparti sur 3 sites de recherche (Campus d’Orsay Vallée de l’Université Paris-Saclay, Campus du CNRS de Gif sur Yvette et Campus du CEA / Centre de Saclay) au sein de 14 bâtiments jusqu’au rassemblement programmé sur le campus du CNRS de Gif-sur-Yvette.

|
Scooped by
Life Sciences UPSaclay
March 23, 2024 10:07 AM
|
Implanté sur le campus INRAE de Jouy en Josas, le plateau de phénotypage in vivo et transparisation est entièrement ouvert à la communauté pour l’étude in vivo de processus biologiques complexes sur modèles rongeurs et poissons. Il regroupe différents équipements de pointe, distribués au sein des animaleries, pour l’imagerie in vivo des animaux en expérimentation (confocal, 2photon et IVIS), la cytométrie en flux de larves de poissons zèbres (COPAS) ou plus récemment l’étude comportementale des poissons (Viewpoint). Les ingénieurs du plateau possèdent également une expertise reconnue en transparisation et imagerie 3D à feuille de lumière déployées sur différents types d’organes (cerveau, poumon, intestin, placenta, ganglion lymphatique, foie... ) isolés de nombreuses espèces (porc, cheval, brebis, souris, truite, carpe et poisson zèbre). Créé en 2012 à INRAE, ce plateau est intégré à l’infrastructure scientifique collective IERP pour l’Infectiologie expérimentale sur modèles rongeurs et poissons, les équipements étant distribués au sein des locaux confinés (NSL2) de la pisciculture expérimentale et de l’animalerie rongeurs. IERP est partie intégrante de l’Institut des Sciences de l’Animal Paris Saclay (SAPS) et de l'infrastructure nationale de recherche EMERG'IN pour la lutte contre les maladies infectieuses émergentes et zoonotiques par des explorations fonctionnelles in vivo. Au cours de cette journée, les scientifiques utilisateurs du plateau de phénotypage in vivo et transparisation présenteront leurs projets, et illustreront ainsi la diversité des expertises et techniques disponibles sur le site. Aussi, cette journée a pour vocation de promouvoir les échanges entre utilisateurs et au-delà puisque l’événement est ouvert à tous. La journée se déroulera en présentiel uniquement. Elle sera éligible au titre de la formation continue en expérimentation animale (une attestation de présence vous sera remise à ce titre). L'inscription est gratuite mais obligatoire ! En savoir plus ? Contact : Manon Mehraz (manon.mehraz@inrae.fr), Maxence Frétaud (maxence.fretaud@inrae.fr) et Christelle Langevin (christelle.langevin@inrae.fr). Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en juin 2020, mars 2021 et mars 2023, l’IERP publiait ses premiers FOCUS PLATEFORME … Redécouvrez- les aujourd’hui ! INRAE / Infectiologie expérimentale des rongeurs et poissons (IERP). L'infrastructure scientifique collective IERP a pour principales missions la production d'animaux à statut sanitaire et génétique défini, la réalisation in vivo d'expérimentation en infectiologie et le phénotypage des animaux infectés par imagerie. Dans le cadre de projets collaboratifs ou de prestations de service, l'infrastructure mène des projets en infectiologie, immunologie, vaccinologie, génétique, physiologie et microbiologie. Distribuée dans plusieurs bâtiments du campus, notre infrastructure réalise des projets multi-espèces au sein d'une animalerie rongeurs pour les modèles souris et rat et d'une pisciculture expérimentale hébergeant les espèces aquacoles (truite et carpe) et poisson-zèbre. Chaque installation dispose de laboratoires de confinements (BSL2/3) équipés de technologies avancées (IVIS, bi-photon) pour le suivi par imagerie des animaux infectés.

|
Scooped by
Life Sciences UPSaclay
March 10, 2024 2:01 PM
|
Hébergée au Laboratoire de génomique et radiobiologie de la kératinopoïèse (LGRK, CEA, Institut Jacob, Institut de radiobiologie cellulaire et moléculaire (IRCM)), la plateforme d’irradiation expérimentale dispose d’un générateur de rayons X Faxitron MultiRAD 350 (PXI/Edimex) financé par Genopole et de l’expertise nécessaire à l’exposition aux rayonnements de matériel biologique et non-biologique. La plateforme d’irradiation expérimentale est un réel atout pour les entités présentes sur le biocluster Genopole et le territoire de Paris-Saclay ! La peau constitue un modèle adapté pour étudier les cellules souches des tissus humain. En effet, plusieurs réservoirs de cellules souches y résident, notamment des cellules souches épithéliales au sein de l’épiderme. Le LGRK, dirigé par Nicolas Fortunel, étudie les propriétés et la capacité régénératrice des cellules souches de la peau, et plus globalement les effecteurs de l’homéostasie cutanée. L’exposition aux rayonnements ionisants est étudiée comme modèle d’exploration des perturbations cutanées induites par l’exposome. Un axe de recherche amont développé au LGRK porte sur la connaissance des caractéristiques fondamentales des cellules souches et progéniteurs épithéliaux de l'épiderme. Les points d'intérêt comprennent la recherche de critères caractéristiques du statut de « cellule souche », ainsi que le décryptage des réseaux de régulation de la balance « immaturité versus différenciation ». Ces recherches intègrent le génome conventionnel codant le protéome, ainsi que les gènes produisant des transcrits non codants. Ces derniers interviennent dans de nombreuses fonctions physiologiques et dans des processus pathologiques. Un axe translationnel mené par le LGRK a pour objectif de générer des innovations dans le domaine de la thérapie cellulaire cutanée. Une piste de travail porte sur le développement d'effecteurs permettant une préservation plus efficace des cellules souches épidermiques ex vivo, dans le contexte de la bio-ingénierie en culture de substituts de peau. Le gain visé concerne la qualité de régénération. Une seconde piste de travail concerne la problématique de rejet immunitaire, qui restreint les champs d'utilisation de greffons allogéniques. L'approche explorée consiste à vectoriser des facteurs promoteurs d'un signal tolérogène, afin de produire des greffons de peau présentant une immunogénicité atténuée. Enfin, le LGRK s’intéresse aux conséquences cutanées des stress génotoxiques induits par les rayonnements ionisants, notamment les expositions de la peau saine inhérentes à des applications médicales (imagerie, radiothérapies). Un premier aspect étudié est l'impact de cet exposome médical sur l'intégrité et les fonctions des cellules souches et des progéniteurs de l'épiderme. Un aspect d'intérêt concerne les fibroblastes dermiques, étudiés pour leur statut cellules effectrices primaires du développement des fibroses cutanées radio-induites. Ainsi, disposer d’un générateur de rayons X tel que le Faxitron MultiRAD 350 (PXI/Edimex) constitue un réel support pour les activités du LGRK, en particulier pour son axe de recherche portant sur la radiobiologie et les radio-pathologies cutanées. Le générateur de rayons X Faxitron MultiRAD 350 (PXI / Edimex), qui permet de délivrer des débits de dose de 100 mGy/min à 7 Gy/min, est accessible à la communauté scientifique et industrielle francilienne. Très récemment, en plus de Sandra Moratille qui est la scientifique référente pour l’utilisation du générateur de rayons X, plusieurs scientifiques référents ont été formés à son utilisation, permettant ainsi une continuité d’activité de prestations de service tout au long de l’année. Pour connaître les modalités d’accès à la plateforme d’irradiation expérimentale, n’hésitez pas, contactez-nous ! Contacts : nicolas.fortunel@cea.fr ou sandra.moratille@cea.fr Plug In Labs Université Paris-Saclay : cliquer ICI IRCM / Plateforme d’irradiation expérimentale - Genopole. La plateforme d’irradiation expérimentale offre les compétences et le savoir-faire nécessaire à l’utilisation d’un générateur de rayons X de dernière génération pour l’irradiation de matériel biologique et non biologique. Les principales applications sont la préparation de cellules nourricières utilisées pour la culture de cellules souches, l’étude fondamentale des lésions occasionnées par le rayonnement X, l’étude des mécanismes de développement des pathologies radio-induites, et les recherches visant à développer des approches médicales personnalisées (ex : irradiation de cellules dans le cadre d’études de la radiosensibilité individuelle). A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à la bioéconomie, Genopole réunit 65 entreprises, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : favoriser l’émergence et la croissance de sociétés de biotechnologie, le transfert d’innovations vers le secteur industriel, le développement de la recherche et l’enseignement supérieur en sciences de la vie. Dirigé par Gilles Trystram, Genopole est un Groupement d’intérêt public principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes technologiques labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, contactez Julien Picot (julien.picot@genopole.fr).
|



 Your new post is loading...
Your new post is loading...



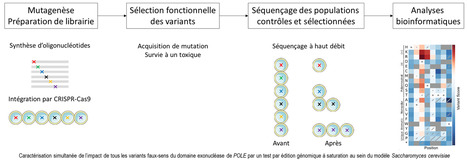


![FOCUS PLATEFORME : [11C]UCB-J / SV2A, ou l’étude de la densité synaptique in vivo par imagerie TEP (Tomographie par Emission de Positons) | Life Sciences Université Paris-Saclay | Scoop.it](https://img.scoop.it/iG8AYYB4Nf1f2-L8ool6Tzl72eJkfbmt4t8yenImKBVvK0kTmF0xjctABnaLJIm9)