Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
December 21, 1:21 PM
|
L’unité mixte de service UMS-IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique, Faculté de Pharmacie, Bâtiment Henri Moissan, Orsay) s’est engagée depuis plusieurs années dans une démarche qualité ambitieuse visant à optimiser la performance de ses plateformes technologiques et à renforcer leur visibilité. Portée par deux référents qualité — Claudine Deloménie (responsable de la plateforme de transcriptomique ACTAGen) et Séverine Domenichini (responsable de la plateforme d’imagerie MIPSIT) — cette initiative structurante a bénéficié en 2020 d’un financement « Moyens de Recherche Mutualisés » (MRM) de 18 k€ accordé par l’Université Paris-Saclay. Ce soutien a permis d’intégrer à la démarche un accompagnement professionnel par un qualiticien expert. Grâce à une mobilisation collective, cette démarche a abouti à la formalisation d’un système de management de la qualité (SMQ) partagé, incluant des indicateurs annuels de performance et de suivi, et à l’identification de priorités stratégiques pour le développement des services de l’unité. Ces efforts ont porté leurs fruits : en 2024, le SMQ de l’UMS-IPSIT a passé avec succès un audit de pré-certification conduit par le Réseau Inserm Qualité. Ce résultat marque une étape clé vers l’objectif de certification ISO 9001, envisagée dès 2026. Ce succès collectif témoigne de l’engagement constant de l’équipe de l’IPSIT en faveur de l’excellence opérationnelle et de l’amélioration continue de l’UMS. -> Contacts : Valérie Domergue (valerie.domergue@universite-paris-saclay.fr) / Claudine.Delomenie (claudine.delomenie@universite-paris-saclay.fr) / Séverine Domenichini (severine.domenichini@universite-paris-saclay.fr) Plug-In-Labs Université Paris-Saclay : cliquer ICI A propos de l’IPSIT. L’IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes techniques, organisées en deux pôles technologiques (IMCELLF et OMICS) et quatre plateformes. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi ses FOCUS PLATEFORME décrivant toutes ses expertises !

|
Scooped by
Life Sciences UPSaclay
December 16, 4:41 PM
|
Le traitement de tumeurs radio-résistantes telles que le glioblastome multiforme demeure un défi majeur, aggravé par leur forte composante immunosuppressive. La radiothérapie (RT) exerce un rôle immunomodulateur en induisant à la fois des effets immunosuppresseurs et immunostimulateurs. La nature de ces effets dépend de plusieurs paramètres, notamment la dose totale, la dose par fraction, le mode de délivrance de la dose et la durée du traitement. Il est généralement admis que l’hypofractionnement tend à favoriser une stimulation du système immunitaire. Toutefois, son utilisation reste limitée dans les tumeurs volumineuses, comme les gliomes, en raison d’un risque élevé de toxicité. Dès lors, l’identification de nouvelles stratégies capables d’induire des réponses immunitaires plus favorables tout en réduisant les effets indésirables sur les tissus sains apparaît essentielle pour améliorer l’efficacité des traitements anticancéreux. Dans ce contexte, une étude parue dans Cancer Immunology Research, menée dans l’équipe Nouvelles Approches en Radiothérapie - NARA du Laboratoire signalisation, radiobiologie et cancer (UMR 3347 CNRS/UMR-S 1021 INSERM/Institut Curie/UPSaclay, Orsay), s’intéresse à l’évaluation de la réponse immunitaire antitumorale induite par la radiothérapie par mini-faisceaux de protons (pMBRT), dont les effets immunomodulateurs restent encore imparfaitement caractérisés. Une analyse approfondie de la réponse immunitaire à une dose curative de pMBRT a été réalisée dans un modèle préclinique orthotopique de glioblastome chez le rat. Les résultats mettent en évidence une association étroite entre la pMBRT et l’activation du système immunitaire. En particulier, la pMBRT induit une augmentation plus marquée de la densité lymphocytaire intratumorale que la protonthérapie conventionnelle. L’analyse transcriptomique unicellulaire a permis d’identifier plusieurs populations immunitaires ainsi que des signatures transcriptionnelles spécifiques au sein des cellules immunitaires tumorales après pMBRT, caractérisées notamment par une augmentation de la production d’anticorps, de l’expression de cytokines chimiotactiques et des réponses de type interféron. Ces observations soulignent le rôle central de l’immunité adaptative, et en particulier des lymphocytes T, dans le mécanisme d’action de la pMBRT. La capacité de la pMBRT à induire une réponse immunitaire antitumorale efficace en une seule séance de radiothérapie, tout en préservant les tissus sains, en fait une approche prometteuse pour de futurs essais cliniques et pour des stratégies combinant radiothérapie et immunothérapie. -> Contact : yolanda.prezado@curie.fr / yolanda.prezado@usc.es

|
Scooped by
Life Sciences UPSaclay
December 16, 4:58 PM
|
Dans une étude publiée dans PNAS, l’équipe Dynamique des Chromosomes de l’Institut des Sciences des Plantes de Paris-Saclay - IPS2 (CNRS/INRAE/UEVE/UPSaclay, Gif-sur-Yvette) met en évidence l’existence d’un mécanisme global de répression de la transcription chez la plante modèle Arabidopsis thaliana. À l’aide d’un crible génétique, les chercheurs ont montré que la protéine LUMINIDEPENDENS (LD) intervient dans la réponse au stress réplicatif. L’analyse de ses cibles, réalisée par immunoprécipitation de la chromatine et des approches transcriptomiques normalisées par spike-in, révèlent que LD agit comme un régulateur négatif de la transcription à l’échelle du génome. L’étude de ses partenaires protéiques suggère que LD module la transcription par divers mécanismes, notamment la modification des histones, la phosphorylation de l’ARN polymérase II ou encore son recrutement sur la chromatine par le complexe Mediator. Ainsi, LD apparaît comme un régulateur global de la transcription, dont le rôle dans la réponse au stress réplicatif pourrait s’expliquer par une réduction des conflits transcription-réplication, sources majeures de stress réplicatif et d’instabilité génomique. -> Contact : cecile.raynaud@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 16, 5:09 PM
|
Dans une revue de la série « Darwin Reviews » du Journal of Experimental Botany, les chercheurs de l’équipe Carbone, Allocation, Transport et Signalisation - CATS de l’Institut Jean-Pierre Bourgin - Sciences du Végétal - IJPB (INRAE/AgroParisTech/UPSaclay, Versailles), en collaboration avec l’équipe du Pr H. E. Neuhaus (Université de Kaiserslautern, Allemagne) et l’équipe Métabolisme (META) de l’UMR Biologie du Fruit et Pathologie (BFP, INRAE Bordeaux), proposent une synthèse approfondie sur les transporteurs de sucres tonoplastiques. Cette revue a pour objectif de consolider un corpus croissant de connaissances et d’examiner plus en profondeur les mécanismes moléculaires de régulation qui gouvernent ces transporteurs clés. Elle s’appuie également sur une méta-analyse de jeux de données transcriptomiques issus de RNA-seq bulk et single-cell, afin d’évaluer le potentiel de ces transporteurs en tant que cibles pour renforcer la résilience des plantes face au changement climatique, en particulier dans le contexte de l’élévation du CO₂ atmosphérique. Enfin, cette étude offre une nouvelle perspective soulignant l’importance stratégique d’étudier les transporteurs de sucres du tonoplaste afin de développer des approches innovantes pour renforcer la tolérance des plantes aux stress environnementaux. -> Contact : rozenn.le-hir@inrae.fr

|
Scooped by
Life Sciences UPSaclay
December 17, 4:03 PM
|
Dans une étude publiée dans Nature Communications, des épidémiologistes du laboratoire « Épidémiologie et modélisation de l’échappement aux anti-infectieux » de l’Institut Pasteur, rattaché au Centre de Recherche en Epidémiologie et Santé des Populations – CESP (INSERM/UVSQ/UPSaclay), en collaboration avec les Instituts Pasteur de Madagascar et du Cambodge, se sont intéressés au portage d’entérobactéries résistantes aux antibiotiques chez les nouveau-nés de ces deux pays, en particulier celles productrices de β-lactamases à spectre étendu. Ces bactéries sont responsables d’une morbi-mortalité élevée chez les nouveau-nés, une population particulièrement vulnérable aux infections. Les difficultés de prise en charge sont encore accrues dans les pays à revenu faible ou intermédiaire, en raison d’un accès limité aux antibiotiques de dernier recours. En comparant les génomes chromosomiques et plasmidiques des entérobactéries portées par les mères au moment de l’accouchement et par leurs nouveau-nés au cours des trois premiers jours de vie, les chercheurs ont montré que seulement 16,5 % des colonisations néonatales pouvaient être attribuées à une origine maternelle. Ces résultats suggèrent un rôle majeur d’autres sources de transmission, notamment nosocomiales, qui restent à identifier précisément. Ces conclusions contrastent avec une grande partie de la littérature existante, principalement issue de pays à haut revenu, et soulignent l’importance de mieux comprendre les voies de transmission dans les contextes à ressources limitées afin de mettre en place des stratégies de prévention efficaces, notamment en matière d’hygiène. -> Contact : annelisebeaumont@gmail.com

|
Scooped by
Life Sciences UPSaclay
December 21, 12:47 PM
|
Des chercheurs du Service de Chimie Bioorganique et de Marquage (SCBM, Département Médicaments et technologies pour la Santé, Institut Joliot, CEA, Centre de Paris-Saclay) ont développé une nouvelle version de leur vecteur nanométrique micellaire destiné à l’imagerie tumorale par résonance magnétique du fluor-19. Cette version doit permettre d’augmenter le contraste IRM en éteignant le signal à la demande dans certaines zones, tout en le préservant dans les zones pathologiques à imager. Ce travail a été mené en collaboration avec le laboratoire CIEL de BAOBAB (NeuroSpin). Lire la suite de l’Actu CEA Joliot et l’article paru dans Nanoscale Horizons. -> Contact : eric.doris@cea.fr / edmond.gravel@cea.fr / sebastien.meriaux@cea.fr

|
Scooped by
Life Sciences UPSaclay
December 21, 12:56 PM
|
Décerné par l’Académie Nationale de médecine de France, ce Prix remis à Béchir Jarraya le 16 décembre 2025 vient saluer l’excellence de ses travaux de recherche, ainsi que son engagement constant au service de l’innovation médicale. Béchir Jarraya, PU-PH à l'UFR Simone Veil -Santé, en neurochirurgie à l’hôpital Foch et chercheur en neurosciences, a effectué ses études de médecine à Lyon avant de réaliser son internat à Paris. Titulaire d’une thèse de science au CEA, il a enrichi son parcours par un séjour à Harvard, avant de fonder une équipe Inserm Avenir à NeuroSpin (CEA) puis une unité de neuromodulation à l’hôpital Foch. Actuellement, il dirige une équipe INSERM au sein de l’unité dirigée par Stanislas Dehaene à NeuroSpin (CEA). Ses recherches visent à restaurer la conscience après un coma, grâce à la stimulation cérébrale profonde du thalamus chez des primates anesthésiés, avec des applications cliniques prévues à l’hôpital Foch. Lauréat du prix Liliane Bettencourt pour les jeunes chercheurs et du prix MANDE de l’Académie Nationale de Médecine en 2011 pour ses travaux sur la thérapie génique appliquée à la maladie de Parkinson, il est également membre de l’Office parlementaire d’évaluation des choix scientifiques et technologiques. Le Prix Emile Delannoy-Robbe de l'Académie Nationale de Médecine est attribué à un chirurgien pour ses travaux de recherche scientifique. -> Contact : bechir.jarraya@cea.fr

|
Scooped by
Life Sciences UPSaclay
December 19, 5:09 PM
|
À 62 ans, le Professeur Marc Humbert est une figure singulière de la médecine française. Pneumologue et spécialiste mondial de l’hypertension artérielle pulmonaire (HTAP), ce médecin-chercheur se consacre à une maladie qui ne touche que 15 à 50 personnes par million d’habitants. Son travail a récemment abouti à la découverte d’un traitement révolutionnaire et à la remise de plusieurs prix prestigieux. Installé dans son modeste mais lumineux bureau de doyen de la faculté de médecine de l’université Paris-Saclay, Marc Humbert affiche un grand sourire : « Mes fenêtres donnent sur le laboratoire de recherche, la faculté et le service de pneumologie sont à deux pas. Cette position géographique reflète parfaitement mon engagement : être au carrefour de l’enseignement, de la recherche et du soin. Lire la suite de l’article dans Le Point (sur abonnement) -> Contact : marc.humbert@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 21, 12:41 PM
|
La Graduate School Health and Drug Sciences de l'Université Paris-Saclay organise un cycle de conférences sur la "Transition écologique du secteur du médicament : enjeux et solutions", donné par Edouard Dossetto, Professeur attaché en développement soutenable de la GS et directeur de projet transition écologique à la Direction Générale des Entreprises. Ce cycle de trois conférences se déroulera le vendredi 9 janvier 2026, de 14h à 15h, le jeudi 15 janvier 2026, de 14h à 15h et le mardi 17 février 2026, de 14h à 15h. - Vendredi 9 janvier 2026, 02:00 pm - 03:00 pm
- Lieu : Bâtiment Henri Moissan

|
Scooped by
Life Sciences UPSaclay
December 21, 12:17 PM
|
À l'attention de la communauté universitaire,
Du 7 janvier au 1er avril 2026, l'Université Paris-Saclay organise les midis de L’Entrepreneuriat.
Il s'agit d'une série de webinaires gratuits sur l'émergence d'un projet entrepreneurial.
Vous souhaitez en découvrir plus sur l'entrepreneuriat, le développement d'un projet, la création de start-up ? Vous avez envie d'échanger avec des professionnels de l'écosystème entrepreneurial, des alumni, des doctorants ou encore des étudiants entrepreneurs qui sont engagés dans leur projet ?
Les Midis de l'Entrepreneuriat sont faits pour vous ! C'est une série de webinaires d'une heure (de 12h30 à 13h30) sur l'émergence d'un projet. L'objectif principal de cet évènement est d'échanger de manière informelle entre les participants et des experts sur une thématique.
Programme - 7 janvier : Une technologie optique deeptech pour révolutionner le traitement des maladies cutanées
- 15 janvier : Le modèlé économique
- 21 janvier : Un projet entrepreneurial qui automatise la création de semelles sur-mesure pour les rendre accessibles à tous
- 28 janvier : Débuter sans argent
- 4 février : Une plateforme pour aider les étudiants et jeunes actifs à maîtriser simplement leurs finances
- 11 février : Les étapes de la création d'entreprise
- 25 février : Des ateliers culturels avec des animations fun et des activités fédératrices
- 11 mars : Une deeptech qui décarbone grâce à l'optimisation de la photosynthèse et transforme la biomasse en engrais organique
- 18 mars : Des enzymes mangeuses de polluants
- 25 mars : Une approche pour transformer une simple présence en ligne en véritable aventure digitale
- 1er avril : La démarche d'innovation
S'inscrire

|
Scooped by
Life Sciences UPSaclay
December 21, 12:01 PM
|
Assemblée Générale annuelle de la GS HeaDS - 13 janvier 2026
Nous avons le plaisir de vous convier à l’Assemblée Générale annuelle de la Graduate School Health and Drug Sciences de l’Université Paris-Saclay qui se tiendra le : Mardi 13 janvier 2026 | 12h30 à 13h30 Amphithéâtre D Bâtiment Henri Moissan 6 17 avenue des sciences 91 400 ORSAY Également en visioconférence via ce lien L’Assemblée Générale sera suivie d’un moment de convivialité autour d’un café.

|
Scooped by
Life Sciences UPSaclay
December 21, 12:24 PM
|
Prix Léon Velluz d’un montant de 10 000€ Prix biennal à décerner en 2026, sans aucune considération d’âge, de position ou de nationalité, à l’auteur d’une découverte de chimie ou de biochimie organiques intéressant la thérapeutique humaine. Il ne pourra être partagé, si ce n’est en cas de nécessité, au sein d’une même équipe de travail devant être tenue pour l’auteur collectif de la découverte.

|
Scooped by
Life Sciences UPSaclay
December 21, 11:56 AM
|
La Graduate School (GS) « Health & Drug Sciences » (HeaDS) coordonne les forces de recherche et de formation dans le domaine de l’innovation thérapeutique, du médicament et des produits de santé. La GS lance son appel à projets « New Collaborations ». Cet AAP s’adresse aux ingénieurs, chercheurs, enseignants-chercheurs permanents contribuant par leurs activités aux thématiques de la GS HeaDS (médicaments, produits de santé, diagnostic et innovation thérapeutique). L’AAP « New Collaborations » cherche à favoriser l’obtention de résultats préliminaires en vue de répondre à d'autres sources de financement. Il vise à soutenir et à promouvoir la recherche en innovation thérapeutique en initiant de nouveaux projets collaboratifs (« New Collaborations ») entre au moins 2 équipes de recherche de la GS (hors plate-forme). Le financement sera au maximum de 35 000 € par projet. -> Contact : contact.gs-heads@universite-paris-saclay.fr
|

|
Scooped by
Life Sciences UPSaclay
December 16, 4:38 PM
|
L'émergence de Clostridioides difficile comme principale cause d'infections intestinales nosocomiales associées aux soins de santé nécessite de nouvelles stratégies thérapeutiques, en particulier dans le contexte de la résistance aux antibiotiques et des infections récurrentes. La phagothérapie est une approche prometteuse, mais son application clinique contre C. difficile reste entravée par notre manque de compréhension des facteurs déterminant la spécificité de l'hôte. En effet, il est essentiel de comprendre comment les phages interagissent spécifiquement avec leur hôte afin de pouvoir sélectionner les meilleurs candidats pour la préparation d'un cocktail. La protéine SlpA, principale protéine de la couche de surface, est un récepteur clé du phage, mais les détails moléculaires déterminant les interactions entre le phage et le récepteur restent flous. Dans une étude publiée dans PLoS Pathogens, les chercheurs de l’Institut de biologie intégrative de la cellule – I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette), en collaboration avec les collègues Canadiens de l'Université de Sherbrooke, ont disséqué les caractéristiques structurelles de SlpA nécessaires à l'infection par les phages grâce à des isoformes SlpA modifiées et à des modifications de domaine, et démontré comment des régions spécifiques de SlpA interviennent dans l'adsorption et l'infection par les phages. Ces nouvelles connaissances sur les interactions entre les phages et les récepteurs seront déterminantes pour orienter la conception future de phages thérapeutiques à large spectre d'hôtes. -> Contact : olga.soutourina@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 16, 4:48 PM
|
Les agrégats de protéines alpha-synucléine et tau sont systématiquement retrouvés chez les patients atteints des maladies de Parkinson et d'Alzheimer. Les évènements menant à leur agrégation ne sont pas bien compris, malgré de nombreuses descriptions structurales à haute résolution in vitro, ou chez des modèles animaux à plus basse résolution.
Dans leur étude publiée dans Journal of the American Chemical Society, des scientifiques du département de Biochimie-Biophysique-Biologie Structurale de l'I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) ont développé et utilisé une nouvelle approche expérimentale, pour caractériser à l'échelle atomique les formes solubles de ces protéines en cellule vivante. Les auteurs ont utilisé la RMN pour caractériser des cellules vivantes insérées dans le spectromètre, et plus particulièrement pour observer sélectivement alpha-synucléine et tau exprimées juste auparavant dans un milieu contenant des acides aminés enrichis en 13C-15N. La RMN a en effet deux atouts : elle est non-invasive, et fournit de l’information au niveau atomique. Ainsi, la RMN dite "in-cell" permet d’observer des processus chemo-biologiques dans des systèmes vivants, et de faire le pont entre in vitro et in vivo. Ici, les auteurs ont observé l'existence d'alpha-synucléine et tau à l'état "désordonné", c'est-à-dire sans structure tridimensionnelle fixe. Ils ont pu également cartographier les zones d'interaction avec des partenaires cellulaires (lipides, chaperons, tubuline, ...). Cette étude a montré la faisabilité de l'approche dans des cellules modèles HEK. L'équipe développe actuellement des modèles similaires en cellules souches pluripotentes pour mener ces caractérisations RMN avec des organoïdes-cérébroïdes. Ceux-ci, produits à partir de fonds génétiques variés, seront de bons modèles pour comprendre l'évolution de ces maladies neurodégénératives. -> Contact : francois-xavier.theillet@cnrs.fr

|
Scooped by
Life Sciences UPSaclay
December 16, 5:05 PM
|
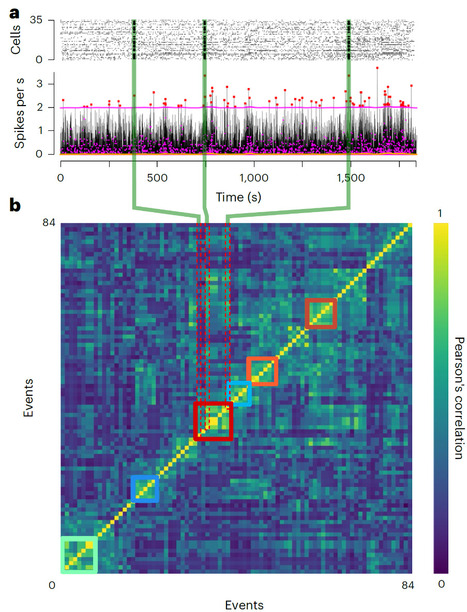
Dans une étude publiée dans Nature Neuroscience, les chercheurs de l'Institut des neurosciences Paris-Saclay - NeuroPSI (CNRS/UPSaclay, Gif-sur-Yvette) ont utilisé des ensembles de données comprenant des enregistrements de l'activité des neurones corticaux ainsi que la cartographie complète de leur connectivité structurelle. Aucune prédiction de la théorie de la fonction corticale en termes d'attracteurs, telle que les "pattern completion units", n'a été trouvée dans les données. Les réseaux corticaux semblent plutôt fonctionner selon des flux continus d’information, qui peuvent être reproduits par de simples modèles de réseaux neuronaux récurrents, sans avoir recours aux attracteurs. -> Contact : alain.destexhe@cnrs.fr

|
Scooped by
Life Sciences UPSaclay
December 16, 5:12 PM
|
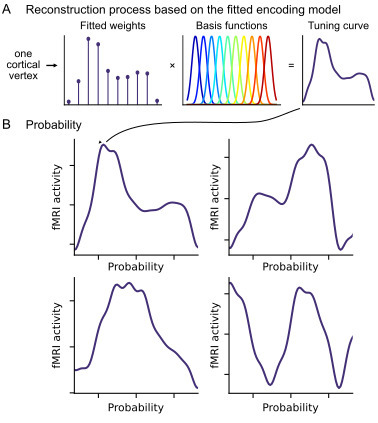
Dans une étude publiée dans Nature Communications, des scientifiques de l’Unité de recherche en Neuroimagerie Cognitive - Unicog (UPSaclay/Inserm/CEA/CNRS, Gif-sur-Yvette) de Neurospin ont identifié un code cérébral original pour représenter la probabilité du prochain événement dans une séquence d’observations. À l’aide d’une IRM fonctionnelle à très haut champ (7 Tesla), ils montrent que des régions fronto-pariétales encodent cette probabilité. Les profils de réponse découverts sont non monotones, contrairement à l’hypothèse souvent faite : différentes parties du cortex cérébral répondent préférentiellement à différentes plages de probabilité, plutôt que de simplement augmenter ou diminuer leur activité avec celles-ci. Dans cette étude, les participants estimaient, au fil d’une séquence d’images A/B, la probabilité cachée d’apparition de chaque image. Un modèle bayésien normatif permet d’estimer, essai par essai, la probabilité cachée et la confiance associée à cette estimation. Le code neuronal de ces quantités a ensuite été établi en combinant deux méthodes d’analyse : celle du modèle linéaire généralisé, et de l’approximation par des fonctions de bases. Les résultats révèlent une différence fondamentale entre le code neuronal de la probabilité et celui de la confiance : alors que la confiance est encodée de façon essentiellement monotone dans un vaste réseau fronto-pariétal, la probabilité des événements neutres suit un code hautement non monotone. Ce travail suggère que le cerveau utilise un code neuronal particulier pour représenter les probabilités, similaire à celui utilisé pour représenter les nombres et les fractions. Ce code permet de représenter des probabilités abstraites, ouvrant de nouvelles perspectives sur la manière dont l’humain anticipe les événements du monde. -> Contact : florent.meyniel@cea.fr

|
Scooped by
Life Sciences UPSaclay
December 17, 4:24 PM
|

|
Scooped by
Life Sciences UPSaclay
December 16, 4:32 PM
|
Portrait Jeune Chercheuse – Elsa Bernard, chercheuse en oncologie computationnelle
Elsa Bernard est chercheuse à Gustave Roussy, où elle dirige le laboratoire d’Oncologie Clinique Computationnelle au sein de l’UMR-S 981 (INSERM/UPSaclay/Gustave Roussy, Villejuif). Avec son équipe, elle développe des approches computationnelles de pointe au service de l’oncologie de précision. Après une formation pluridisciplinaire en géosciences et mathématiques à l’École Normale Supérieure (Ulm), elle obtient un Master 2 en probabilités et statistiques à l’Université Paris VII, puis un doctorat en biologie computationnelle en 2016, réalisé entre les Mines ParisTech et l’Institut Curie sous la direction du Pr Jean-Philippe Vert. Sa thèse, consacrée au développement de méthodes d’inférence de l’épissage alternatif et leurs applications en cancérologie, ancre durablement son intérêt pour l’oncologie computationnelle. Elle rejoint ensuite le Memorial Sloan Kettering Cancer Center (2016–2021) pour se spécialiser dans la génomique du cancer, en particulier des leucémies myéloïdes. Ses travaux conduisent à l’identification de biomarqueurs diagnostiques et pronostiques majeurs, notamment la caractérisation de l’état allélique de TP53, désormais intégré à la Classification 2022 de l’OMS, ainsi qu’au développement de systèmes innovants de stratification du risque et de classification moléculaire (IPSS-M, nouvelle taxonomie moléculaire des myélodysplasies) aujourd’hui largement adoptés en pratique clinique internationale. De 2021 à 2023, elle poursuit son parcours au Francis Crick Institute (Londres, Royaume-Uni) au sein du laboratoire du Pr Charles Swanton. Elle y dirige un projet sur l’impact de l’hématopoïèse clonale sur le microenvironnement tumoral et la progression des tumeurs pulmonaires. Ces travaux, combinés à ses contributions sur la transition d’états précurseurs (hématopoïèse clonale) vers des néoplasies myéloïdes, se situent au carrefour de la biologie du vieillissement, de la génomique évolutive et de l’écologie tumorale, avec le potentiel d’ouvrir la voie à de nouvelles stratégies thérapeutiques. Depuis janvier 2024, Elsa a rejoint Gustave Roussy en tant que cheffe de groupe ATIP-Avenir. Son laboratoire s’intéresse à la détection précoce et l'interception des cancers secondaires, notamment liés aux traitements anticancéreux, au rôle de l’hématopoïèse clonale dans la progression tumorale et la réponse aux traitements, ainsi qu’au développement d’outils prédictifs basés sur la biopsie liquide (ADN tumoral circulant) pour anticiper réponse, rechute, et évolution des maladies. Ce programme de recherche bénéficiera en 2026 du soutien d’une ERC Starting Grant. “Prediction is very difficult, especially of the future” - Niels Bohr -> Contact : elsa.bernard@gustaveroussy.fr

|
Scooped by
Life Sciences UPSaclay
December 21, 11:38 AM
|
Avec - Marie Darrieussecq, écrivaine
- Grégoire Danger, maître de conférences au Laboratoire de physique des interactions ioniques et moléculaires de l’Université d’Aix Marseille/CNRS. Chercheur associé au Laboratoire d’Astrophysique de Marseille (LAM).
- Purificación López-García, biologiste, directrice de recherche CNRS au Laboratoire Ecologie Société Evolution - ESE (CNRS/UPSaclay/AgroParisTech, Gif-sur-Yvette) de l’Université Paris Saclay.
Podcast de l’émission -> Contact : puri.lopez@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 17, 4:15 PM
|
Le prix Rozén Linné de Botanique est décerné à Catherine Bellini en reconnaissance de ses travaux de recherches et de son investissement pour la promotion de la recherche internationale. Ces prix sont décernés tous les trois ans à deux chercheurs méritants en botanique et zoologie résidant en Suède Les missions de longue durée et les mises à disposition constituent à INRAE des dispositifs de mobilité stratégiques favorisant le développement de collaborations scientifiques au sein de laboratoires partenaires, tant en France qu’à l’étranger. Ces dispositifs représentent un levier majeur pour l’établissement et la consolidation de partenariats de recherche pérennes, notamment dans le cadre de projets de coopérations internationales. C’est dans ce contexte que Catherine Bellini a pu étendre ses activités de recherche au sein d’un laboratoire « hors les murs » mis en place dès 2005 entre l’Umeå Plant Science Centre -UPSC et INRAE. Directrice de recherche INRAE à l’Institut Jean-Pierre Bourgin - Sciences du Végétal - IJPB (INRAE/AgroParisTech/UPSaclay, Versailles) et professeure à l'UPSC (Université d'Umeå, Suède), Catherine Bellini conduit des recherches sur deux domaines différents. A l’UPSC, son équipe étudie le développement des racines adventives, une étape clé et limitante dans la propagation végétative d'espèces d'arbres économiquement importantes. A l’IJPB, dans l'équipe "Carbone, allocation, transport, signalisation" CATS dirigée par Sylvie Dinant, elle s'intéresse au transport du sucre et à l'allocation du carbone, ainsi qu’à leur rôle dans le développement des plantes et leur interaction avec l'environnement. Lire la suite de l’Actu IJPB -> Contact : catherine.bellini@umu.se / catherine.bellini@inrae.fr

|
Scooped by
Life Sciences UPSaclay
December 21, 11:48 AM
|
Dans le cadre de l'Objet Interdisciplinaire METABIODIVEX de l'Université Paris-Saclay, une journée scientifique autour des deux thématiques Biocatalyse et Valorisation de la Biomasse est organisée vendredi 16 janvier 2026 dans l'amphithéâtre de l'ICSN à Gif-sur-Yvette. La matinée sera dédiée à la Biocatalyse et aux Enzymes en Action et l'après-midi aux Approches de Valorisation de la Biomasse. Pour chacune de ces thématiques, deux conférences plénières sont programmées. Ces conférences encadreront 4 FlashComm de jeunes chercheurs du périmètre Paris-Saclay. Des communications d'acteurs industriels et des acteurs de la valorisation des résultats scientifiques sont également programmées En fin de journée, des tables rondes seront organisées afin de pouvoir échanger autour de ces deux thématiques. Cette journée scientifique gratuite est ouverte à toute personne présentant un intérêt pour l’une de ces deux thématiques. Le programme de cette journée scientifique et les inscriptions sont disponibles sur le site de SciencesConf à l’adresse : https://metabiodivex26.sciencesconf.org/ Deux FlashComm pour chacune de deux thématiques sont proposées. N’hésitez pas à postuler en écrivant aux organisateurs (erwan.poupon@universite-paris-saclay.fr / aurelien.alix@universite-paris-saclay.fr / jean-francois.betzer@cnrs.fr). L'Institut de Chimie des Substances Naturelles (ICSN) est situé sur le campus CNRS au 1 avenue de la Terrasse à Gif-sur-Yvette. L'amphithéâtre est situé au 1er étage du bâtiment 27. L'ICSN est facilement accessible à pied depuis la gare du RER B Gif-sur-Yvette. Pour plus d'informations sur l'accès au campus CNRS et à l'ICSN, rendez-vous sur la page Informations Générales / Accès.

|
Scooped by
Life Sciences UPSaclay
December 21, 12:06 PM
|
La santé mentale est l'affaire de tous et toutes, et il nous appartient d'en prendre soin. L'Université Paris-Saclay propose de nombreuses actions tout au long de l'année. La Graduate School Health and Drug Sciences, la Graduate School Sport, Mouvement et Facteurs Humains, la Faculté de Pharmacie et la Faculté des Sciences du Sport s'associent à la mairie du Plessis-Robinson pour la 4e édition du symposium grand public : "Sport et santé mentale, quand l'impact devient une force" Mercredi 8 avril 2026 à 19h00 Maison des Arts du Plessis-Robinson Le programme, en cours d'élaboration, sera transmis ultérieurement. L'inscription est gratuite mais obligatoire (dans la limite des places disponibles) -> Contact : lea.remaud@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 21, 12:31 PM
|
La Fondation d’Entreprise Bristol Myers Squibb pour la Recherche en Immuno-Oncologie soutient des projets innovants et d’intérêt général visant à faire progresser la recherche, les parcours de soins et la qualité de vie des patients en oncologie.

|
Scooped by
Life Sciences UPSaclay
December 21, 12:10 PM
|
La Loi de programmation de la recherche pour les années 2021 à 2030 prévoit de « reconnaître, valoriser et encourager l’engagement des acteurs de la recherche dans les liens entre science et société ». Il est précisé dans son annexe qu’INRAE, en lien avec le Ministère en charge de la Recherche, décernera chaque année un Prix pour récompenser des travaux de recherche menés selon une démarche participative.
|





 Your new post is loading...
Your new post is loading...