Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
February 1, 11:22 AM
|
Aujourd’hui, un FOCUS PLATEFORME en lien avec le SESAME 2025, dont les résultats globaux pour Paris-Saclay vous ont été présentées le 1er décembre dernier. La plateforme PICCEL (Plateforme d’Imagerie et de Criblage Cellulaire, anciennement plateforme PARi) du Département Radiobiologie Cellulaire et Moléculaire (DRCM, UMR SGCSR Université Paris-Saclay, Université Paris-Cité, INSERM, CEA Centre de Fontenay-aux-Roses (DRF, Institut Jacob)) accompagne depuis de nombreuses années les équipes académiques et industrielles françaises dans la mise en œuvre de criblages cellulaires à haut débit. Son expertise et ses technologies ont contribué à l’identification de nouveaux biomarqueurs, voies de signalisation et petites molécules impliqués dans divers processus biologiques physiologiques ou pathologiques En savoir plus sur les travaux du laboratoire ? Forissier V et al., Theranostics. 2025 ; Rouault CD et al., Nat Commun. 2025 ; Ariey-Bonnet J et al., EBioMedicine. 2023 ; Pina et al., Methods Cell Biol. 2022 ; Villa et al., Cell Death Dis. 2020 ; Lebraud E et al., Nucleic Acids Res. 2020. Grâce à un financement SESAME Île-de-France porté par Karine Dubrana et Guillaume Pinna, la plateforme se dotera en 2026 d’un microscope à haut contenu et à ultra-haut débit, unique en France, renforçant sa capacité à explorer les dynamiques cellulaires à grande échelle. Cet équipement positionnera la plateforme PICCEL parmi les infrastructures nationales de pointe en imagerie cellulaire multiparamétrique et ouvrira la voie à de nouvelles approches de caractérisation fonctionnelle pour la recherche biomédicale. Il permettra notamment la recherche de nouvelles molécules radio-sensibilisantes et/ou radio-protectrices des tissus sains utilisables en radiothérapie pour lutter contre les cancers. L’équipement sera un système d’imagerie par épifluorescence ultra-rapide conçu pour relever les défis émergents du criblage phénotypique multiparamétrique. Il permettra l’acquisition d’images en microplaques (jusqu’au format 1536 puits) dans quatre canaux de fluorescence ainsi qu’en lumière transmise. Le système sera entièrement automatisé grâce à son intégration avec un robot passeur de plaques, assurant la gestion complète du flux expérimental, depuis le stockage des microplaques jusqu’à leur acquisition et leur analyse. Associé à un logiciel avancé d’analyse d’images, il permettra le phénotypage cellulaire multiparamétrique. Dans cette configuration, le système pourra imager et quantifier jusqu’à 300 plaques 384 puits par jour, levant ainsi le principal verrou de débit des approches de crible par imagerie traditionnelles et permettant l’exploration de tests cellulaires complexes, multiplexés ou en cinétique multipoints. Cet équipement offrira à la plateforme PICCEL la capacité d’interroger une large variété de banques de dimensions et de natures très diverses (banques de petites molécules, banques CRISPR ou ARN interférentes) pour répondre à des questions biologiques jusque-là hors de portée. La plateforme PICCEL verra ses capacités techniques profondément transformées, donnant accès à ses partenaires à un outil unique en France pour l’imagerie cellulaire multiparamétrique à haut contenu. Au-delà des criblages, l’équipement soutiendra de nombreux projets en biologie cellulaire, radiobiologie, immunologie ou cancérologie, et permettra la formation de jeunes chercheurs à l’imagerie automatisée et à l’analyse quantitative. La plateforme PICCEL est ouverte à l’ensemble de la communauté scientifique académique et industrielle. Pour toute demande ou projet, n’hésitez pas à nous contacter. -> Contact : Guillaume Pinna (Responsable Plateforme PICCEL) — guillaume.pinna@cea.fr; Karine Dubrana (Directrice des plateformes DRCM) — karine.dubrana@cea.fr Plug In Labs Université Paris-Saclay : cliquer ICI DRCM / Plateforme d'Imagerie et Criblage Cellulaire (PICCEL). La Plateforme d'Imagerie et Criblage Cellulaire (PICCEL), située dans le Département de Radiobiologie Cellulaire et Moléculaire (DRCM, CEA), est dédiée au criblage à haut débit (HTS) et à haut contenu (HCS) sur modèles cellulaires. La plateforme dispose de l’expertise et de l’infrastructure nécessaires pour la conception, la miniaturisation et l’automatisation de tests cellulaires, le criblage systématique et parallèle de collections de perturbateurs biologiques (siARNs, CRISPRi/a, miRNAs, petites molécules, composés médicaments…), la quantification de systèmes rapporteurs simples ou multiplexés, ainsi que l’analyse statistique de jeux de données de grande dimension. L’imagerie à haut contenu constitue notre approche privilégiée pour le profilage phénotypique, incluant des mesures multiplexées à partir de marquages fluorescents ou label-free. En complément de ses activités de criblage, PICCEL met également à disposition ses équipements robotiques et d’imagerie pour des applications de caractérisation et d’exploration cellulaire de routine. La plateforme travaille en étroite collaboration avec les autres plateformes du DRCM pour proposer des solutions adaptées à chaque projet. L’équipe adopte une démarche qualité rigoureuse, garantissant la traçabilité et la confidentialité des résultats tout au long des projets de criblage.

|
Scooped by
Life Sciences UPSaclay
Today, 5:29 AM
|
La 7e édition de notre Parcours de formation "VALORISATION DE PROJETS DE RECHERCHE INNOVANTS" démarrera le 10 février 2026. Ouverts aux personnels et aux doctorants de l'Université, 6 modules sont proposés afin de se former à la valorisation de la recherche et à la gestion d'un projet de recherche innovant : MODULE 1 ― INTRODUCTION A LA VALORISATION Découvrir la valorisation des résultats de recherche MODULE 2 ― PROTEGER SES RESULTATS Protéger les résultats de la recherche par la PI MODULE 3 ― PARTENARIATS et INGENIERIE CONTRACTUELLE Conduire des projets collaboratifs en partenariat MODULE 4 ― MARKETING DE L’INNOVATION Identifier le potentiel technico-économique de son projet MODULE 5 ― LE TRANSFERT DE TECHNOLOGIES Transférer ses résultats de recherche à la société MODULE 6 ― LA CREATION D’ENTREPRISE Développer son projet de start-up (en lien avec le Parcours Entrepreneuriat Deeptech) - 09.04.2026/10.04.2026 (EN)
- autres dates : 05.05.2026/06.05.2026 (FR)
Les modules sont animés par des professionnels de la valorisation (chargés d’affaires valorisation et juristes de l’Université Paris-Saclay, experts en entrepreneuriat et transfert, collaborateurs de la SATT Paris-Saclay). A chaque session, les participants ont l'opportunité de mettre en pratique les notions et compétences développées au cours de la journée, via des études de cas ou mises en situation. Ces modules sont également l'occasion de développer son réseau, d'identifier les ressources et partenaires sur lesquels s'appuyer. Le parcours complet se déroule de février à mai 2026, à raison de 2 jours par mois pour un total de 6 modules. Les modules sont indépendants et complémentaires. Les doctorants bénéficient par ailleurs d'une labellisation dans le cadre d'une participation à l'ensemble des modules. Informations complémentaires Inscriptions : via GEFORP (05-Former aux métiers de gestion) ou ADUM.

|
Scooped by
Life Sciences UPSaclay
Today, 5:10 AM
|
Registration is now open for a half-day Master Class on “Model-Based Clustering and Classification (MBCC)” on 19 March 2026 from 9:30 a.m. to 1:00 p.m. on the AgroParisTech campus. Model-based clustering and classification methods allow for the discovery of structure in complex data and the assignment of observations to latent groups. These methods are widely used in statistics and machine learning, with applications ranging from social sciences to genomics. This half-day master class will introduce the main ideas underlying model-based clustering and classification, with an emphasis on mixture models, likelihood-based inference with the EM algorithm, and probabilistic interpretation of clusters and classes. Lectures and R-code demonstrations will be delivered in English by Professor Brendan Murphy (University College Dublin), an internationally recognized expert in statistical modeling applied to a wide range of domains, including agri-food, biomedical and social sciences. PhD students and early career researchers in applied statistics with familiarity in linear models and R are particularly encouraged to attend. Registrations are open until February 27, 2026: https://mbcc2026.sciencesconf.org Note that registration is free but mandatory and subject to validation by the organizers, due to a limited number of seats. Funding for the Master Class has been provided by the PHC Ulysses INTEGRATE project. The MBCC Master Class organization committee

|
Scooped by
Life Sciences UPSaclay
Today, 5:01 AM
|
Le Fonds Marion Elizabeth Brancher propose une bourse doctorale pour financer une thèse dans les domaines cardiaque ou vasculaire, ainsi qu’une bourse de prolongation de thèse. Le prix Marion Elizabeth Brancher, doté de 5 000 €, récompensera un chercheur ou une chercheuse pour la qualité de ses travaux en cardiologie ou recherche vasculaire.

|
Scooped by
Life Sciences UPSaclay
Today, 4:43 AM
|
Le prochain Rendez-Vous BioAnalyse aura lieu jeudi 19 février 2026. Nous échangerons autour de la présentation de Mathias Mericskay (UMR-S 1180 Inserm/UPSaclay, Orsay) sur le thème « Vitamine B3 et métabolome cardiaque et systémique ». Ce sera dans la nouvelle salle de séminaire à AdvanThink, bâtiment Euclide du parc des Algorithmes (Route de l’Orme, 91190 Saint-Aubin). Inscription

|
Scooped by
Life Sciences UPSaclay
February 4, 12:07 PM
|
Sous l’impulsion d’Emmanuel Macron, la France tente d’ériger à Villejuif un incubateur géant pour aider les scientifiques à transformer leurs idées en médicaments. C’est vrai que l'on s’y perd dans ces couloirs où rien n’est indiqué, au milieu de toutes ces salles nues, qui attendent encore qu’on les habille de meubles et de moquettes. Le "Paris-Saclay Cancer Cluster", projet d’incubateur inédit en France, n’est pas tout à fait fini, et cela se devine à tous ces salariés le nez en l’air, qui piquent une tête aux intersections, avant de vite faire demi-tour. Même le responsable des lieux, le très influent Eric Vivier, immunologiste de référence, hésite, hume un couloir, ouvre une porte puis la referme, pas encore tout à fait maître en son royaume, construit en un éclair. En 2021, ce scientifique d’Aix-Marseille, à l’allure vive et au phrasé soutenu, s’est vu confier la transformation de ce lopin de terre au sud de Paris, coincé entre l'autoroute A6 et l’immense centre de lutte contre le cancer Gustave Roussy. Emmanuel Macron voulait en faire une structure pour accompagner les jeunes entreprises en oncologie, et développer de nouveaux médicaments contre cette pathologie, qui touche de plus en plus de Français. C'est quasiment chose faite : en quelques mois, trois immeubles dans le style brique et murs épais du New York des années cinquante ont poussé. Alors que tous les étages ne sont pas encore ouverts, des entrepreneurs de tous horizons se bousculent déjà pour bénéficier de précieux conseils. Lire la suite de l’article de l’Express

|
Scooped by
Life Sciences UPSaclay
February 4, 8:27 AM
|
Fabrice André, directeur de la recherche de Gustave Roussy, oncologue spécialiste du cancer du sein, professeur à l’université Paris-Saclay et Karin Tarte, cheffe du service d'immunologie au CHU de Rennes, présidente du Comité d’orientation de la recherche (COR) de la Fondation ARC sont nos invités. La France est le pays qui a le plus grand nombre de cancers détectés par habitant. "C'est plus de 400 000 patients diagnostiqués de cancers", rappelle Fabrice André. "Les principaux cancers sont le cancer du sein chez les femmes, de la prostate chez les hommes, cancers du poumon, cancers du côlon. Il faut rappeler qu'entre 40 et 50 % des cancers sont évitables ! On ne le rappellera jamais assez, 20 % sont liés au tabac. Ensuite il y a l'alcool comme facteur de risque, et d'autres qu'on pourrait avoir par la suite. Et en France, il faut le souligner, il n'y a pas assez d'adhérence aux programmes de dépistage." Fabrice André, directeur de la recherche de Gustave Roussy, oncologue spécialiste du cancer du sein, professeur à l’université Paris-Saclay et Karin Tarte, cheffe du service d'immunologie au CHU de Rennes, présidente du Comité d’orientation de la recherche (COR) de la Fondation ARC sont nos invités. Réécouter l’émission du 4 février 2026 -> Contact : fabrice.andre@gustaveroussy.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 9:39 AM
|
Le sommeil joue un rôle majeur dans le maintien des apprentissages en mémoire. Ce processus, appelé « consolidation », confère stabilité et persistance aux informations et compétences acquises en journée, et serait dépendant de la réactivation lors du sommeil des mêmes réseaux de neurones que ceux mobilisés lors de l’apprentissage. Parmi ces réactivations lors du sommeil figurent des ondes rapides générées par une zone cérébrale profonde, le thalamus. Ces ondes, appelées fuseaux du sommeil, présentent la particularité d’apparaître soit isolées, soit regroupées en « trains ». Dans une étude publiée dans Communications Biology, des chercheurs du laboratoire Complexité, innovation, Activités Motrices et Sportives - CIAMS (UPSaclay, Orsay), en collaboration avec le Centre du Sommeil et de la Vigilance (AP-HP, Hôpital Hôtel-Dieu, Paris), ont exploré le rôle différencié de ces deux types de fuseaux du sommeil dans la consolidation d’un apprentissage moteur. Après la pratique répétée d’une tâche motrice, certains participants de l’étude ont eu le bras immobilisé pendant une journée afin de modifier leur expérience sensorimotrice. Les chercheurs ont ensuite étudié la nuit suivante pour mesurer l’effet de cette immobilisation sur les rythmes du sommeil. Les résultats montrent que la rythmicité des fuseaux du sommeil demeure stable malgré la perturbation sensorimotrice. En revanche, leur synchronisation avec d’autres grands rythmes du sommeil est altérée spécifiquement pour les fuseaux regroupés en trains. Des effets complémentaires sont retrouvés : alors que les fuseaux en trains permettent de consolider les apprentissages grâce à des réactivations successives et rapprochées des réseaux neuronaux engagés lors de la pratique, les fuseaux isolés pourraient quant à eux permettre de remodeler ces apprentissages afin de les transférer à de nouvelles situations. Ces travaux éclairent sur la manière dont le cerveau arbitre entre stabilisation et flexibilité des apprentissages et pourraient avoir avec des implications notamment pour la rééducation de pathologies neurologiques. -> Contact : arnaud.boutin@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 9:21 AM
|
La prophase de la première division méiotique est une étape clé durant laquelle les chromosomes homologues s’apparient et établissent des liens physiques essentiels à leur ségrégation correcte. Durant cette phase, les chromatides sœurs sont organisées en boucles ancrées à un axe chromosomique formé par les complexes de cohésine et des protéines spécifiques de la méiose, notamment Hop1 et Red1. Toutefois, les interactions fonctionnelles entre les complexes de cohésine et les protéines de l’axe, ainsi que leur coordination avec les différents événements de la prophase I, demeurent encore imparfaitement caractérisées. Dans une étude parue dans PLOS Genetics, les scientifiques de l’équipe Meiotic Recombination and Pairing de l’I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) ont étudié les interactions fonctionnelles et épistatiques entre Hop1, Red1 et plusieurs composants et régulateurs du complexe cohésine (Rec8, Scc1/Rad21, Spo76/Pds5, Wapl et Sororine) chez le champignon modèle Sordaria macrospora. L’analyse de mutants nuls simples et multiples a permis de révéler un réseau hiérarchique contrôlant le recrutement, la stabilité et la dynamique des complexes de cohésine le long de l’axe chromosomique. Les auteurs montrent que la fonction de Wapl dans la régulation des cohésines est conservée chez Sordaria et que Spo76/Pds5 constitue une cible majeure de son activité. De manière inattendue, les protéines de l’axe Hop1 et Red1, en coopération avec Sororine, assurent une protection stade-spécifique de Spo76/Pds5 contre l’activité dissociative médiée par Wapl au cours de la prophase I. Enfin, les résultats soulignent le rôle central de Spo76/Pds5 dans la stabilité des kléisines, qu’il protège d’une dégradation protéasomale dépendante de Slx8/STUbL. Ce travail met en évidence l’importance d’un dialogue dynamique et finement régulé entre les protéines de l’axe chromosomique et les complexes de cohésine pour préserver l’intégrité de l’axe, coordonner les événements majeurs de la prophase I et assurer une progression correcte de la méiose. -> Contact : stephanie.boisnard@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 8:44 AM
|
Le bisou magique anti-bobo, ça marche ?
France 5 aborde le concept du « bisou magique », souvent utilisé par les parents pour apaiser la douleur de leurs enfants. Didier Bouhassira, neurologue et directeur de l’unité Physiopathologie et Pharmacologie Clinique de la Douleur à l’Inserm (UMR-S 987 Inserm/UVSQ/UPSaclay/APHP/CHU Ambroise Paré, Boulogne Billancourt), confirme son efficacité en expliquant que « le bisou magique est un des moyens de mettre en jeu les systèmes de contrôle de la douleur dans notre cerveau ». Le bisou agit comme un placebo, son effet étant influencé par les attentes de l’enfant. Bien que le bisou magique soit utile, il ne remplace pas les traitements médicaux nécessaires. Les parents jouent un rôle crucial dans la gestion de la douleur de leurs enfants. Ecouter l’émission sur France 5 -> Contact: didier.bouhassira@inserm.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 5:00 AM
|
L'intérieur des cellules est encombré d’organelles, de macromolécules, du cytosquelette et de nombreuses autres structures. Et pourtant, pour remplir leurs fonctions biologiques quotidiennes, les cellules doivent veiller à ce que diverses biomolécules soient efficacement transportées d'un endroit à un autre au sein de l’espace intracellulaire. Comment ces structures influencent-elles la diffusion des molécules à l'intérieur des cellules ? Peut-on comprendre ce mécanisme de transport moléculaire à l'aide d'une approche ingénierie ? Pour répondre à ces questions, des chercheurs en mécaniques des fluides (Laboratoire d’Energétique Moleculaire et Macroscopique, Combustion - EM2C, CNRS/CentralSupélec/UPSaclay, Gif-sur-Yvette) et en biologie cellulaire (Institut Jacques Monod, Paris ; Universität Erlangen, Allemagne) ont collaboré pour mesurer la distribution des obstacles et le coefficient de diffusion d'une molécule fluorescente dans différentes zones de cellules. Sur la base de ces observations, ils ont développé un modèle de transport en milieux poreux capable d'expliquer l'interaction entre les structures et la diffusion des molécules à l'intérieur des cellules. Ils ont notamment mis en évidence qu'une quantité similaire à la porosité cellulaire était un paramètre clé pour prédire la diffusion des molécules passives, indépendamment de leur emplacement dans la cellule ou du type de cellule. En montrant des similarités entre l'intérieur d'une cellule et un milieu poreux, ces travaux, publiés dans PNAS, aident à mieux comprendre la diffusion intracellulaire, un mécanisme au cœur de processus cellulaires importants tels que la migration des cellules métastatiques. -> Contact : morgan.chabanon@centralesupelec.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 4:33 AM
|
Portrait Jeune Chercheur – Florian Touitou, Maître de conférences en alimentation animale
Florian Touitou est maître de conférences en alimentation animale à l’École nationale vétérinaire d’Alfort (EnvA) et y enseigne depuis 2024. Animé à la fois par une curiosité scientifique qui le pousse à comprendre les mécanismes du vivant dans toute leur complexité et par un goût prononcé pour la transmission et la scène, il s’est naturellement orienté vers une carrière d’enseignant-chercheur. À ses yeux, enseigner revient à incarner le savoir et à le rendre accessible, à la manière d’un comédien face à son public. Docteur vétérinaire, diplômé de l’École Nationale Vétérinaire de Toulouse (ENVT) et titulaire d’un doctorat en infectiologie, physiopathologie, toxicologie, génétique et nutrition délivré par l’Institut National Polytechnique de Toulouse (INP-T), il a réalisé sa thèse au sein de l’UMR GenPhySE où ses travaux portaient sur la compréhension des mécanismes sous-jacents de l’efficience alimentaire chez des jeunes agneaux. Cette approche intégrative combinait l’étude du microbiote ruminal, acteur clé des premières étapes de la digestion des aliments chez les ruminants, l’analyse de son activité par l’intermédiaire du métabolome lipidique et des métabolites polaires issus de la dégradation des aliments ainsi que l’étude du métabolisme de l’hôte. Parallèlement à ses travaux de recherche, il dispensait des cours et travaux dirigés en alimentation animale, en particulier des ruminants, à l’ENVT. Après avoir travaillé sur la communauté microbienne ruminale et ses liens avec les phénotypes des ovins, son intégration dans l’unité de Biologie de la Reproduction, Environnement, Epigénétique et Développement - BREED (INRAE/UPSaclay, Jouy-en-Josas) dirigée par Pascale Chavatte-Palmer lui a permis de mettre ses compétences en nutrition au service de l’étude de la programmation fœtale et des mécanismes épigénétiques influençant le développement des gamètes, de l’embryon et la santé de la descendance. Son arrivée à l’EnvA, d’abord comme contractuel d’abord puis comme maître de conférences, lui a offert l’autonomie nécessaire pour bâtir un programme de nutrition fondamentale s’articulant avec les enseignements de ses collègues afin de préparer au mieux préparer les étudiantes et étudiants à leur futur métier de vétérinaire. Au sein de l’UMR BREED, il a rejoint l’équipe DREAM (Développement, Reproduction des Ruminants, Épigénétique, Adaptations et Modélisation) participant à plusieurs projets s’intéressant à l’influence de l’environnement (en particulier nutritionnel) sur l’épigénétique et les phénotypes associés aux performances de reproduction. « Entre ce que je pense, ce que je veux dire, ce que je crois dire, ce que je dis, ce que vous avez envie d’entendre, ce que vous entendez, ce que vous comprenez… il y a dix possibilités qu’on ait des difficultés à communiquer. Mais essayons quand même. » - Bernard Werber -> Contact : florian.touitou@vet-alfort.fr

|
Scooped by
Life Sciences UPSaclay
January 27, 10:17 AM
|
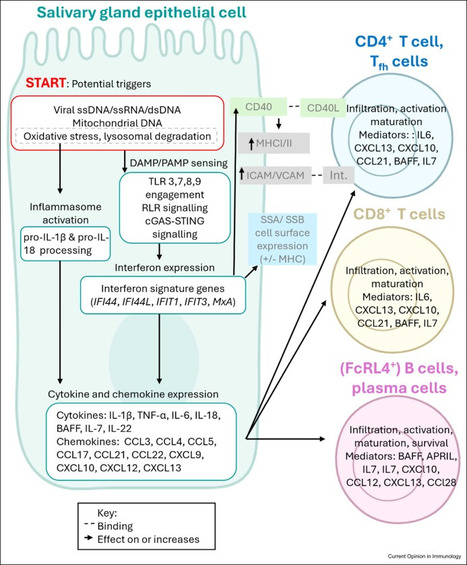
La maladie de Sjögren (SjD) est une maladie auto-immune systémique caractérisée par une inflammation chronique des glandes salivaires et lacrymales, entraînant une sécheresse buccale et oculaire et pouvant se compliquer d’atteintes d’organes multiples. La cellule épithéliale est au centre de la pathogénie et il est essentiel de pouvoir mieux modéliser les interactions entre l’épithélium et le système immunitaire. Une revue récente publiée dans Current Opinion in Immunology et coordonnée par Gaetane Nocturne de l’UMR-S 1184 Center for Immunology of Viral Infections and Autoimmune Diseases-IDMIT (UPSaclay/Inserm/CEA, AP-HP, Le Kremlin-Bicêtre) met en lumière le potentiel des organoïdes de glandes salivaires (SGOs) pour modéliser l’épithélite auto-immune dans la SjD. Ces structures 3D, dérivées de cellules souches de patients, reproduisent fidèlement l’architecture et les fonctions des glandes salivaires humaines. Elles permettent d’étudier l’activation de l’épithélium, les interactions épithélium-immune, les défauts fonctionnels. Ces modèles offrent une plateforme innovante pour le criblage de médicaments : tester l’efficacité de thérapies ciblant les voies de l’interféron (ex. inhibiteurs de JAK) ou les interactions épithélium-immune (ex. anti-BAFF, anti-CD40). Bien que prometteurs, les SGOs doivent encore intégrer des composantes immunitaires et stromales pour mieux mimer l’environnement pathologique. Mais demain, leur utilisation pourrait s’étendre à la compréhension des atteintes systémiques du SjD (reins, poumons) et ouvrir la voie à une approche de médecine personnalisée avec le développement de modèles "patient-spécifiques" pour prédire la réponse aux traitements. -> Contact : gaetane.nocturne@aphp.fr
|

|
Scooped by
Life Sciences UPSaclay
Today, 5:34 AM
|
SAVE THE DATE ! Inauguration of The HIVE & Kadans Oncology Summit – 4 June 2026
On Thursday, 4 June 2026, Kadans Science Partner will host a high-level event bringing together science, innovation and public leadership: The Kadans Oncology Summit, within The HIVE at the heart of the Campus Grand Parc in Villejuif. This day will mark the official launch of the State-designated Campus Grand Parc oncology cluster: a new centre of excellence designed to accelerate breakthroughs by uniting world-class research, industry leaders, institutions and public stakeholders. 🗓 Date: Thursday, 4 June 2026 🕔 Time: From 9:30 am 📍 Location: The HIVE - Campus Grand Parc, Villejuif What to expect: - High-level panel discussions and presentations led by leading oncology experts;
- Strategic discussions and networking sessions fostering collaboration between; research, industry and public authorities
- The official inauguration of The HIVE, a new flagship for oncology innovation.
Add to your calendar

|
Scooped by
Life Sciences UPSaclay
Today, 5:23 AM
|
La conférence de Carole Escartin "Les cellules gliales dans les maladies neurodégénératives : rôles émergents et potentiel thérapeutique" aura lieu le jeudi 2 avril 2026 à 16h à l'Auditorium Pierre Lehmann au bâtiment 200 du laboratoire IJCLab, sur le campus de l'Université Paris-Saclay. La conférence sera également retransmise via Zoom. Résumé Les neurones ne sont pas seuls dans le cerveau. A leur côté, plusieurs types de cellules, collectivement appelées « cellules gliales », remplissent de nombreux rôles essentiels pour le bon fonctionnement et la survie des neurones. Ces cellules leur apportent des substrats énergétiques, assurent la défense contre des pathogènes, éliminent certains déchets cellulaires, et même régulent la façon dont les neurones transmettent et traitent l’information nerveuse. Au final, les cellules gliales qui restent souvent dans l’ombre des neurones, sont des partenaires actifs permettant au cerveau de fonctionner, mais aussi d’apprendre et de s’adapter. Depuis plusieurs années, les chercheurs découvrent que ces cellules jouent aussi un rôle important dans de nombreuses maladies qui touchent le cerveau. En effet, ces cellules se transforment en contexte pathologique, elles sont dites « réactives ». Ces changements peuvent avoir des conséquences majeures - positives ou négatives - sur les neurones et leur survie. Les cellules gliales sont l’objet d’études variées pour comprendre leurs rôles dans le cerveau sain et pathologique, ouvrant des nouvelles pistes thérapeutiques pour de nombreuses maladies cérébrales. Carole Escartin est directrice de recherche au CNRS, responsable de l’équipe « Signalisation astrocytaire en physiologie et dans les maladies neurodégénératives » à l’Institut des neurosciences Paris-Saclay - NeuroPSI (CNRS/UPSaclay) à Saclay. -> Contact : carole.escartin@cnrs.fr

|
Scooped by
Life Sciences UPSaclay
Today, 5:05 AM
|
Financement européen – IHI Call 12
L’Innovative Health Initiative (IHI), partenariat public-privé européen dédié à l’innovation en santé, ouvre son appel à propositions (environ 163 M€) en réponse aux défis actuels et futurs de santé publique. Cet appel, en une seule étape, couvre cinq thématiques : - mieux comprendre les facteurs qui influencent la santé et les maladies ;
- favoriser la coordination des projets de recherche et d’innovation ;
- développer des solutions adaptées aux besoins des patients ;
- exploiter les outils numériques et les données ;
- mesurer l’efficacité et l’impact des solutions innovantes.

|
Scooped by
Life Sciences UPSaclay
Today, 4:53 AM
|
Le Consulat général de France à Québec soutient l’accueil en France de doctorants et post-doctorants québécois. Ces bourses (2 000 €) financent des séjours de recherche d’au moins un mois dans un laboratoire français.

|
Scooped by
Life Sciences UPSaclay
Today, 4:35 AM
|
SQY Therapeutics, société de biotechnologie spécialisée dans le développement de thérapies avec des oligonucléotides antisens tricyclo-DNA (tcDNA-ASO), annonce aujourd’hui les résultats d’une étude préclinique publiée le 22 janvier 2026 dans NAR Molecular Medicine. Cette étude, réalisée en collaboration avec une équipe de l’unité PHARMAColigo de l’UVSQ, démontre la faisabilité de l’administration par voie pulmonaire de l’oligonucléotide antisens SQY51, un tcDNA-ASO (en essai clinique par SQY Therapeutics pour la myopathie de Duchenne). Cette voie d’administration permet une distribution systémique efficace, une biodisponibilité prolongée et une excellente tolérance, ouvrant la voie à une alternative moins invasive que l’injection intraveineuse (IV) pour le traitement de maladies chroniques. Lire la suite du communiqué de presse -> Contact : christine.saulnier@sqy-therapeutics.com

|
Scooped by
Life Sciences UPSaclay
February 4, 11:34 AM
|
À l’occasion de la Journée mondiale de lutte contre le cancer, le professeur Éric Solary revient sur les avancées de la recherche et le rôle croissant de l’intelligence artificielle qui ouvre de nouvelles perspectives pour mieux détecter et traiter la maladie. Avec - Eric Solary, vice président de la Fondation ARC pour la recherche sur le cancer et Professeur émérite à la Faculté de médecine de l'Université Paris-Saclay
Aujourd'hui, plus de 3,8 millions de personnes vivent avec un diagnostic de cancer en France et les chiffres sont en augmentation partout dans le monde. En cette journée mondiale de lutte contre le cancer, on se penche sur la manière dont la technologie, plus spécifiquement les algorithmes basés sur l’IA, peuvent aider à améliorer la détection et le traitement de ces maladies. Pour parler de ces avancées de la recherche nous recevons Eric Solary, professeur émérite d'hématologie à la faculté de médecine de l'Université Paris-Saclay et vice-président de la fondation ARC dédiée à la recherche contre le cancer. Réécouter l’émission du 4 février 2026 -> Contact : eric.solary@gustaveroussy.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 9:47 AM
|
BRCA2 (BReast CAncer 2) protège l’intégrité du génome chez les eucaryotes en facilitant la réparation de l’ADN par trois mécanismes distincts : la réparation des cassures double-brin de l’ADN (DSB) par recombinaison homologue, la protection des fourches de réplication et la suppression des brèches d’ADN simple brin. Une caractéristique fondamentale des orthologues de BRCA2 est leur capacité à se lier à l’ADN. Dans un forum article publié dans Trends in Biochemical Sciences, des chercheurs de l’équipe MeioMe de l’Institut Jean-Pierre Bourgin - Sciences du Végétal - IJPB (INRAE/AgroParisTech/UPSaclay, Versailles) ont mis en lumière les rôles émergents de plusieurs modules de liaison à l’ADN, qui agissent de manière complémentaire et contribuent chacun de façon unique aux fonctions de BRCA2. -> Contact : rajeev.kumar@inrae.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 9:31 AM
|
Organe ouvert sur le milieu extérieur, les poumons se caractérisent par une grande surface d’échange alvéolocapillaire permettant la diffusion sanguine de nombreuses molécules inhalées. Ils représentent ainsi une voie d’entrée prometteuse pour l’administration systémique de médicaments, les nanoparticules pouvant traverser l’épithélium alvéolaire de faible épaisseur pour atteindre le réseau sanguin, tout en contournant le premier passage hépatique. Les progrès de la nanotechnologie ont mis en évidence le potentiel de l’appareil respiratoire comme voie efficace d’administration d’une grande variété de thérapeutiques, notamment de petites molécules, d’acides nucléiques, de protéines et de peptides comme l’insuline par exemple. Une étude préclinique publiée dans NAR Molecular Medicine a évalué l’instillation intratrachéale (IT) comme voie alternative pour l’administration systémique d’oligonucléotides antisens tricyclo-ADN (tcDNA-ASO) ciblant la dystrophie musculaire de Duchenne (DMD). L’administration IT a été comparée à l’injection intraveineuse (IV) chez la souris sauvage par l’analyse de la pharmacocinétique, la biodistribution, la localisation tissulaire et des effets immunologiques. Les résultats ont montré : i) que l’instillation IT permettait au tcDNA-ASO d’atteindre la circulation sanguine via la voie pulmonaire. Comparée à l’injection IV, l’administration IT entraînait des concentrations plasmatiques maximales plus faibles, une clairance plus lente et une détection prolongée ; ii) une accumulation plus importante dans le poumon mais des niveaux réduits dans le rein, le cœur et le diaphragme, indiquant un ciblage dépendant de la voie d’administration ; iii) une architecture pulmonaire préservée et l’absence d’inflammation significative après administration IT. En conclusion, une dose pulmonaire unique s’est révélée sûre et bien tolérée, sans effet délétère sur la structure pulmonaire ni effet immunitaire indésirable chez la souris. Ces données suggèrent l’utilisation de la voie pulmonaire comme une voie alternative non invasive envisageable pour l’administration systémique de tcDNA‑ASO chez l’homme. L’équipe de Thérèse Deramaudt et Marcel Bonay responsable de cette étude ont pour projet d’explorer le potentiel thérapeutique de nouvelles voies d’administration chronique des tcDNA‑ASO sur des modèles précliniques dans le cadre de la nouvelle Unité PHARMAColigo de l’UVSQ/UPSaclay (Montigny-le-Bretonneux) en collaboration avec le laboratoire SQY Therapeutics. Légende Figure : représentation schématique d’une souris traitée au tcDNA-ASO par instillation intratrachéale (IT) et par injection intraveineuse (IV). -> Contact : therese.deramaudt@uvsq.fr / marcel.bonay@aphp.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 9:09 AM
|
Dans une étude publiée dans Journal of Affective Disorders sous la coordination d’Amandine Luquiens (Centre de Recherche en Epidémiologie et Santé des Populations – CESP, INSERM/UVSQ/UPSaclay, Villejuif, et CHU de Nimes), les auteurs ont analysé les verbatims issus des séances d’intégration après administration de psilocybine chez des patients vivant avec un trouble de l’usage d’alcool récemment sevrés gardant des symptômes dépressifs. L’équipe a utilisé une approche mixte combinant analyses qualitative et quantitative pour identifier des dimensions expérientielles exprimées dans les récits des participants, utilisant une méthode de machine learning sur IRAMUTEQ®, permettant une analyse sans a priori. Les résultats ont révélé un verbatim riche et une analyse de bonne qualité (88.27% du texte classé) consistant en deux catégories principales : une décrivant les aspects sensoriels de l’expérience (44.2% des occurrences classées), essentiellement abordés par les non-répondeurs à la thérapie, et une autre reflétant un dialogue intérieur actif, caractérisé par une restructuration cognitive, une prise de distance vis-à-vis des anciennes habitudes de consommation, l’émergence de stratégies de coping basées sur l’acceptation et un engagement vers le changement (55.8%), portée par les personnes qui seront abstinentes 3 mois plus tard. Le dialogue intérieur en autonomie lors des sessions sous psychédéliques et rapporté lors de l’intégration semble un mécanisme thérapeutique en soi et pourrait être un indicateur de réponse. Les auteurs soulignent l’importance de ce résultat qui ouvre une piste afin de maximiser les bénéfices potentiels dans les troubles associés à l’alcool et la dépression via le travail de préparation et la psychothérapie associée aux psychédéliques. Légende Figure : nuage de mot des sessions d’intégration. -> Contact : amandine.luquiens@umontpellier.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 5:44 AM
|
Les thymomes sont des tumeurs rares du thymus, dérivées des cellules épithéliales thymiques. Ils se caractérisent par la présence d’une proportion élevée mais variable de lymphocytes, dont la fonction demeure mal définie. Une pathologie auto-immune est observée chez environ un tiers des patients dès le diagnostic. La thymopoïèse désigne le processus de différenciation et de maturation des lymphocytes T au sein du thymus. Dans le contexte du thymome, les scientifiques de l’Institut Curie Paris et Saint-Cloud (rattaché à l’UVSQ/UPSaclay) montrent dans un article publié dans Cancer Immunology Research que ce processus est altéré, avec des anomalies majeures affectant les mécanismes de sélection négative. Ces dysfonctionnements peuvent compromettre l’élimination des lymphocytes T autoréactifs, un mécanisme essentiel au maintien de la tolérance immunitaire centrale, et ainsi contribuer à la survenue de maladies auto-immunes chez ces patients. Par ailleurs, les auteurs mettent en évidence une diminution significative des lymphocytes T régulateurs. Cette réduction, associée à une sélection négative inefficace, favoriserait la persistance de lymphocytes T potentiellement pathogènes. Pour explorer ces mécanismes, les auteurs ont combiné plusieurs approches expérimentales complémentaires, incluant une analyse phénotypique par cytométrie de flux, une analyse transcriptomique par séquençage d’ARN en cellule unique, ainsi qu’une analyse spatiale par immunohistochimie multiplexe. Un résultat majeur, confirmé par plusieurs méthodes, est l’existence d’un blocage de la maturation des lymphocytes T à un stade intermédiaire de leur développement, le stade double positif (DP, CD4⁺CD8⁺). La trajectoire de maturation apparaît ainsi interrompue à ce stade chez les patients atteints de thymome. Ces résultats suggèrent que la production des lymphocytes T dans le thymome est anormale et pourrait expliquer la fréquence élevée des maladies auto-immunes associées à cette pathologie. Légende Figure : UMAP illustrant la trajectoire de vélocité de l’ARN dans des thymus adultes contrôles (panneau gauche, n = 4) et dans des thymomes (panneau droit, n = 8). -> Contact : clemence.basse@curie.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 4:46 AM
|
Dans une étude publiée dans Conservation Genetics, les scientifiques les scientifiques du laboratoire EGCE (CNRS/IRD/UPSaclay, Gif-sur-Yvette) (IDEEV) et de NeuroPSI (CNRS/UPSaclay, Gif-sur-Yvette) rapportent les résultats d’études de capture-recaptures pour évaluer les tailles des populations d’Astyanax mexicanus peuplant certaines grottes mexicaines. Les populations cavernicoles d'Astyanax mexicanus du nord-est du Mexique font l'objet d'études approfondies en tant que modèle pour l'étude des processus évolutifs dans les environnements souterrains. Alors que certaines questions clé sur les mécanismes de leur l'évolution ainsi que les questions de conservation nécessitent un cadre démographique bien défini, peu d'estimations de la taille des populations ont été obtenues. Ici, les chercheurs ont utilisé l'identification génétique d'individus cavernicoles capturés à différents intervalles de temps, allant de quelques jours à plusieurs années, et dans plusieurs grottes, afin de mieux comprendre la dynamique de la taille des populations, la structure des populations et leur connectivité. Ils ont utilisé 18 à 24 marqueurs microsatellites pour génotyper et identifier sans ambiguïté 702 poissons. La distance génétique entre les poissons échantillonnés, ainsi que les recaptures à court et à long terme, démontrent que la population d'Astyanax la plus étudiée, dans La Cueva de El Pachón, est petite (comprenant quelques centaines d'individus) et isolée. En revanche, les populations de La Cueva de Los Sabinos et d'El Sótano de Las Piedras, qui sont montrées comme un groupe de grottes bien connectées, pourraient être plus importantes, comptant plusieurs milliers d'individus. À une échelle géographique plus large, les données suggèrent que la distribution des populations d'A. mexicanus dans les eaux souterraines peut être décrite comme un réseau peu connecté d'oasis au sein d'un désert karstique. Légende Figure : Plan et résultats des CMR (Capture-Marque-Recapture). Le nombre de poissons capturés et génotypés chaque année est indiqué entre parenthèses. Le nombre de recaptures au cours d'une même année et d'une année à l'autre est indiqué sous les lignes reliant les échantillons. Deux poissons capturés deux fois au cours d'une même année lors d'un CMR à court terme, et re-capturés une autre année sont indiqués par un astérisque. Dans la grotte Pachón, des recaptures systématiques et très nombreuses sont observées, et les calculs indiquent une taille de populations de quelques centaines d’individus, un chiffre alarmant en termes de conservation. Tel n’est pas le cas pour la population de la grotte Sabinos, estimée à quelques milliers d’individus. -> Contact : sylvie.retaux@cnrs.fr / didier.casane@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
February 3, 4:23 AM
|
Disparition d'Amélie Bochot
C’est avec une profonde émotion et une très grande tristesse que nous avons appris le décès d’Amélie Bochot, samedi 31 janvier, à 56 ans. Amélie nous a quittés à la suite d’une maladie qu’elle combattait avec un courage exceptionnel depuis plus de dix ans. Docteur en Pharmacie, diplômée de l’Université de Bourgogne, Amélie est arrivée en octobre 1993 dans notre faculté à Chatenay-Malabry pour suivre le DEA de Pharmacotechnie et Biopharmacie. Après son stage dans l’UMR CNRS 8612, elle poursuit dans la même équipe par un doctorat, soutenu en 1998. Recrutée comme maître de conférences en Pharmacotechnie dans notre faculté puis promue professeure des universités en 2014, elle n’a jamais cessé, pendant toute sa carrière, y compris tout au long de sa maladie, de s’investir dans différentes tâches et missions au service de notre collectivité. Elle a ainsi régulièrement siégé à la commission de pédagogie ainsi qu’au conseil d’UFR. Elle a coordonné toute la partie pédagogie de l’opération Biologie-Pharmacie-Chimie en vue du déménagement de notre faculté sur le plateau de Saclay. Elle s’est impliquée très fortement dans les enseignements de Pharmacotechnie, discipline dont elle a assuré la responsabilité pendant plusieurs années. Elle a également pris en 2010, la responsabilité du parcours de master Technologie et management de la production pharmaceutique. Amélie était une excellente pédagogue, proche des étudiants pour lesquels elle se rendait toujours disponible et était, pour nombre d’entre nous, un modèle inspirant. Cet investissement en pédagogie avait été reconnu par l’obtention du grade de Chevalier dans l’ordre des Palmes académiques. En parallèle, elle a mené au sein de l’Institut Galien Paris-Saclay, une recherche originale et productive portant sur la conception de formes pharmaceutiques innovantes pour l’administration locale de substances actives. Elle a dirigé ou co-dirigé 13 thèses de doctorat d’université. Notre collègue représentait également notre faculté au sein d’instances extérieures, elle était membre du conseil de l’APGI (Association de Pharmacie Galénique Industrielle), du groupe Formulation de la SCF (Société Chimique de France) ainsi que membre titulaire au sein de l’Académie Nationale de Pharmacie. Amélie était une personne d’une droiture absolue, rigoureuse et qui mettait un haut niveau d’exigence dans son travail, associée à une bienveillance envers tous. Au-delà de ses qualités professionnelles, c’était une femme très attachante qui aimait la vie et qui incarnait l’élégance et le raffinement dans tout ce qu’elle entreprenait. Elle était appréciée et respectée de toute notre communauté et restera sans aucun doute dans les pensées de tous ceux d’entre nous qui l’ont côtoyée. Nous adressons nos plus sincères condoléances à son compagnon, sa famille, son équipe de recherche et à tous les amis qu’elle avait su se faire au sein de la Faculté. Claire Janoir, au nom de l’UFR Pharmacie.
|





 Your new post is loading...
Your new post is loading...