Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
December 5, 2021 11:00 AM
|
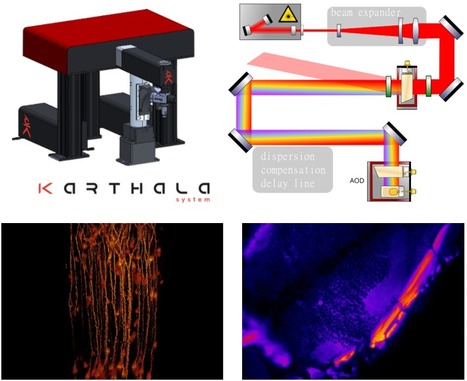
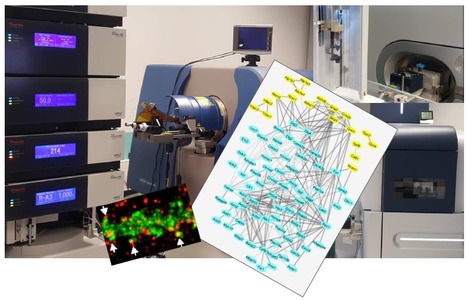
L'institut des Neurosciences Paris-Saclay (NeuroPSI) étudie le cerveau, de la molécule à la cognition. Une des forces de cet Institut est la large palette des espèces étudiées, depuis la larve de drosophile aux modèles poissons, oiseaux et rongeurs. Aujourd’hui, alors que l'étude du cerveau en action devient centrale en Neuroscience, la visualisation de l’activité de populations neurales au cours d’un comportement est désormais fondamentale pour ces recherches, en particulier parmi les équipes de NeuroPSI, et plus largement de la communauté des neuroscientifiques du territoire Paris-Saclay et d’Ile-de-France. Face à ce besoin, peu d’outils sont disponibles pour les chercheurs, notamment car leur financement est un vrai défi à l’échelle d’une équipe. Pour combler ce manque, les chercheurs Glenn Dallérac et Luc Estebanez, soutenu par le CNRS et l’Université Paris-Saclay, ont déposés le projet BeAM (Behavioral Analysis under Multiphoton imaging) dans le cadre de l’AAP SESAME IdF 2021. BeaM fait aujourd’hui partie des 4 projets UPSaclay sélectionnés par la région Île-de-France, et recevra à ce titre une subvention de 410 k€ (66% du montant de l’équipement visé), le complément étant assuré par le CNRS. BeaM repose sur l'achat d'un microscope biphoton de pointe, environné d'un dispositif expérimental de comportement multi-espèces ambitieux. Ce dispositif versatile permettra d'explorer le lien entre comportement et activité neuronale, dans un premier temps chez la drosophile et la souris, puis dans la diversité de tous les modèles animaux étudiés dans l'institut. L’équipement visé inclus un microscope du type de celui proposé par Karthala, société basée en Ile de France, et qui repose sur la technologie AOD, offrant un balayage ultra-rapide du cerveau, en lecture et écriture, dans un silence précieux pendant les expériences de comportement. Cet outil se différenciera aussi des autres microscopes multiphoton présents sur le plateau de Saclay, notamment celui de la plateforme d’imagerie neuronale profonde (CEA/Neurospin) et celui du laboratoire d’optique et biosciences (École Polytechnique), par l’environnement unique où il sera positionné, à l’interface des expertises de deux plateaux techniques, PSI-CO et NEURO-PICT. Ouverts à la communauté scientifique, le premier propose une offre complète d'hébergement couplée à l’étude du comportement des sujets expérimentaux et le second bénéficie d’une forte expertise forte en microscopie photonique. Contact : Glenn Dallérac (glenn.dallerac@cnrs.fr) et Luc Estebanez (Luc.Estebanez@cnrs.fr) En savoir plus sur la plateforme PSI-CO ? La plateforme de comportement PSI-CO dispose de plusieurs zones dédiées à l’étude du phénotype comportemental chez plusieurs modèles animaux aquatiques (Danio rerio, Oryzias latipes) et terrestres (souris, rats). Équipée de divers dispositifs associés à des caméras et logiciels de vidéotracking performants ainsi qu’un microscope biphoton de pointe, la plateforme PSI-CO permet d'analyser finement les comportements moteurs, spontanés (locomotion, exploration), sociaux, émotionnels, ainsi que de nombreuses fonctions cognitives (apprentissages, mémoires, prise de risque …). Des pièces de chirurgie, salles d’injection et salles de perfusions sont également réservables. En savoir plus sur la plateforme NEUROPICT ? La plateforme d’imagerie NEUROPICT a pour principal objectif de proposer à la communauté scientifique un ensemble de méthodologies indispensables dans le domaine des Neurosciences. Elle permet de faire de l’imagerie multi-échelle de la cellule à l’organisme entier, sur tissus ou organismes vivants, fixés ou transparisés. La plateforme propose son expertise ainsi que plusieurs équipements de microscopie photonique : des microscopes à épifluorescence ou à illumination structurée (Apotome), un ultramicroscope à feuille de lumière, deux microscopes confocaux à balayage laser, un microscope multiphoton. Nous disposons également d’une station d’analyse d’images Imaris indispensable pour traiter les jeux de données obtenus. La plateforme va également acquérir au cours de l’année 2022 un microscope bi-photon bi-directionnel haute résolution associé à des dispositifs comportementaux permettant d’établir les causalités entre dynamiques neurales et fonctions cérébrales intégrées sur l’animal vigile. Le personnel investi dans cette plateforme forme les nouveaux utilisateurs et leur propose aide et conseils. Nous pouvons également collaborer à des projets scientifiques et technologiques.

|
Scooped by
Life Sciences UPSaclay
November 14, 2021 1:04 PM
|
La plateforme de microscopie de fluorescence à super-résolution offre ses services à des équipes de recherche françaises et internationales et fait partie du pôle des plateformes de Biophysique de l’Institut de Biologie intégrative de la Cellule (I2BC, CEA-Joliot/CNRS/UPSaclay, Gif-sur-Yvette). La plate-forme est dédiée à l'imagerie en super-résolution d'échantillons biologiques et son laboratoire d’adossement se spécialise sur les propriétés physico-chimiques des cofacteurs pigmentés en biologie (caroténoïdes, chlorophylles, hèmes…) y compris des études in vivo des réactions biochimiques, photo-induites et régulatrices. L’originalité de la plateforme tient en son équipement, un prototype ou système original de microscopie de fluorescence développé au laboratoire et aujourd’hui associé à une jeune start-up : Linseg Tech ! Concrètement, comment se passe une étude ? Les échantillons sont tout d’abord placés entre un sandwich de lamelles de verre, qui est ensuite inséré dans le porte-échantillon d'un microscope à balayage laser « fait maison ». La fluorescence émise est ensuite capturée par une caméra EMCCD, s’ensuit l’analyse des images. Nos longueurs d'onde d'excitation standards sont 405 nm, 488 nm et 561 nm, cependant, d'autres longueurs d'onde sont disponibles (par exemple 375 nm, 413 nm et 637 nm) avec la possibilité d’enregistrer deux longueurs d'onde d'émission simultanée. Un exemple choisi d’application ? De nos jours, les cyanobactéries sont des systèmes modèles attrayants car elles peuvent utiliser l'énergie solaire, l'eau, le dioxyde de carbone et les minéraux pour produire des molécules pertinentes sur le plan biotechnologique. Cela nécessite souvent l'introduction et l'expression de gènes hétérologues, mais malheureusement, seules quelques souches modèles (par exemple Synechocystis PCC 6803 ou Synechococcus PCC 7942) ont un système génétique bien établi. L'équipe Biology and biotechnology of Cyanobacteria (Franck Chauvat, I2BC et CEA-Joliot) a récemment mis au point à partir des systèmes génétiques créés pour Synechocystis et Synechococcus, une souche Cyanothece PCC 7425 qui produit du limonène, un terpène à haute valeur ajoutée, qui a un large éventail d'applications (biocarburants, bioplastiques, cosmétiques, etc.). Cyanothece PCC 7425 a été choisi car il possède de nombreux attributs intéressants : grande taille de cellule, capacité à fixer l'azote atmosphérique et peut se développer sur l'urée (un polluant fréquent). Afin de démontrer une expression hétérologue, la protéine CcmK1 du carboxyzome a été marquée avec une protéine fluorescente (Green Fluorescent Protein, GFP) couramment utilisée. Dans l'image ci-dessus, est révélée la localisation de la membrane photosynthétique (autofluorescence de chlorophylles - en rouge) et le carboxysome (la CcmK1 protéine de carboxysome marquée avec GFP – en vert) dans Cyanothece PCC 7425, lorsqu'elle est excitée à 488 nm (notez la barre d'échelle : 500 nm). Notre méthode de microscopie se révèle non seulement très performante comparée aux systèmes actuellement présents sur le marché, mais aussi et surtout plus simple à utiliser et moins onéreuse, à la fois en terme de matériel et en terme d’investissement humain. En savoir plus ? Chenebault et al., Front. Microbiol. 2020. Contact : Andrew Gall (andrew.gall@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Plateforme de microscopie de fluorescence à super-résolution. La plate-forme est dédiée à l'imagerie super-résolution d'échantillons biologiques. Les échantillons sont placés entre un sandwich de lamelles de verre qui est ensuite inséré dans le porte-échantillon d'un microscope à balayage laser. La fluorescence émise est capturée par une caméra EMCCD. Nos longueurs d'onde d'excitation standard sont 405, 488 et 561 nm, cependant, d'autres longueurs d'onde sont disponibles. Cette plateforme fait partie du pôle des plateformes de Biophysiques de l'I2BC qui comprend les plateformes de RPE, FTIR, Résonance Raman, Spectroscopies Electroniques et Microscopie de fluorescence à super-résolution. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 70 équipes de recherches et 15 plateformes technologiques, provenant de 8 unités de recherches (CGM, IBBMC, IGM, ISV, LEBS, VMS, SB2SM, SBiGeM). L’institut est réparti sur 3 sites de recherche (Campus d’Orsay Vallée de l’Université Paris-Saclay, Campus du CNRS de Gif sur Yvette et Campus du CEA / Centre de Saclay) au sein de 14 bâtiments jusqu’au rassemblement programmé fin 2021 sur le campus du CNRS de Gif-sur-Yvette.

|
Scooped by
Life Sciences UPSaclay
October 31, 2021 1:18 PM
|
L’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) compte à ce jour 11 plateformes dont la plateforme de bioinformatique (IPSIT BioInfo). Cette dernière à une activité transversale dans l’UMS car elle intervient dans l’analyse des données produites par différentes plateformes. Dans ce contexte, nous développons des pipelines d’analyse spécifiques aux différentes technologies. Pour ce focus, nous vous présentons des résultats de tests réalisés sur notre pipeline d’analyse de données omiques à partir d’un article auquel nous avions collaboré [Hervé B et al., Clin Genet. 2016]. Ce type de données est générée par des biotechnologies à haut-débit de type spectrométrie de masse, séquençage ou encore puce à ADN. Reproduire la séquence d’analyse des données d’un travail publié est un exercice courant en bioinformatique. Cela permet autant de rechercher de nouvelles variables d’intérêts qui n’ont pas fait l’objet principal de l’article que de tester ses propres procédures d’analyse afin de voir si on est capable de reproduire les résultats obtenus. En outre, que cela soit pour répondre à des critères qualités ou aux besoins de nos collaborateurs il est indispensable de pouvoir reproduire des analyses réalisées auparavant. Classiquement, il est indispensable d’effectuer un prétraitement des données pour obtenir une matrice. Cette procédure est propre à chaque technologie mais peut être résumée par les étapes suivantes : extraction des données à partir des fichiers bruts, contrôles qualités, filtrage et normalisation. On s’assure ainsi que les données sont formatées correctement pour pouvoir y appliquer des modèles statistiques. Cette matrice est aussi le point d’entrée du pipeline d’analyse. Ce dernier est utilisable sous forme d’une simple fonction intégrée à un package R. L’un des objectifs présenté dans l’article était d’établir un lien de corrélation entre la présence d’un chromosome surnuméraire, ce qui est le cas chez des embryons atteints de trisomie, avec une augmentation de l’expression des gènes portés par ce dernier. Des puces d’expression ont donc été utilisées afin de mesurer l’expression de l’ensemble des gènes sur des embryons atteints de trisomie 13, 18, 21 et sur des échantillons contrôles. Les prélèvements ont été effectués sur deux types tissulaires, le chorion pour les échantillons de trisomie 21 et du liquide amniotique pour les trisomies 13 et 18. Des échantillons contrôles non-porteurs du syndrome ont été prélevés également pour les deux conditions. Les résultats que nous avons cherché à reproduire sont les valeurs d’enrichissement obtenues qui ont permis de mettre en évidence le lien entre surexpression des gènes et présence du chromosome surnuméraire. Le but du travail a donc été de reproduire l’analyse pour tester deux fonctionnalités de notre pipeline : i) L’enrichissement (appelé aussi analyse fonctionnelle) qui permet de rendre compte si une liste de gènes différentiellement exprimés contient un grand nombre de gènes liés à un processus biologique connu (ici, on cherche à savoir si les listes de gènes obtenus contiennent plus de gènes liés aux chromosomes 13, 18 et 21 ; et ii) la prise en compte d’un facteur expérimental (ici les deux types tissulaires), dans le modèle d’analyse de variance. Cette fonctionnalité implémentée à partir du package limma [Ritchie E et al., Nucleic Acids Res. 2015] retire la variance considérée comme un biais expérimental de la matrice de données. Cette transformation, appelé RBE pour remove batch effect, permet une meilleure estimation des moyennes et des variances du facteur d’intérêt. Le calcul des p-values et des fold-changes est ainsi amélioré permettant de diminuer les faux positifs et les faux négatifs dans la liste de gènes sélectionnés (p < 0.05 et f > 1.5). Cette transformation ouvre également l’analyse à des méthodes basées sur de la corrélation comme le clustering hiérarchique et permet également d’améliorer la représentation graphique des données. Comme montré sur la figure ci-dessus, on voit que le pipeline a permis de reproduire les résultats d’enrichissement publiés. On note également que ces valeurs sont améliorées avec l’utilisation de la fonction RBE qui permet donc de prendre en compte un biais expérimental dans les calculs. Ce travail nous permet de nous assurer que notre pipeline fonctionne et qu’il peut être utilisé sur des jeux données dont les différences entre groupes de traitement ou phénotypes portent sur des signaux plus faibles et donc plus difficiles à valider biologiquement. Merci à nos étudiants stagiaires, Louise Weber en licence de bioinformatique à l’Université d’Evry et Paul Savescu étudiant à la faculté de pharmacie de Paris-Saclay ainsi qu’à nos collègues de l’IPSIT pour leurs contributions à ce travail. Contact : Florent Dumont (florent.dumont@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IPSIT BioInfo. La plateforme de bioinformatique de l’UMS IPSIT propose des prestations de services en bioinformatique et biostatistiques. Nous mettons l’accent sur le suivi des projets, la formation et l’autonomisation de nos utilisateurs. La création d’outils sous forme de packages R accessibles et portables permet de faciliter le transfert de compétence.

|
Scooped by
Life Sciences UPSaclay
October 17, 2021 1:07 PM
|
En plus de leur rôle dans le stockage et le transfert de l'information génétique, les acides nucléiques (ADN et ARN) sont impliqués dans de nombreuses voies de régulation des processus cellulaires. En effet, ils peuvent former une myriade de structures tridimensionnelles parmi lesquelles certaines peuvent favoriser une activité catalytique ou une interaction avec des protéines ou d'autres partenaires. Des techniques d’évolution moléculaire in vitro ont été développées pour exploiter ces propriétés et identifier des ligands de hautes affinités appelés aptamères. MIRCen/Aptamère est une plateforme rattachée au département MIRCen de l'institut de biologie François Jacob du CEA et localisée sur le site CEA Paris-Saclay de Fontenay-aux-Roses. Elle fournit des méthodologies, équipements et expertises pour la sélection, la caractérisation et l'ingénierie d'aptamères pour des équipes de recherche académiques et des partenaires industriels qui souhaitent utiliser ces macromolécules pour diverses applications : sondes d’imagerie, procédés de bio-purification, kits de diagnostic, ou médicaments. Afin d’améliorer l’identification des aptamères, MIRCen/Aptamère a développé une méthode (PATTERNITY.seq) pour étudier l’évolution moléculaire in vitro à l'aide du séquençage à haut débit. En analysant plusieurs millions de séquences à partir de chaque cycle d’évolution, cette méthode permet d’identifier de meilleurs aptamères en utilisant moins de cycles de sélection. Cette méthode est bien connue par la communauté « aptamère » et la plateforme est actuellement sollicitée tous les mois pour des prestations par des laboratoires du monde entier (USA, Brésil, Allemagne...). Grâce à un soutien de l’ex-département des Sciences de la Vie de l’Université Paris-Saclay (lauréat à l’AAP Petits Équipements de Laboratoire | PEL-SDV-UPSAY-2019), la plateforme s’est d’ailleurs équipée d’un petit séquenceur à haut-débit (iSeq 100 system, ILLUMINA). Jusqu’à présent la méthode PATTERNITY.seq n’avait analysé que des évolutions de structures d’acides nucléiques. Mais, MIRCen/Aptamère a récemment démontré que la méthode pouvait être étendue à l’étude de l’évolution moléculaire de protéines. Pour cela, l’équipe CREAB du laboratoire SyMMES du CEA Grenoble a confié à la plateforme des banques d’ADN issues d’un processus d’évolution de peptides connu sous le nom de « Phage display ». La plateforme a réalisé le séquençage et l’analyse de ces banques et a pu mettre en évidence un enrichissement de certains motifs de peptides qui aurait été difficile à analyser par la méthode de clonage et séquençage classique. La plateforme a également étendu l’utilisation de PATTERNITY.seq à du criblage de banques de composés chimiques marqués par des codes-barres ADN en collaboration avec une équipe du Département de Chimie Moléculaire (DCM), UMR CNRS 5250 de l’Université Grenoble Alpes. L’intérêt du séquençage à haut-débit pour le criblage de composés est donc en pleine expansion et l’expertise de MIRCen/Aptamère dans le traitement et l’analyse de ces données pourra être bénéfique à de nombreux laboratoires. N’hésitez pas à nous contacter ! Plug In Labs Université Paris-Saclay : cliquer ICI Contact : Frédéric Ducongé (frederic.duconge@cea.fr) MIRCen / Aptamère propose des méthodologies, équipements et expertises pour la sélection, la caractérisation et l'ingénierie d'aptamères ADN, ARN ou acides nucléiques résistant aux nucléases. Ces aptamères peuvent servir dans différentes applications comme le développement d'agent de contraste, de procédés de bio-purification, de kit de diagnostic, ou de nouvelles thérapies.La plateforme est ouverte aux prestations ou collaborations avec les industriels ou laboratoires académiques et permet d'identifier des aptamères contre tout type de cible (petites molécules, peptides, protéines, cellules...). En s'assurant de la protection des travaux (brevets, accord de confidentialité, MTA...), nous travaillons en étroite relation avec nos partenaires afin d'être au plus proche de leurs besoins. Un soin particulier est porté sur les protocoles de sélection afin de garantir que les aptamères répondent au mieux à leur utilisation finale. Notre plateforme peut également être utilisée pour caractériser des aptamères en terme d'affinité, de spécificité et d'activité biologique. Récemment la plateforme a développé une méthode (PATTERNITY.seq©) pour améliorer la sélection d'aptamères à l'aide du séquençage à haut débit. Cette méthode permet d'identifier plus rapidement les aptamères et a reçu des prix aux deux derniers congrès internationaux sur les aptamères.

|
Scooped by
Life Sciences UPSaclay
October 3, 2021 11:10 AM
|
L’unité expérimentale INRAE/Sciences de l’Animal et de l’Aliment de Jouy (SAAJ, UE 1298) a pour vocation principale de fournir et héberger des animaux (lapins, ovins, caprins) utilisés à des fins scientifiques dans le cadre d’études physiopathologiques variées, de participer au développement de modèles expérimentaux sur ces animaux, de fournir des prestations expérimentales ainsi que tout type de matériel biologique issus de ces animaux et nécessaire aux différentes recherches menées en biologie animale et humaine. Exemples choisis : Dans le cadre de collaborations étroites avec le plateau de Chirurgie et Imagerie Médicale chez l'Animal (CIMA, plateforme MIMA2) et différentes unités de recherche de notre Centre INRAE de Jouy-en-Josas, nous réalisons actuellement des prestations pour un projet de greffe utérine mené sur des brebis (Hôpital FOCH – Unité BREED INRAE/UVSQ/UPSaclay), pour une étude par édition de génome des gènes liés à l’apparition de mammites et à la production laitière chez les ovins, ou encore pour une étude de gènes liés à la résistance au prion chez des chèvres (Unité BREED). De leur côté, nos lapins participent à de nombreuses études, en particulier des études centrées sur le développement embryonnaire précoce et le développement fœtal, réalisées sur des animaux conventionnels comme sur des lignées de lapins OGM générées et entretenues dans l’unité SAAJ en collaboration avec nos collègues de l’unité BREED. Nos lapins participent aussi à des études visant à caractériser les effets d’expositions maternelles à la pollution par des nanoparticules (diesel, or, etc.) et à leurs effets trans-générationnels (Unité BREED INRAE), mais aussi à des études explorant le potentiel thérapeutique de certaines bactéries du microbiote intestinal sur l’hypercholestérolémie (Institut MICALIS) et à la mise au point de nouvelles stratégies vaccinales. En savoir plus ? Carbonnel et al., PLoS One 2021 (modèles ovins de transplantation utérine), Calderari S et al., Biol Reprod 2021 (collecte de fluides intra-utérins chez le lapin), Rousseau-Ralliard D et al., Sci Rep. 2019 (lapins et exposition in utero au diesel). Pour toutes ces études, nous disposons des installations et des compétences pour préparer les animaux, puis les suivre tout au long du protocole expérimental prévu jusqu’à la fin de ce dernier, y compris la fourniture de tous les prélèvements et des tissus requis. Besoin d’une expertise sur ces sujets, ou d’une collaboration ? N’hésitez pas à nous contacter ! Contact : Patrice Congar (patrice.congar@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI INRAE/UNITÉ EXPÉRIMENTALE SCIENCES DE L'ANIMAL ET DE L'ALIMENT DE JOUY (UE SAAJ). Les missions confiées à SAAJ sont doubles : i) fournir des animaux de qualité, correspondant à la demande scientifique dans le cadre notamment de recherches centrées sur le développement embryonnaire et foetal, le comportement animal et la mise au point de stratégies vaccinales ; ii) répondre aux besoins en régimes expérimentaux des laboratoires de recherche de l'INRAE en réalisant, à la demande des régimes alimentaires particuliers. Dans le cadre de son activité "expérimentation animale", l'UE SAAJ produit des modèles expérimentaux à des stades physiologiques donnés ou pour des études de pathologies humaines, fournit du matériel biologique tel que ovocytes, ovaires, embryons, foetus, cellules et tissus issus d'animaux vivants ou après euthanasie. Elle entretient des animaux "précieux" comme des individus transgéniques, immunisés ou opérés. Elle assure des prestations techniques sur animaux dont des méthodes d'échographie uniques. Trois espèces sont disponibles : lapin, ovin, caprin. Dans le cadre de son activité de fabrication de "Régimes à façon", l'UE SAAJ prépare des régimes expérimentaux particuliers et en petites quantités pour des laboratoires de recherche de l'INRAE en réalisant, à la demande et de façon fiable (incorporation de micro-quantités), des régimes alimentaires (études de carence ou de toxicité par exemple).

|
Scooped by
Life Sciences UPSaclay
September 19, 2021 10:45 AM
|
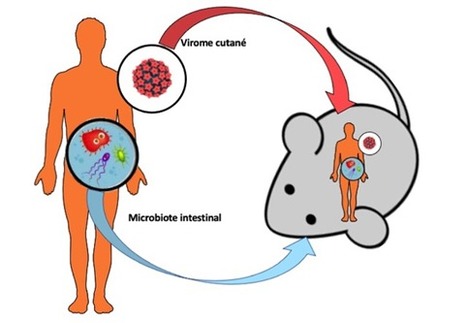
La Plateforme AnimEx (Animalerie et Exploration fonctionnelle du petit animal) de l’UMS IPSIT (Faculté de pharmacie, Université Paris-Saclay) offre à la communauté scientifique des structures d’hébergement et d’élevage d’animaux et des équipements pour l’exploration fonctionnelle du petit animal. La plateforme développe ses activités sur deux sites géographiques : la Faculté de Pharmacie de Châtenay-Malabry (AnimEx1) et le centre INSERM de l’Hôpital Antoine-Béclère à Clamart (AnimEx2), objet de ce FOCUS PLATEFORME ! Quelle spécificité pour AnimEx2 ? AnimEx2 a développé une forte expertise des modèles murins visant à analyser l’impact du microbiome humain dans le développement de différentes pathologies. Le microbiome regroupe l’ensemble des microorganismes, bactériens, fongiques et viraux que l’on retrouve au niveau des muqueuses et épithéliums. Il existe pour une majorité de ces microorganismes, une spécificité d’espèces qui représente un frein pour l’étude des microbiomes humains chez l’animal et c’est dans ce cadre que les équipes utilisatrices d’AnimEx 2 ont développé des stratégies d’humanisation de souris immunocompétentes. Deux exemples concrets associés à l’UMR-S 996 INSERM – UPSaclay (Inflammation, Microbiome and Immunosurveillance) ? Exemple 1 - Equipe II (Immunoregulation, chemokines and viral persistence) : Cette équipe, spécialiste des interactions entre le papillomavirus humain (HPV) et son hôte humain, développe des stratégies de greffes d’équivalents de peau humaine générée in vitro pour implanter le virome cutané humain, et en explorer les effets sur l’homéostasie tissulaire. Par ailleurs, l’équipe étudie les aspects pathogènes du virome cutané dans des modèles transgéniques d’oncogenèse causée par HPV ayant permis de mettre en évidence des facteurs de susceptibilité de l’hôte humain (Freitas et al, 2016 ; Gallego et al, 2021). Exemple 2 - Equipe III (Microbiome in liver disease: from susceptibility to treatment) : Pour l’étude du microbiote intestinal dans le cadre des hépatopathies nutritionnelles, cette équipe transplante des microbiotes intestinaux de patients à des souris. Ces modèles ont déjà permis de mettre en évidence le rôle causal du microbiote intestinal dans la maladie alcoolique du foie (Llopis et al, 2016; Wrzosek et al, 2020) et visent actuellement à apporter des perspectives thérapeutiques qui seront testées lors d’études preuves de concept. Contact : Valérie Domergue (valerie.domergue@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Lire à nouveau le précédent FOCUS PLATEFORME d’AnimEX (octobre 2019) ? Cliquer ICI La Plateforme AnimEx (Animalerie et Exploration fonctionnelle du petit animal) est située sur deux sites de l’Université Paris-Sud, la Faculté de Pharmacie de Châtenay-Malabry et le Centre INSERM de l’Hôpital Antoine-Béclère à Clamart. Sa mission principale est d’offrir à la communauté scientifique du secteur public ou privé, des structures d’hébergement et d’élevage d’animaux et des équipements pour l’exploration fonctionnelle du petit animal. AnimEx, est membre de CAPSud (Consortium des Animalerie Paris-Sud), est rattaché au Comité d’éthique CEEA n°26 et utilise le logiciel Tick@Lab pour la gestion de ses animaux. AnimEx1 (site de Châtenay) : Capacité d'hébergement : 3 500 cages (souris, rats, hamsters, cobayes, lapins). Superficie : 1 200 m². AnimEx2 (site de Clamart) : Capacité d'hébergement : 840 cages (3 500 souris). Superficie : 200 m².

|
Scooped by
Life Sciences UPSaclay
September 5, 2021 12:03 PM
|
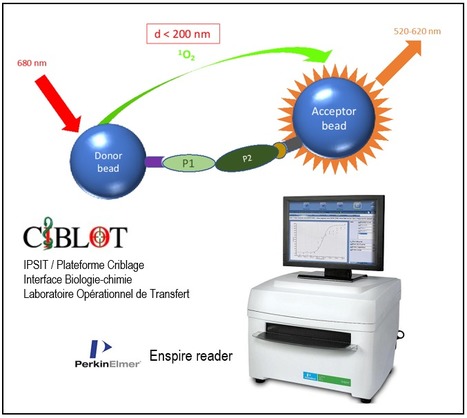
La plateforme CIBLOT (Criblage Interface Biologie-chimie Laboratoire Opérationnel de Transfert) fait partie de l’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique, Université Paris-Saclay/INSERM/CNRS) et offre ses services aux équipes académiques et industrielles. Dans la chaîne du médicament, CIBLOT intervient dans la découverte des « touches » ou hits qui sont des molécules actives qui peuvent être représentées comme des clés pouvant se loger dans des serrures, c'est-à-dire des protéines cibles. Pour cela, CIBLOT accueille et forme des biologistes qui conçoivent, adaptent et miniaturisent des tests biologiques permettant l’identification et la confirmation de ces molécules bioactives. Ensuite les chimistes vont modifier ces molécules en ajoutant ou retirant des fonctions chimiques pour obtenir une clé idéale pouvant s’insérer uniquement dans une protéine visée (cible) ce qui correspond à l’étape d’optimisation dite « hit-to-lead ». Cette plateforme se situant donc à l’interface entre la biologie et la chimie, implique une étroite collaboration entre les biologistes, les chimistes et de ce fait, favorise le transfert de compétences entre les scientifiques des différents domaines. Des développements technologiques ? CIBLOT a notamment contribué au développement d’un test d’interaction protéine-protéine par la technologie dite Alphascreen (Perkin Elmer). Cette technologie est basée sur l’utilisation de deux billes, l’une « donneuse » et l’autre « acceptrice » sur lesquelles sont greffées les deux protéines d’intérêt (P1 et P2, voir figure). L’interaction entre les 2 protéines génèrent le rapprochement physique des 2 billes. L’excitation par un laser à 680 nm de la bille « donneuse » conduit à la libération de singulets d’oxygène qui diffusent vers la bille « acceptrice » générant une réaction chimique provoquant la libération de lumière mesurable à l’aide d’un lecteur Enspire. Les composés inhibiteurs vont rompre l’interaction entre les 2 protéines et provoquer une baisse de lumière émise. Les tests sont réalisés en plaques 384 puits et adaptées au criblage à haut débit. Un exemple choisi d’application ? Récemment, par criblage de la banque Prestwick (1200 molécules) et d’une banque de petites molécules à façon fournie par l’infrastructure nationale ChemBioFrance (3300 molécules), des composés inhibiteurs de deux protéines mitochondriales ont pu être identifiés dans le but de développer de nouveaux inhibiteurs métaboliques à visée anti-cancéreuse. Ce projet porté par Catherine Brenner (CNRS UMR 9018, Gustave Roussy, Université Paris Saclay) a permis le dépôt d’un brevet par l’Université Paris-Saclay, l’INSERM et Gustave Roussy (Brenner & Modjtahedi, 2020) et se poursuit par la recherche des meilleurs candidats et l’étude de la spécificité tumorale des composés dans les cancers pédiatriques (Dr B. Geoerger, Gustave Roussy, Villejuif). Ces études sont financées par l’INCa, Enfants et Santé et la Société Française de Lutte contre les Cancers et Leucémies de l'Enfant et de l'Adolescent (SFCE). L'objectif principal de CIBLOT est d'aider les chercheurs à identifier des petites molécules qui puissent être utilisées comme sondes pour moduler des fonctions biologiques, et pour fournir des têtes de séries pour découvrir des médicaments dirigés contre diverses maladies. CIBLOT constitue une interface privilégiée de collaboration entre biologistes et chimistes, et un cadre de transfert de compétences scientifiques et technologiques entre eux. CIBLOT est aujourd'hui partie intégrante de C@PS, une plateforme réunissant sous une bannière unique les activités de criblage sur le plateau de Saclay, labélisée IBISA fin 2018). C@PS agrège les compétences i) de la plateforme CIBI (ICSN, CNRS, Gif-sur-Yvette) pour les mesures de cytotoxicité, de criblage d’interactions protéines/ligands et criblage in ovo, ii) de la plateforme CTPF (ICSN, CNRS, Gif-sur-Yvette) pour les mesures d’interactions protéines/ligands par thermal shift assay et d’analyses de transcriptomes par PCR quantitative, iii) de la plateforme CCCHD (Joliot, CEA, Saclay) pour la réalisation de criblages biologiques à haut débit ainsi que la préparation de chimiothèques ciblées et iv) de la plateforme CIBLOT (Faculté de Pharmacie, Université Paris-Saclay, Châtenay-Malabry) pour les mesures d’interactions protéines/ligands par technologie alpha-screen et la quantification de paramètres cellulaires. Contacts : CIBLOT : Delphine Courilleau (delphine.courilleau@universite-paris-saclay.fr); Catherine Brenner (catherine.brenner@universite-paris-saclay.fr) ; C@PS : Jean-Christophe Cintrat (jean-christophe.cintrat@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
July 25, 2021 12:20 PM
|
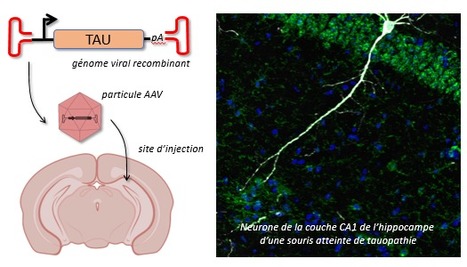
MIRCen (Molecular Imaging Center, Institut de Biologie François Jacob, CEA / Fontenay-aux-Roses) rassemble sur près de 8500 m2 des expertises et méthodologies complémentaires dédiées à l’exploration et la validation de concepts, ainsi qu’à l’évaluation préclinique de nouvelles stratégies thérapeutiques géniques, cellulaires ou médicamenteuses, principalement dans le domaine des maladies neurodégénératives. Exemple choisi d’application des technologies développées par l’une de ses plateformes, MIRCEN / Biologie moléculaire et production virale, experte dans la production de virus adéno-associés (AAV) et lentiviraux Le transfert de gènes est aujourd’hui devenu un outil incontournable en biologie, et ces dernières années ont vu l'explosion de l'utilisation de vecteurs viraux (AAV, lentivirus) pour modifier génétiquement certains organes ou certaines cellules. On parle alors de transgénèse somatique, manipulation permettant de sur-exprimer un gène d’intérêt, de réprimer un gène endogène voir plus récemment, de modifier le génome avec les nouveaux outils d’édition génomique (Crisp-Cas9). Ces outils sont utilisés non seulement pour la mise au point de thérapies dites géniques, mais également pour répondre à des questions biologiques in vivo, et dans le cas du présent FOCUS PLATEFORME, appliqués dans le cadre d’études physiopathologiques des maladies neurodégénératives (Alzheimer en particulier), et plus particulièrement les tauopathies. La protéine Tau est exprimée spécifiquement dans les neurones. Elle est codée par le gène MAPT, et sa fonction principale est de stabiliser le cytosquelette axonal de microtubules. Son affinité de liaison aux microtubules est finement régulée par son état de phosphorylation qui peut avoir lieu sur de nombreux sites physiologiques et pathologiques. Dans le cas des tauopathies, la protéine est hyperphosphorylée ce qui diminue son affinité pour les microtubules et augmente sa concentration sous forme libre dans le cytoplasme, entrainant alors son agrégation progressive d’abord sous formes d’oligomères puis en agrégats matures conduisant à la dégénérescence des neurones. Afin de déterminer la toxicité des différentes formes de Tau, MIRCEN / Biologie moléculaire et production virale a mis au point des vecteurs AAV capables de sur-exprimer la protéine Tau hyperphosphorylée soit sous forme d’oligomères solubles, soit sous forme fortement agrégées. L’injection de ces vecteurs dans l’hippocampe, un noyau cérébral responsable des processus mnésiques qui est fortement atteint lors de la maladie d’Alzheimer, a permis de montrer que, contrairement à ce que l’on imaginait, ce sont les formes oligomériques solubles qui sont les plus toxiques pour les neurones (d’Orange et al., Brain 2018). Incidemment, lors de cette étude, il est apparu qu’après injection des AAV dans l’hippocampe de rat, des astrocytes étaient également atteints de tauopathie. MIRCEN / Biologie moléculaire et production virale a alors élaboré et produit une nouvelle série de vecteurs permettant d’exprimer et d’agréger la protéine Tau spécifiquement dans les neurones ou les astrocytes. Ces nouveaux outils ont permis de montrer que les neurones sont capables d’échanger des formes pathologiques de la protéine Tau avec les cellules gliales, ce qui de plus s’est avéré particulièrement toxique pour certaines populations d’astrocytes de l’hippocampe. Ces résultats, qui ont un impact important sur notre compréhension des mécanismes physiopathologiques des tauopathies, viennent d’être publiés (Maté de Gérando et al., Brain 2021). MIRCEN / Biologie moléculaire et production virale propose son expertise pour l'élaboration de vecteurs viraux dérivés d'AAV (adeno-associated virus) et de lentivirus, leur production et leur caractérisation. Ces vecteurs viraux permettent le transfert de gène de façon stable ou transitoire in vitro dans des lignées cellulaires ou des cultures primaires et in vivo dans un organisme entier (rongeur, primate non humain, etc.). Ils peuvent être utilisés pour surexprimer un gène d'intérêt, inhiber un gène endogène (shRNA), ou exprimer un gène rapporteur. La plateforme est composée de 2 plateaux techniques : i) un laboratoire L1 de biologie moléculaire et microbiologie cellulaire dédié au clonage de gènes d'intérêt dans des plasmides d'expression eucaryotes mais aussi à l'étude de l'expression des gènes dans des modèles animaux; ii) un laboratoire L3 composé de 10 pièces dédiées à la production et à la manipulation des vecteurs viraux. Les vecteurs viraux y sont titrés et testés pour leur efficacité. L'évaluation de la biodistribution et de l'effet biologique de ces vecteurs est ensuite prise en charge principalement par la plateforme d'histologie mais également par la plateforme d'imagerie in vivo (et notamment imagerie TEP) et la plateforme d'analyse comportementale. Contact : Alexis Bemelmans (alexis.bemelmans@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI A propos de MIRCen. MIRCen - Molecular Imaging Research Center - est une installation de recherche préclinique développée par le CEA et l'INSERM. Cette installation, basée sur le site CEA de Fontenay-aux-Roses, est constituée d'un ensemble de plateformes dédiées au développement et à la validation de modèles animaux pertinents de pathologies humaines. Ces modèles sont utilisés pour le développement et la validation de nouvelles techniques permettant de détecter des déficits à un stade précoce de la maladie. Ils sont également utilisés pour évaluer des thérapies innovantes dans le domaine des maladies neurodégénératives. Ce centre fait partie intégrante de l’Infrastructure Nationale en Biologie et Santé (INBS) Neuratris, infrastructure dédiée aux thérapies innovantes en neurosciences.

|
Scooped by
Life Sciences UPSaclay
July 4, 2021 11:12 AM
|
Focus sur une expertise relativement rare sur le territoire Paris-Saclay : La purification de cellules ou d’organites par le tri par cytométrie en flux ! La cytométrie en flux permet de mesurer, évènement par évènement, des caractéristiques morphologiques (taille, complexité/granularité) et de fluorescence sur une population de cellules ou d’organites en suspension. Le tri par cytométrie en flux rend également possible la séparation physique et donc la purification de ces évènements suivant un ou plusieurs critères définis (morphologie, fluorescence) et à partir de populations hétérogènes. Mais concrètement, étape par étape, comment cela fonctionne-t-il ? Explication illustrée avec l’utilisation du cytomètre « Analyseur-Trieur » MoFlo ASTRIOS® (Beckman-Coulter) disponible sur la plateforme de cytométrie de l’I2BC (Institut de Biologie Intégrative de la Cellule, Imagerie-Gif, campus CNRS de Gif-sur-Yvette). Les cellules (ou autres particules en suspension) préparées dans leur milieu sont introduites dans le trieur (encadré 1) et soumises à une pression, qui permet de les faire circuler dans le système fluidique. Parallèlement, un liquide de gaine salin est également soumis à une pression, inférieure à celle de l’échantillon. La « rencontre » entre l’échantillon et le liquide de gaine soumise à une pression différentielle induit la formation d’un entonnoir virtuel (encadré 2) permettant aux évènements de passer devant les lasers un à un. Chaque évènement est alors identifié selon ses critères morphologiques et/ou fluorescents. Le système fluidique est soumis à une vibration de fréquence et d’amplitude connues, qui va induire la formation de gouttelettes (encadré 3). Les gouttelettes composées de liquide salin (liquide de gaine) et enfermant un évènement (exemple une cellule) vont pouvoir être chargées électriquement, et déviées par des plaques en métal également chargées. La charge apposée à la gouttelette va dépendre de l’identité de l’évènement qu’elle enferme, afin d’être déviée dans la voie de tri choisie. Cette technologie permet une séparation jusqu’à 6 voies simultanément (encadré 4). Il est possible de trier dans des tubes type Eppendorf, des tubes à hémolyse, des tubes de 15 mL ou 50 mL ou encore directement en plaque de 6 à 96 puits, avec le nombre choisi de cellule(s) par puits, ce qui en fait une technique très adaptée au clonage. Le tri peut se faire sur des cellules fixées ou des cellules vivantes, avec une vitesse allant jusqu’à 15 000 évènements par seconde pour des cellules eucaryotes voire 35 000 évènements par seconde pour des micro-organismes. Les cellules triées sont viables, rendant possible la remise en culture. Le tri peut également être couplé à toutes les techniques -omiques en aval, ou à de l’imagerie en dynamique cellulaire. Cette technique ouvre aussi de nombreuses possibilités en séquençage dit sur cellule unique. Vous pouvez également trouver cette technologie sur la plateforme d’imagerie et de cytométrie PFIC (UMS AMMICA, Gustave Roussy, Villejuif), OCCIGEN (Genopole, Evry), et depuis quelques semaines seulement, sur la plateforme PLAIMMO (UMS IPSIT, Châtenay-Malabry) également. Contact : Mickael Bourge (mickael.bourge@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, le 20 septembre 2020, la plateforme de cytométrie publiait son premier un Scoop-it® / FOCUS PLATEFORME : La puissance statistique de la cytométrie au service de l’étude de flux ioniques intracellulaires en temps réel. Le lire à nouveau ? I2BC / Plateforme de cytométrie (I2BC - plateformes IMAGERIE-GIF). Plus de 25 ans de service ! la plateforme réalise environ 300 prestations par an pour divers groupes de recherche appartenant à différents organismes de tutelles ou de sociétés privées. Les publications du service traduisent les nombreuses collaborations développées avec les différents instituts de la communauté Paris-Saclay ainsi qu’avec d’autres partenaires tels que l’INRA, l’INSERM, l’IRD, le CIRAD, des universités françaises et étrangères. Son expérience polyvalente et son expertise en sondes fluorescentes permettent d’adapter la cytométrie à des projets très divers issus de laboratoires publics et privés. Son partenariat avec SPS (Labex Saclay Plant Sciences) contribue à une activité importante dans le domaine de la biologie végétale. La plateforme de cytométrie en flux propose la mesure de fluorescence d'un ou plusieurs (> 10) fluorochromes simultanément, cellule par cellule. Cette technologie permet d'étudier: i) le dosage de la quantité d’ADN nucléaire en vue de l’étude de cycles cellulaires et d’endoréplication, ii) le dosage d’ADN à des fins de recherche en écologie et systématique, et d’amélioration des variétés (analyse de ploïdies), iii) le suivi de l’activité génique par l’expression d’un ou plusieurs gènes rapporteurs (tels que celui de la "Green Fluorescent Protein"-GFP et autres protéines fluorescentes), iv) des mesures d’activités métaboliques de la cellule (biosenseurs): dosage de calcium, pH, potentiel membranaire, poussées oxydatives, glutathion..., v) des analyses immunologiques, vi) le tri de cellules animales, de levures, de bactéries, de protoplastes et d’organites cellulaires. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 70 équipes de recherches et 15 plateformes technologiques, provenant de 8 unités de recherches (CGM, IBBMC, IGM, ISV, LEBS, VMS, SB2SM, SBiGeM). L’institut est réparti sur 3 sites de recherche (Campus d’Orsay Vallée de l’Université Paris-Saclay, Campus du CNRS de Gif sur Yvette et Campus du CEA / Centre de Saclay) au sein de 14 bâtiments jusqu’au rassemblement programmé en 2020 sur le campus du CNRS de Gif-sur-Yvette.

|
Scooped by
Life Sciences UPSaclay
June 20, 2021 5:33 PM
|
Localisée au Centre des matériaux de MINES ParisTech, la plateforme de microscopie électronique de Genopole est accessible aux entités franciliennes ayant besoin d’analyses en microscopie électronique à balayage (MEB) ou en transmission (MET). Fabrice Gaslain, ingénieur spécialisé en MEB (photo de gauche), apporte son expertise aux biologistes souhaitant analyser leurs échantillons sur l’un des MEB comme il nous le confie : « j’oriente systématiquement les chercheurs en biologie sur le Nova NanoSEM 450 (ThermoFisher) qui est le plus adapté car sa colonne haute résolution permet d’atteindre le nanomètre via un grandissement allant jusqu’à x200.000. Sa largeur de champ comprise entre 3,6 mm et 200 nm et son mode low-vacuum sont adaptés aux préparations biologiques. Ce MEB dispose de nombreux détecteurs pour l’imagerie et la microanalyse X, et d’un système d’acquisition multi-champs automatique permettant de réaliser très facilement une mosaïque de l’échantillon observé. Ceci est toujours très apprécié ! ». Son collègue Mohamed Sennour, ingénieur spécialisé en MET (photo de droite), utilise quant à lui quotidiennement le microscope Tecnai F20-ST (ThermoFisher), très performant et parmi les plus polyvalents de la région : « En plus de son optique permettant à la fois l’observation de champs très larges (caméra CCD grand-angle) et d’atteindre la résolution atomique (2.4 Å), ce MET opérant à 200 kV peut également fonctionner en mode balayage en transmission (STEM) en combinant plusieurs détecteurs d’images. Également équipé d’un porte-objet spécial (± 80°), il permet de réaliser de la tomographie électronique. Tous ces dispositifs intégrés permettent des observations et des analyses simultanées sur des échantillons biologiques en mettant en œuvre une dizaine de techniques différentes : diffraction, imagerie conventionnelle, haute résolution et de contraste de numéro atomique, analyses chimiques par dispersion d’énergie de rayons X (EDX) ou des électrons (EELS), tomographie, etc. » avant d’ajouter « ainsi les chercheurs génopolitains qui viennent utiliser le MET ont le choix des techniques d’analyses ! ». La plateforme de microscopie électronique de Genopole est accessible à l’ensemble de la communauté francilienne, laboratoires publics ou entreprises privées, implantées en Région Ile-de-France. Alors si vous avez des besoins en microscopie électronique, n’hésitez pas, contactez-nous ! Contact : Julien Picot (Julien.Picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ICI GENOPOLE / Plateforme de Microscopie électronique. La plateforme de microscopie électronique, mise en place par Genopole, l’Inserm et MINES ParisTech (UMR CNRS 7633) accueille, à parts égales, des activités de biologie et de physique des matériaux. Les domaines d’activités pour la biologie cellulaire et tissulaire sont d’une part, la morphologie et l’immunocytochimie ultra-structurale et, d’autre part, le développement de nanoparticules pour la vectorisation de protéines. Les domaines d’activités liés à la physique des matériaux sont, quant à eux, l’analyse de nanomatériaux et de nouveaux alliages, l’endommagement, la structure et les propriétés des interfaces et enfin l’analyse de procédés de protection des matériaux et des multimatériaux. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 77 entreprises de biotechnologies, 18 laboratoires de recherche, 26 plateformes technologiques, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole, dirigé par Gilles Lasserre, est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
May 29, 2021 4:29 AM
|
La plateforme Chiral-IST (Faculté de Pharmacie, Université Paris-Saclay) propose à des équipes de recherche académiques ou à des entreprises du territoire Paris-Saclay d’étudier la stabilité et la conformation de protéines par dichroïsme circulaire et fluorescence. Retour sur une collaboration réussie impliquant une équipe de recherche d’une Université brésilienne, une équipe INRAE / Université de Tours (UMR 1282, Infectiologie et Santé Publique) et Chiral-IST. Courant 2017, Chiral-IST est contactée par Philippe Billiald (MCAM, UMR MNHN-CNRS 7245, Muséum national d’Histoire naturelle, Paris), ce dernier collaborant avec une équipe brésilienne (Universidade Federal do Paraná, Curitiba) sur l'envenimation due aux piqûres d'araignées Loxosceles, une maladie négligée et un problème médical majeur en Amérique du Sud. À ce jour, il n'y a pas de consensus pour le traitement des patients envenimés, et des anti-venins polyclonaux de cheval sont généralement perfusés aux patients atteints. Pourtant, il est bien connu que les protéines de venin dans la gamme 30–35 kDa avec la sphingomyélinase D (SMasesD), reproduisent la plupart des effets toxiques observés dans le loxoscelisme. Des fragments d'anticorps monoclonaux ciblant ces toxines pouvant constituer une alternative sûre et un traitement effectif, ont donc été étudiés … et c’est à partir d’un anticorps monoclonal ciblant les SMasesD du venin de L. intermedia et neutralisant son activité dermonécrotique, que nous avons conçu les domaines variables d'anticorps humanisés, sous forme de chaîne unique recombinante de fragments d'anticorps (scFv). Ces molécules ont ensuite été caractérisées en termes d'humanisation, d’activité et neutralisation du venin et pour la stabilité de structure par notre plateforme. L'analyse in silico des interactions antigène/anticorps acides aminés a contribué à la compréhension du mécanisme de neutralisation et conduit à reformater le fragment d'anticorps humanisé pour récupérer les caractéristiques fonctionnelles pour une neutralisation efficace, in vitro, du venin. Ce travail a démontré la fonctionnalité de l’anticorps pour une approche thérapeutique et l’étude pris en charge par Chiral-IST (dichroïsme circulaire, voir figure) a permis de caractériser sa stabilité jusqu’à 40°C. La suite ? Pour une utilisation en milieu tropical, la nécessité d’améliorer la stabilité thermique de cet anticorps par « Protein engineering » a été requise (50°C à minima) pour lui permettre une utilisation pratique thérapeutique en conditions locales. En savoir plus ? Karim-Silva et al., Toxins 2020. Contact : Arnaud Leroy (arnaud.leroy@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI CHIRAL-IST (CHIRALité et Innovation Spectroscopique et Technologique). La plateforme propose la caractérisation spectroscopique (propriétés dichroïques et fluorescence) de molécules en solution et dans différents environnements liquides, et leurs interactions avec une autre molécule partenaire (ex : interaction protéine-protéine ou protéine-petite molécule). Les molécules concernées par nos analyses peuvent être des composés de petit poids moléculaire et d’intérêt thérapeutique, issus de synthèse, d’hémi-synthèse ou de purification, ou des macromolécules biologiques (peptides synthétiques, protéines et fragments d’ADN).

|
Scooped by
Life Sciences UPSaclay
May 16, 2021 5:06 PM
|
Clostridium difficile est une bactérie entéropathogène responsable de 15 à 25% des diarrhées consécutives à une antibiothérapie. Un traitement à base de métronidazole est en place, cependant, une modification de sensibilité de certaines souches, des rechutes plus fréquentes et donc une moins bonne efficacité du traitement au métronidazole ont été observées. Séverine Péchiné (Equipe Bactéries Pathogènes et Santé, pôle Adaptation Bactérienne et Pathogénèse – Institut Micalis) et sa doctorante Tri-Hanh-Dung Doan, investiguent alors les raisons possibles de cette diminution d’efficacité. Des essais préliminaires montrent que, contrairement aux souches de Clostridium difficile sensibles, pour des souches de sensibilité diminuée au métronidazole, une concentration sub-inhibitrice induit in vitro et en 48 heures une augmentation par 4 de la production d’un biofilm, et une modification morphologique par élongation des bactéries. La plateforme de protéomique de l’IPSIT est alors appelée en renfort, pour analyser, par chromatographie liquide couplée à la spectrométrie de masse (LC-MS), le protéome de 2 souches de Clostridium difficile de sensibilités différentes au métronidazole. Ainsi, dans un véritable coup de filet, IPSIT / Plateforme de protéomique met en évidence des différences qualitative et quantitative pour 665 protéines exprimées par les bactéries en présence de concentrations différentes de métronidazole. Bien qu’en l’absence de métronidazole les souches présentent des protéomes similaires, elles adoptent des réponses hétérogènes lorsqu’elles sont soumises au stress antibiotique. Parmi les nombreuses observations faites, dans le cas de la souche la moins sensible au métronidazole, la synthèse de deux protéines particulières, la protéase (Cwp84) et une aminotransférase de la famille de MocR impliquées dans la formation du biofilm, a été altérée par le métronidazole. Cette modification pourrait expliquer la production plus importante de biofilm observée préalablement. Les protéines impliquées sont maintenant identifiées et mises en examen, reste à déterminer les connexions et les rouages de cette machination. Ces premières conclusions corroborent donc les autres en cours dans la lutte contre le développement de résistance aux antibiotiques. Enquête à suivre. En savoir plus ? Tri-Hanh-Dung Doan et al., PLOS ONE, 2020. Contact : Stéphanie Nicolay (stephanie.nicolay@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IPSIT / Plateforme de protéomique. L’UMS IPSIT, pour Ingénierie et Plateformes au Service de l’Innovation Thérapeutique, est une Unité Mixte de Service de l’Université Paris-Saclay qui regroupe 10 plateformes technologiques, à l’interface entre les domaines de la biologie, de la chimie et des sciences médicales. La plateforme de Protéomique, intégrée au sein du réseau de Plateformes de Protéomique Paris-Saclay (P2S), ouvre son expertise aux différents acteurs de la recherche, qu’ils soient chercheurs de l’UFR de Pharmacie ou laboratoires extérieurs, de statut public ou privé. Dans le cadre de l’étude du protéome, la plateforme de protéomique utilise une approche dite Bottom-Up. Cette dernière implique de digérer dans un premier temps les protéines, puis d’analyser le mélange peptidique obtenu par nano-chromatographie liquide couplée à un spectromètre de masse haute résolution. Les peptides sont alors élués, détectés en fonction de leur rapport masse/charge et fragmentés de manière contrôlée. Les informations obtenues sur la masse exacte des peptides et la séquence en acides aminés permettent alors de remonter à l’identification des protéines et à leur quantification relative. Selon leur nature, les modifications post-traductionnelles (MPT) peuvent être étudiées, tout dépend de leur stabilité dans les conditions d’analyse en place sur la plateforme.

|
Scooped by
Life Sciences UPSaclay
May 2, 2021 4:18 PM
|
Créée en 2017, la plateforme d’histologie – anatomocytopathologie de Genopole propose les équipements, le savoir-faire et l’expertise nécessaires pour répondre aux besoins croissants des entités du biocluster Genopole dont l’activité est liée au domaine de la Santé. Les prestations de service proposées par cette plateforme sont tout naturellement la préparation des tissus (cryoconservation, inclusion en paraffine ou en résine), leur section (cryosection, microtomie ou ultra-microtomie), leurs colorations histologiques dites classiques ou bien la réalisation de marquages immunohistochimiques (DAB ou fluorescents) via des automates et enfin leur exploration en microscopie en vue d’une analyse morphométrique précise des tissus observés. Pour y arriver, des technologies et des compétences complémentaires sont requises à chacune de ces étapes. D’autre part, l’expertise sur lames par les médecins pathologistes du Centre Hospitalier Sud Francilien (CHSF, Corbeil-Essonnes) peut, dans certains cas, constituer l’étape finale de cette chaine de préparation des tissus, étape qui nécessite un savoir-faire et une technicité certaine afin de préserver la structure des tissus parfois très fragiles et précieux. Vous l’aurez compris, la singularité de la plateforme d’histologie – anatomocytopathologie est qu’elle s’appuie sur l’expertise des membres du service « anatomie & cytologie pathologiques - ACP » du CHSF dont l’effectif vient d’augmenter significativement suite au regroupement avec le service du même nom du Groupe Hospitalier Nord Essonne (Juvisy-sur-Orge / Longjumeau / Orsay) comme nous l’indique le Dr. Virginie CAHN, Cheffe du Service ACP du CHSF : « En effet, trois médecins pathologistes, cinq techniciens, une aide de laboratoire et une secrétaire médicale sont venus renforcer l’équipe déjà présente. Nous sommes tous ravis de cette nouvelle dynamique de groupe mais également de pouvoir collaborer avec les entités génopolitaines qui nous en font la demande, à l’image de la société Xentech par exemple, avec qui nous collaborons depuis de nombreuses années » avant d’ajouter « c’est un réel plaisir pour nous de pouvoir diversifier notre activité et de mettre à disposition notre savoir-faire et nos compétences au service de projets aussi intéressants qu’ambitieux ». Si vous avez des besoins en préparation de tissus, en coloration ou en expertise sur lames, n’hésitez pas, contactez-nous ! Contact : Julien Picot (julien.picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ici GENOPOLE / Plateforme d'histologie-anatomocytopathologie. La plate-forme d'histologie-anatomocytopathologie offre des équipements et le savoir-faire associé nécessaire à la préparation des tissus, leurs colorations histologiques et/ou immunohistochimiques en vue de leur exploration par imagerie photonique ou électronique. Cette plate-forme s'appuie sur l'expertise de médecins anatomocytopathologistes du Centre Hospitalier Sud-Francilien pour la bonne réalisation de projets précliniques et cliniques. A propos de Genopole. Biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 78 entreprises de biotechnologies, 18 laboratoires de recherche, 26 plateformes technologiques, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole, dirigé par Gilles Lasserre, est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).
|

|
Scooped by
Life Sciences UPSaclay
November 19, 2021 10:13 AM
|
Depuis 2012, le département IDMIT (CEA, Institut de biologie François Jacob) encore « IMETI/SIV » à ce moment-là s’efforce de mettre en place un système de gestion connecté et sécurisé de ses données de laboratoire. La plateforme informatique et bioinformatique (IBI) et son équipe lance alors le projet BATLab. Déposé à l’Agence de Protection des Programmes, le logiciel web intègre alors petit à petit des fonctionnalités de LIMS pour la gestion des projets, autorisations, équipements et autres résultats d’expériences. Pour des raisons d’optimisation et d’interopérabilité entre les différents modules, la planification, le recensement et la recherche des prélèvements et échantillons devient vite incontournable : BATLab fait dès lors office de centre de ressources biologiques au sein du département, avec des centaines de nouvelles entrées chaque jour étiquetés par code-barres. Enfin, pour le pilotage de ses animaleries uniques en Europe, il dispose de plusieurs modules dédiés à la gestion des différentes zones ainsi qu’à la planification des interventions et observations cliniques, le tout développé dans un le cadre ISO9001 d’IDMIT et orienté AAALAC. Aujourd’hui, il est le centre névralgique des données et de la gestion du département ; la sécurité et la flexibilité de son utilisation font qu’il est déployé sur différents sites partenaires, intra et extra CEA, notamment, au CIPHE (Centre d’immunophénomique) de Marseille, et différents autres sites en cours de déploiement. Contact: Brice Targat (brice.targat@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IDMIT / Plateforme informatique et bioinformatique (IBI). Au sein du département IDMIT de l'Institut François Jacob, l'équipe IBI développe et maintient depuis 2012 un logiciel de gestion de données de laboratoire (LIMS) nommé BATLab. Grâce à sa situation et aux interactions quotidiennes avec les acteurs de l'immunologie, il est devenu la clé de voute de l'organisation d'IDMIT. Les nombreux projets de nos différents laboratoires y sont planifiés des mois à l'avance pour gérer et optimiser le calendrier de nos plateformes, tout en gardant une grande flexibilité pour s'adapter aux besoins de dernière minute, dont le récent COVID-19. La gestion de nos animaleries primates (A1, A2, A3, A3 aéro) y est également essentielle, et la préoccupation du bien-être animal (AAALAC) est omniprésente. Notre activité repose également sur une gestion critique de notre centre de ressources biologiques (CRB) et nos stocks de réactifs, anticorps, médicaments, (...) ou autre pathogènes. Les Micro-Organismes et Toxines (MOT) et les études s'y rapportant y sont par ailleurs gérés avec une attention particulière qui a pleinement satisfait l'ANSM. Les résultats d'expérience des différentes plateformes (cytométrie, multiplex, charges virales, nfs …) y sont hébergés et visualisés – comme le reste des informations du LIMS – à l'aide du logiciel de visualisation de données Tableau Software. En fin de pipeline, BATLab gère finalement certains aspects de facturation et d'unités d'œuvre, dont les couts d'hébergement des animaux pour les différents projets. A propos d’IDMIT. IDMIT (Infectious Disease Models and Innovative Therapies) est une infrastructure nationale en biologie et santé (INBS) dédiée aux recherches précliniques sur les maladies infectieuses humaines. Ses fondateurs institutionnels sont le CEA, l’Institut Pasteur, l’INSERM mais aussi l’ANRS (Agence nationale de Recherche sur le SIDA et les Hépatites Virales) et l’Université Paris-Saclay. Localisée sur le centre CEA de Fontenay-aux-Roses, IDMIT offre aujourd’hui à la communauté scientifique nationale et internationale une infrastructure unique en Europe, une expertise scientifique, différents modèles d’infections virales humaines - notamment chez le primate non-humain (PNH) - pour l’étude de l’infection par le VIH, le SARS-Cov2, les virus de la grippe, du chikungunya, de la dengue, de la fièvre jaune, et également par les agents du paludisme, de la coqueluche, et des chlamydioses. Ses objectifs ? i) d’étudier la pathogénèse des maladies infectieuses humaines dans des modèles in vivo, ii) caractériser les réponses immunes innées et adaptatives aux infections, iii) étudier les interactions hôtes-pathogènes et enfin, iv) développer des modèles précliniques pour évaluer l’efficacité de nouvelles stratégies préventives et thérapeutiques, notamment chez le PNH.

|
Scooped by
Life Sciences UPSaclay
November 6, 2021 2:03 PM
|
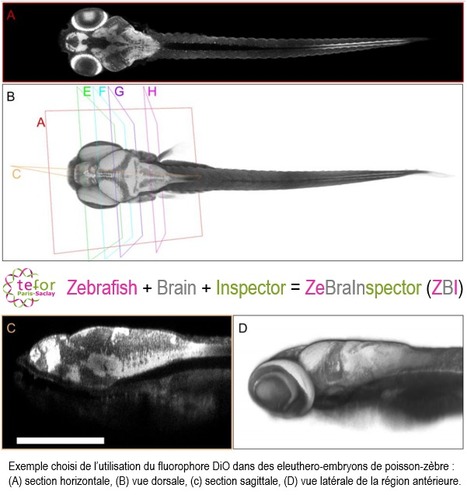
Rattachée à l’unité de services TEFOR Paris-Saclay, TPS-AQUA (ou TEFOR Paris-Saclay / Zootechnie aquatique) s’installe au sein du bâtiment de l’Institut des Neurosciences Paris-Saclay (NeuroPSI) sur le pôle technologique Paris-Saclay. Elle y disposera de 1600 m² d’animalerie aquatique et constituera un pôle français d’exception en recherche sur les modèles aquatiques et l’étude de leur bien-être. Vous souhaitez utiliser des modèles vertébrés aquatiques pour mener à bien vos recherches ? TPS-AQUA (TEFOR Paris-Saclay / Zootechnie aquatique) est là pour vous y aider ! TPS-AQUA, c’est une offre de modèles aquatiques diversifiée… La vedette des espèces modèles aquatiques : le poisson-zèbre Danio rerio, poisson de rivière, originaire d’Asie, est très populaire auprès des chercheurs dans de nombreux domaines (neurosciences, toxicologie, biologie du développement, cardiologie, etc.). Sa petite taille (4 à 5 cm à l’âge adulte), son développement rapide et la transparence des stades embryonnaires en font un animal alliant facilité d’élevage et fort intérêt scientifique ; Un autre poisson de rivière originaire d'Asie, plus précisément du sud-est, le médaka Oryzias latipes, célèbre pour sa capacité à vivre dans des conditions environnementales variées et pour la rapidité de ses réponses à différentes drogues, justifiant sa large utilisation dans le domaine de la toxicologie ; Deux modèles de xénopes : Xenopus laevis et Xenopus tropicalis ; D’autres espèces de poissons les rejoindront : des cichlidés (Neolamprologus brichardi, Maylandia spp.), danionella (Danionella translucida) et killifish (Nothobranchius furzeri). TPS-AQUA, c’est également une offre de services diversifiée… Pour chacune des espèces mentionnées ci-dessus, plusieurs services sont accessibles, tels que i) l’hébergement de vos lignées sauvages ou modifiées, ii) la fourniture de tissus, d’œufs, de larves ou d’individus adultes et iii) un accueil scientifique temporaire et ce, en collaboration avec les autres équipes de l’unité TEFOR Paris-Saclay, pour vous aider à monter de bout en bout votre projet (hôtel à projet « clé en main »). Enfin, TPS-AQUA anime aussi des formations réglementaires pour l’utilisation des espèces aquatiques. Les modèles aquatiques, c’est clairement leur branch(i)e d’expertise et une équipe dédiée ! TPS-AQUA c’est une équipe de 5 agents experts : quatre zootechniciens entourent la responsable Marie-Elise Schwartz (CNRS). Tous ensemble, ils assurent les activités d'élevage et maintiennent les animaux selon des protocoles standardisés et optimisés pour améliorer en tout point le bien-être animal. Contact : Marie-Elise Schwartz (marie-elise.schwartz@cnrs.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
October 24, 2021 1:05 PM
|
La plateforme Anaxem de l’Institut Micalis (INRAE, Jouy-en-Josas) propose à la communauté scientifique, académique comme industrielle, de Paris-Saclay, des animaux axéniques (dépourvus de microbiote) ou à microbiote contrôlé et les équipements nécessaires pour étudier le dialogue entre les microbiotes commensaux et leur hôte. La majorité des animaux utilisés sur la plateforme sont des rongeurs (rats et souris) mais une autre espèce animale est aussi présente : la caille du Japon (Coturnix japonica) ! Cette dernière est utilisée comme modèle d’étude d’une grave maladie du nouveau-né prématuré, l’entérocolite ulcéronécrosante (ECUN). L’incidence de cette maladie est de 2-7% chez les grands prématurés (naissance avant la 32ème semaine d’aménorrhée) et de 5-22% chez ceux dont le poids à la naissance est inférieur à 1000 g. La maladie apparaît de manière brutale et imprévisible et peut conduire à la mort de l’enfant dans 20 à 30% des cas. Son origine est encore mal connue. Elle est très probablement multifactorielle : interviennent la nutrition entérale, l’immaturité intestinale (motilité intestinale réduite, production insuffisante d’enzymes digestives, en particulier de lactase) et des anomalies dans la colonisation bactérienne de l’intestin. En particulier des espèces du genre Clostridium, telles que C. butyricum, C. neonatale et C. perfringens, sont fréquemment isolées des sujets atteints. Compte tenu de ces éléments, la caille du Japon est un modèle très intéressant pour reproduire les lésions de l’ECUN : comme tous les oiseaux, et contrairement aux mammifères, son tube digestif ne produit pas de lactase ; son gros intestin comporte deux longs caeca flexueux, organes creux dont la motilité est réduite et dans lesquels peut se développer un microbiote abondant. En colonisant des cailles axéniques avec des souches de Clostridium isolées de prématurés atteints d’ECUN, et en les nourrissant avec un aliment contenant du lactose à la même concentration que dans le lait, on reproduit en quelques semaines des lésions intestinales typiques de la maladie : pneumatose, inflammation, hémorragie, nécrose. En savoir plus ? Szylit O. et al., The Lancet 1997, Aires J. et al., Impact 2016 et Bellet D. et al., STAL 2017. L’équipe du Prof. Julio Aires (Université de Paris, INSERM, UMR 1139 3PHM, Faculté de Pharmacie de Paris, FHU Prema, Paris) utilise régulièrement le modèle de caille axénique de la plateforme Anaxem pour étudier le rôle et les mécanismes d’action de différentes souches bactériennes de Clostridium dans la pathogénie de l’ECUN mais aussi pour tester les effets préventifs de probiotiques. Contact : Sylvie rabot (sylvie.rabot@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI MICALIS / Animalerie axénique (Anaxem). L'installation expérimentale Anaxem est une animalerie de rongeurs et oiseaux sans microbiote (axéniques) ou à microbiote contrôlé, maintenus en isolateurs. Elle propose aux équipes de recherche de l'Institut Micalis, d'INRAE et d'autres organismes (académiques et privés), les animaux, les infrastructures et l'assistance technique pour mener des protocoles expérimentaux dédiés à l'étude du dialogue entre les microbiotes commensaux et leur hôte. A propos de l’Institut Micalis. Micalis est une unité mixte de recherche (UMR) associant l’INRA et AgroParisTech et faisant partie de l’Université Paris-Saclay (UPSaclay). Sa mission est le développement de recherches novatrices dans le champ de la « Microbiologie de l’Alimentation au service de la Santé ».

|
Scooped by
Life Sciences UPSaclay
October 9, 2021 5:11 PM
|
Cerner l’ennemi (connaitre son identité et son histoire), repérer l’envahisseur dès que possible, décrypter les plans d’attaque du virus, analyser les dommages, vaincre l’adversaire et mieux nous protéger… C’est au travers de ces cinq actions que le GIS IBiSA fait le point sur l’implication de ses plateformes dans la lutte contre la Covid-19. Cet article publié sur le site web d’IBiSA et relayé dans sa dernière newsletter dévoile une liste de travaux de recherche innovants et prometteurs qui témoigne de la forte contribution des plateformes. Le GIS IBiSA est fier de les représenter, de les soutenir et d’encourager cette dynamique, qui montre par ailleurs leur capacité à interagir entre elles, avec les équipes de recherche et le secteur médical, à l’échelle locale, nationale, voire internationale. Parmi les plateformes mises en avant dans l’article et relevant du territoire Paris-Saclay, vous trouverez celles de l’Institut de biologie François Jacob (MIRCen, IDMIT) et de l’Institut des sciences du vivant Frédéric JOLIOT (DMTS/ProGénoMix), également indexées dans PlugInLabs Université Paris-Saclay ! Vous souhaitez recevoir la newsletter d’IBiSA publiée chaque trimestre ? Inscrivez-vous ! Vous souhaitez connaitre les plateformes de l’écosystème Paris-Saclay labélisées IBiSA ? Vous avez besoin d'un équipement de pointe ou des compétences d'une plateforme en sciences du vivant pour faire progresser vos travaux de recherche ? L'annuaire IBiSA vous aide à identifier les structures appropriées sur le territoire national ! A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle du CEA, du CNRS, de l'INRAE, de l'Inria, de l'Inserm, de l’INCa, de la CPU et du Ministère de l’enseignement supérieur, de la recherche et de l’innovation (MESRI), il est l’unique instrument de financement commun à l’ensemble des établissements en sciences du vivant, Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques, et accompagne les démarches qualité en vue de la structuration et certification des plateformes. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire ! Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaire académique comme entreprise, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
September 25, 2021 5:46 AM
|
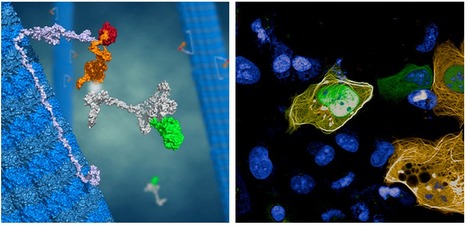
La plateforme de protéomique de Gif-sur-Yvette, créée en 2007 sous le nom de SICaPS (Service d’Identification des Protéines par Spectrométrie de masse), récemment rebaptisée Protéomique-Gif, fait partie intégrante de l’Institut de Biologie Intégrative de la cellule (I2BC, Gif-sur-Yvette) depuis 2015. Elle couvre différents aspects de la protéomique et des analyses de protéines par spectrométrie de masse : du contrôle qualité de protéines à l’analyse en profondeur des interactomes, des protéomes et de leurs modifications. Protéomique-Gif offre un ensemble de spectromètres de masse, de stratégies et de méthodes de préparation des échantillons nécessaires pour identifier et quantifier des protéines et leurs modifications chimiques, co- et post-traductionnelles. L’un des spectromètres de masse, le timsTOF Pro de Bruker (obtenu grâce à des financements SESAME Santé IdF 2017, Plan Cancer 2018, IBiSA, Université Paris-Saclay et CNRS) permet désormais d’identifier plusieurs milliers de protéines à partir de 50 à 200 ng de protéines. Mais que sont des analyses protéomiques ? Des analyses protéomiques consistent à étudier l’ensemble des protéines d’un compartiment cellulaire, d’une cellule, d’un tissu, d’un organisme ou d’un fluide biologique. Cet ensemble de protéines, nommé protéome, résulte de la combinatoire de l’expression des gènes et des modifications post-transcriptionnelles, co- et post-traductionnelles parfois complexes. Des stratégies analytiques dédiées, mettant en jeu des digestions de protéines, des enrichissements de peptides, des séparations chromatographiques et des analyses par spectrométrie de masse, ainsi que des analyses bio-informatiques et statistiques des données générées, permettent d’identifier, quantifier et caractériser finement les protéines d’un échantillon biologique à un instant donné. Elles permettent d’étudier les changements d’expression des protéines, leurs modifications et les interactions qu’elles établissent avec des partenaires protéiques qui contribuent à réguler les fonctions normales et pathologiques des protéines. Un exemple d’application de ces approches et expertises ? Regard sur un interactome choisi ! L’agrégation des protéines Tau et la propagation de ces agrégats dans le cerveau sont liées à la progression de la maladie d’Alzheimer. Les agrégats de Tau se propagent d’une cellule neuronale à une autre en se liant aux membranes des cellules, avant d’être internalisés et amplifiés dans la cellule réceptrice. L’étape clé dans la propagation de ces agrégats pathogéniques est la fixation d’agrégats provenant de cellules neuronales affectées aux membranes de cellules indemnes. Par une approche protéomique de pêche des partenaires des agrégats de protéines Tau fibrillaire, l’équipe de Ronald Melki (MIRCen, CNRS/CEA/Université Paris-Saclay, Laboratoire des Maladies Neurodégénératives, Fontenay-aux-Roses), en collaboration avec l’équipe d’Antoine Triller (Ecole Normale Supérieure, Sorbonne Université et l’Inserm) et la plateforme Protéomique-Gif, a identifié l’interactome membranaire de neurones de souris des agrégats de Tau pathogéniques, et a ainsi pu identifier les cibles de ces agrégats. Ce travail a permis de documenter les conséquences fonctionnelles de l’interaction des agrégats de Tau avec notamment la pompe sodium/potassium et des récepteurs du glutamate, des protéines essentielles à la survie des neurones. En savoir plus ? Shrivastava A et al., EMBO J. 2019. Cette étude illustre l’intérêt et le potentiel des approches de protéomique et le savoir-faire de la plateforme Protéomique-Gif. L’accès au timsTOF Pro et a ses nouvelles performances a récemment permis d’accéder à une profondeur d’analyse des protéomes plus grande encore. Et de nouvelles sensibilités pourraient encore être atteintes dans le futur… Les analyses d’interactomes, de protéomes complexes ou des caractérisations fines de protéines vous intéressent, n’hésitez pas à nous contacter et à nous soumettre vos demandes. Nous élaborerons ensemble la stratégie la plus adaptée à la réalisation de votre projet. Contact : Virginie Redeker (virginie.redeker@cnrs.fr) Plug In Labs Université Paris-Saclay : cliquer ici La plateforme Protéomique-Gif (SICaPS), labellisée IBiSA, est ouverte à l'ensemble de la communauté scientifique. La plateforme propose des méthodologies de protéomique et des technologies de spectrométrie de masse de pointe pour identifier, caractériser et quantifier les protéines et certaines de leurs modifications à partir d'échantillons plus ou moins complexe. Une technologie de dernière génération permet l'identification et la quantification relative de protéines peu abondantes dans des échantillons protéiques très complexes. Des méthodes analytiques adaptées permettent de caractériser des modifications co-, post-traductionnelles ou chimiques des protéines (phosphorylations, lipides, pontages covalents …). La plateforme dispose d'une solide expertise dans la préparation d'échantillons protéiques de nature variée, leur analyse par spectrométrie de masse et le traitement des données. Elle optimise et développe des méthodes analytiques en réponse aux besoins des utilisateurs. La plateforme propose une variété de services sous forme de prestations : i) standards : préparations d'échantillons, mesures de masse exacte, identification de protéines, contrôle-qualités de protéines recombinantes ou ii) collaboratives : quantification relative des protéines, comparaisons approfondies de protéomes, interactomes, caractérisation de déterminants structuraux et de modifications des protéines.

|
Scooped by
Life Sciences UPSaclay
September 11, 2021 7:03 AM
|
La plateforme de biologie structurale hébergée par le laboratoire Structure et Activité des Biomolécules Normales et Pathologiques (SABNP, Inserm UMR-S 1204 – Université d’Evry-Val-d’Essonne – Université Paris-Saclay), situé à Genopole, enrichit son offre technologique d’un imageur HCS (high content screening) de haute résolution : l’Opera Phenix+ (Perkin Elmer). David Pastré, directeur du laboratoire nous confie que « grâce à Genopole et à l’Université d’Évry, mon équipe bénéficie aujourd’hui d’un imageur combinant haut-débit et haute-résolution, indispensable au développement à grande échelle de notre nouvelle technologie brevetée – MicroTubule Bench (MTbench) – mesurant les interactions biomoléculaires, notamment ARN-protéine, dans des cellules vivantes ». L’arrivée de cet imageur donne les moyens de tirer parti du haut potentiel de la technologie développée par le laboratoire et brevetée par Inserm Transfert, grâce à l’acquisition d’une importante quantité d’images dans un temps relativement court (63,000 images en seulement 5 heures) : en multipliant le nombre d’images par test, la fiabilité de ces derniers augmente également. Comme écrit ci-dessus, MTbench est une méthode novatrice pour étudier un phénomène clé du fonctionnement du vivant : les interactions protéine-protéine et ARN-protéine. La méthode les détecte et les quantifie dans les cellules vivantes en utilisant une structure naturelle de la cellule : le réseau de microtubules. David Pastré ajoute que « sa technologie MTbench promet de nombreuses applications, en particulier pour tester l’action ou découvrir de nouveaux médicaments et pour mieux connaitre les dérèglements associés aux maladies. Elle peut par exemple explorer dans des cellules vivantes l’impact de mutations responsables de pathologies, aider à en comprendre le mécanisme, puis à rechercher des molécules thérapeutiques capables de corriger l’effet de ces mutations ». L’imageur HCS à haute résolution Opera Phenix+ apporte ainsi à la plateforme mutualisée de biologie structurale un équipement de très haute technologie qui, associé à la méthode MTbench, inédite et unique au monde, et à l’expertise de l’équipe, ouvre notamment à des études d’interactions protéine-protéine et ARN-protéine d’intérêt majeur en santé humaine, qui pâtissaient jusque-là d’un déficit de technologies. David conclut en rappelant que « cet équipement de pointe est accessible à la communauté scientifique publique et privée ! ». Pour connaitre les modalités d’accès à la plateforme de Biologie structurale, n’hésitez pas, contactez-nous ! Contact : Julien Picot (Julien.Picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ici GENOPOLE / Plateforme de biologie structurale. La plate-forme de biologie structurale offre des équipements de pointe et l’expertise associée en spectroscopie RMN et en microscopie à force atomique. Concernant la spectroscopie RMN, les domaines d’activités sont d’une part l’analyse de la structure de protéines en solution, leur repliement, leur stabilité et leur dynamique en solution, d’autre part l’analyse des interactions ligand-protéine, protéine-protéine et protéine-acides nucléiques. Concernant la microscopie à force atomique, les domaines d’activités sont la caractérisation d’objets biologiques à l’échelle nanométrique, l’observation à l’air ou en milieu liquide de molécules uniques (ADN ou protéines), et l’étude de la formation de complexes ADN-ligands, protéine-protéine et microtubules-partenaires. Enfin, une partie de l’activité de la plate-forme concerne la modélisation de la dynamique moléculaire. La plate-forme de Biologie structurale fait partie de l’infrastructure nationale FRISBI. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 77 entreprises de biotechnologies, 18 laboratoires de recherche, 26 plateformes technologiques, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole, dirigé par Gilles Lasserre, est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
August 29, 2021 10:08 AM
|
Danio rerio se fait appeler Danio zébré, Danio rayé, petit Danio, poisson zèbre et parfois même, poisson pyjama. Convoité par bon nombre de scientifiques pour sa transparence, ses facilités de reproduction et d’élevage, il est le deuxième organisme modèle de laboratoire le plus utilisé dans le monde. Jean-Stéphane Joly présente ce petit poisson sans équivalent, emblème de TEFOR Paris-Saclay à l’honneur dans la newsletter trimestrielle d’IBiSA. Découvrez-le maintenant ! Parmi les autres actualités du trimestre… Bilan de l’appel d’offres Plateformes IBiSA 2020. Sur avis du conseil scientifique, le comité de direction du GIS s’est prononcé début novembre : 6 plateformes ont été labellisées IBiSA et 2 683 000 € ont été distribués à 40 structures labellisées en biologie, santé et agronomie. Retour sur l’édition 2020 de l’appel d’offres Plateformes. Un nouveau réseau de compétences pour les plateformes IBiSA. Le GIS a mis en place un réseau de compétences transversales proposant aux plateformes labellisées des journées thématiques pour répondre à des besoins autres que scientifiques et technologiques. Ces ateliers visent à soutenir le fonctionnement quotidien des plateformes et à créer du lien entre leurs gestionnaires. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! Vous souhaitez connaitre les plateformes de l’écosystème Paris-Saclay labélisées IBiSA ? Vous avez besoin d'un équipement de pointe ou des compétences d'une plateforme en sciences du vivant pour faire progresser vos travaux de recherche ? L'annuaire IBiSA vous aide à identifier les structures appropriées sur le territoire national ! A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle du CEA, du CNRS, de l'INRAE, de l'Inria, de l'Inserm, de l’INCa, de la CPU et du Ministère de l’enseignement supérieur, de la recherche et de l’innovation (MESRI), il est l’unique instrument de financement commun à l’ensemble des établissements en sciences du vivant, Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques, et accompagne les démarches qualité en vue de la structuration et certification des plateformes. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire ! Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaire académique comme entreprise, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
July 9, 2021 6:30 PM
|
Plateforme est un terme inscrit dans l’ADN de l’Université Paris-Saclay et notamment son domaine Sciences de la Vie. Sous cette dénomination générique et utilisée de manière souple à Paris-Saclay, se retrouvent référencées et visibles via l’interface Plug In Labs Université Paris-Saclay, plus de 200 entités - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements – parfois uniques – ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. Un autre point commun à toutes ces plateformes est la volonté d’être aux services de vos demandes – collaborations de recherche ou prestations – que vous soyez un partenaire public ou privé, appartenant à l’écosystème Paris-Saclay ou non. Cette ouverture se caractérise par ailleurs pour près de 20% d’entre elles par le label IBiSA, souvent associé à une certification de type ISO 9001 ou NFX 50-900. Vous souhaitez connaitre les plateformes de Paris-Saclay labélisées IBiSA ? Vous avez besoin d'un équipement de pointe ou des compétences d'une plateforme en sciences du vivant pour faire progresser vos travaux de recherche ? L'annuaire IBiSA vous aide à identifier les structures appropriées sur le territoire national. Aussi, le site web d’IBiSA a récemment fait peau neuve - nouveau logo, nouvelles rubriques - et une newsletter trimestrielle est maintenant programmée. Vous souhaitez la recevoir ? Inscrivez-vous ! Vous souhaitez tout simplement découvrir les missions d’IBiSA, son équipe, son histoire, ses actions, consulter en ligne ses actualités, connaitre ses prochains appels d’offres ou les résultats des précédents ? RDV sur son site web ! Contact : Agnès Ndiayé (agnes.ndiaye@inrae.fr). A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle du CEA, du CNRS, de l'INRAE, de l'Inria, de l'Inserm, de l’INCa, de la CPU et du Ministère de l’enseignement supérieur, de la recherche et de l’innovation (MESRI), il est l’unique instrument de financement commun à l’ensemble des établissements en sciences du vivant, Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques, et accompagne les démarches qualité en vue de la structuration et certification des plateformes.

|
Scooped by
Life Sciences UPSaclay
June 27, 2021 1:09 PM
|
L’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) compte à ce jour 11 plateformes dont PLAIMMO (PLAteforme d'IMmuno-MOnitorage). Son personnel est votre disposition, équipe académique comme industrielle, pour vous aider à la mise au point de techniques utilisables en cytométrie en flux. Parmi ses offres, PLAIMMO propose une activité de tri cellulaire, et opère à ce titre avec un FACAriaTM (BD Biosciences). Ce trieur, comme d’autres équipements concurrents, permet d’isoler et de récupérer une ou plusieurs sous populations cellulaires selon des critères de taille, de granulosité et de fluorescence indiquant, par exemple la présence de protéine d'intérêt, d'une transformation génétique, ou bien les phases du cycle cellulaire. Ce trieur fonctionne en déviant électrostatiquement des gouttelettes contenant les cellules d'intérêt, et ils sont connus pour créer des aérosols du fait du passage de liquide à une forte pression et haute vitesse dans un orifice de faible diamètre (70 à 100 µm). De plus très souvent, le statut sanitaire des cellules triées n’est pas toujours établi et la classification d'un pathogène pour l’être humain peut être modifié du fait de la génération de ces aérosols. Cela entraine dans l'environnement, la dispersion de particules potentiellement infectieuses. De ce fait, il est fortement déconseillé d'effectuer des tris de cellules infectées, humaines ou transfectées. PLAIMMO, naturellement soucieuse de la sécurité et de l’hygiène des utilisateurs et personnels, mais souhaitant également proposer une offre de services étendue à ses usagers, s’est donc récemment équipé d’une hotte de sécurité microbiologique de niveau 2 : une H-Box 17-11® (Noroit). Subventionné par un co-financement du Département Science de la Vie de l'Université Paris-Saclay (AAP « Petits équipements de laboratoire » (PEL-SDV) 2019), installé en juin dernier et validé en fin d’année dernière, cet équipement rend le trieur cellulaire à nouveau opérationnel. Cet équipement est donc à votre disposition pour vos tris sensibles y compris ceux de cellules humaines ou infectées. La plateforme de cytométrie en flux (PLAIMMO) de l'Unité Mixte de Service – Ingénierie et Plateformes au Service de l’Innovation Thérapeutique (UMS-IPSIT) située sur le site de Clamart offre régulièrement ses services aux équipes de recherche académiques du territoire Paris-Saclay ainsi qu'aux industriels. Le personnel de la plateforme est à votre disposition pour vous aider à pour la réalisation de projets de recherche fondamentale, préclinique sur des modèles expérimentaux ainsi que pour des protocoles de recherche clinique. Son personnel est aussi à votre service pour la mise au point de nouvelles techniques utilisant la cytométrie en flux. Les équipements de cytométrie en flux de la plateforme permettent le phénotypage des cellules par la détection de molécules membranaires et intracellulaires (biomarqueurs) mais aussi des études fonctionnelles tel que la détection de phosphorylation des protéines, la prolifération cellulaire, la quantification de cytokines ou chimiokines excrétées ou la détection d'ARN. Enfin, des tris cellulaires à haut débit sont aussi proposés par la plateforme. La plateforme est également équipée pour mesurer de l'expression de gènes grâce à la PCR quantitative en temps réel. Nos activités qui peuvent être en relations avec celle d'autre plateforme, permettent l'identification de nouveaux biomarqueurs qui peuvent être des cibles thérapeutiques. Contact : Marie-Laure Aknin (marie-laure.aknin@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI En savoir plus sur la plateforme de cytométrie en flux PLAIMMO ? Lisez à nouveau leurs précédents FOCUS PLATEFORME : juillet 2020, avril 2021 !

|
Scooped by
Life Sciences UPSaclay
June 10, 2021 5:01 PM
|
Une image vaut mille mots. En biologie, les images générées par les outils d’acquisition modernes contiennent une quantité énorme d’informations : c’est ce que l’on appelle l’imagerie à haut contenu. Le traitement de ces données représente un défi considérable pour les chercheurs, de surcroît lorsque le nombre d’échantillons à analyser est très important. Il est donc indispensable de trouver des solutions informatiques pour parvenir à traiter le nombre croissant de données générées. Cet écueil de traitement de données d’images ne se pose pas uniquement chez les organismes de grande taille. Les organismes modèles de laboratoire, pourtant de petite taille – notamment le poisson-zèbre à TEFOR Paris-Saclay (UMS CNRS 2010 / UMS INRAE 1451 / Université Paris-Saclay) - génèrent eux aussi une quantité importante de données, en particulier lorsqu’ils sont imagés d’un tenant (organisme entier). Un poisson qui vous veut du bien. Le poisson-zèbre s’est imposé comme un modèle vertébré de première importance. Plus de 80% des gènes associés à des maladies humaines trouvent leur équivalent chez ce modèle. Les champs d’application en recherche sont variés : neurosciences, toxicologie, biologie du développement, cardiologie, infectiologie, ou encore oncologie. Le développement du poisson-zèbre très rapide, et sa petite taille (quatre à cinq centimètres à l’âge adulte) sont compatibles avec une imagerie à haute résolution sur organe ou organisme entiers et en peu de temps. Zebrafish + Brain + Inspector = ZBI. En collaboration avec l’ESIEE, CentraleSupélec et la société L’Oréal, TEFOR Core Facility (TCF) a développé ZeBraInspector (ZBI), un outil logiciel de visualisation et d’analyse d’images 3D de larves de poisson-zèbre. Grâce à un marqueur fluorescent des lipides, ce logiciel corrige automatiquement les écarts d’orientations entre les images et facilite ainsi la comparaison des détails morphologiques en parallèle sur plusieurs spécimens. Comme son nom l’indique, ZBI (ZeBraInspector) est particulièrement adapté aux recherches sur le cerveau du poisson-zèbre. Il offre également des algorithmes de calcul du volume de la matière blanche du cerveau, et permet une analyse statistique quantitative des différences de morphologie cérébrale entre les échantillons. En savoir plus ? S. Lempereur et al., BioRxiv 2020 Enfin, ZBI est … GRATUIT ! Téléchargez le via le lien ci-contre : https://tefor.net/portfolio/zebrainspector. Contact : Jean-Stéphane Joly (jean-stephane.joly@cnrs.fr) et Pierre Affaticati (pierre.affaticati@cnrs.fr) Plug In Labs Université Paris-Saclay : cliquer ICI TEFOR Core Facility (TCF). TEFOR Core Facility (TCF) est au coeur de l’unité d’appui à la recherche TEFOR Paris-Saclay. L’espèce modèle principale d’intérêt du TCF est le poisson-zèbre (Danio rerio). Le TCF développe également des protocoles adaptés à d’autres organismes modèles aquatiques. Grâce à la complémentarité des équipes composant la plateforme, le TCF propose à la communauté scientifique des services intégrés : (1) L’équipe TPS-EDIT propose des prestations de création de modèles grâce à l’édition du génome via le système CRISPR-Cas9 (KO, KI) ; (2) L’équipe TPS-PHENO propose des prestations de phénotypage par imagerie 3D à haut débit et haute résolution sur des échantillons fluorescents ; (3) L’équipe TPS-INFO développe des outils permettant la visualisation, l’analyse, le traitement et l’archivage des données images obtenues par microscopie confocale 3D. Dans le cadre des actions de formations menées par la plateforme TEFOR Paris-Saclay, le TCF anime des formations aux techniques de clarification appliquées à l’imagerie 3D.

|
Scooped by
Life Sciences UPSaclay
May 23, 2021 5:28 PM
|
Depuis sa création, le Genoscope-CNS (Centre National de Séquençage) participe à des projets collaboratifs à fort impact scientifique, en fournissant à la communauté scientifique toute l’expertise et les capacités de production et d’analyse de données nécessaires à ces projets. En 2003, il a contribué au projet Human Genome, en publiant la séquence complète du chromosome 14 (Heilig et al., Nature 2003). Depuis, la plateforme de séquençage de Genoscope, située au cœur du biocluster Genopole, a réalisé le séquençage de nombreuses plantes (vigne, riz, blé…), d’animaux (tétraodon, anophèle…), de champignons (truffe, champignons pathogènes du colza et de la vigne) et de plus d’une centaine de procaryotes. Pedro Oliveira, responsable de la plateforme nous indique que « plus de 50 projets d’envergure ont été réalisés, y compris des études agronomiques, océaniques et terrestres sur l’environnement. Parmi eux, le projet Tara Océans qui vise à explorer par une approche métagénomique le plus grand écosystème planétaire que représente le plancton océanique [ndlr Planes et al., PLoS Biology 2019]. Encore très peu étudiés, ces écosystèmes contiennent de 10 à 100 milliards d’organismes par litre d’eau de mer. Leur impact sur les cycles géochimiques globaux et le climat est extrêmement important puisqu’ils absorbent la moitié de notre production en dioxyde de carbone et dégagent 50 % de l’oxygène de la planète » avant d’ajouter « les membres de la plateforme et moi-même, sommes très fiers de contribuer à ce passionnant projet ! ». L’équipe de la plateforme a mis au point des procédures pour traiter des milliers d’échantillons de protocoles méta-omiques pour des projets portant sur le microbiome. Pedro nous confie « qu’une équipe de chercheurs se consacre au développement de nouvelles technologies, y compris l’essai de nouvelles méthodes de séquençage et la bio-informatique associée à des projets environnementaux ». Grâce à sa collaboration avec le Très Grand Centre de Calculs du CEA, situé lui-aussi en Essonne, le Genoscope-CNS a permis de traiter de très grands ensembles de données en utilisant l’informatique haute performance de pointe. Ainsi, depuis sa création, le Genoscope-CNS a contribué à plus de 500 publications et ceci grâce à sa plateforme de séquençage. Pour obtenir des renseignements sur les activités de la plateforme, n’hésitez pas, contactez Pedro OLIVEIRA ! Contact : Pedro OLIVEIRA (pcoutool@genoscope.cns.fr) Plug In Labs Université Paris-Saclay : cliquer ICI. GENOPOLE / Genoscope / Plateforme de Séquençage. Le Genoscope s’est orienté vers l’étude de la biodiversité, à travers le séquençage de novo de grands génomes complets, de métagénomes complexes et plus généralement de collections d’ADN nécessitant une approche de séquençage à très grande échelle. L’équipe du laboratoire de séquençage s’implique dans la prise en main des projets à partir de la réception des échantillons jusqu’à la validation des séquences brutes. Le séquençage est assuré par des séquenceurs très haut débit de deuxième génération. Ces machines permettent d’atteindre un débit autorisant le séquençage de génomes entiers dans un intervalle de temps limité. A propos de Genopole. Biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 77 entreprises de biotechnologies, 18 laboratoires de recherche, 26 plateformes technologiques, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole, dirigé par Gilles Lasserre, est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
May 9, 2021 9:43 AM
|
Le syndrome de l'intestin irritable (SII) est le trouble gastro-intestinal le plus courant et est associé à une réduction importante de la qualité de vie liée à la santé. Le microbiote intestinal est associé à la gravité des symptômes du SII, mais on ignore encore comment la combinaison du régime alimentaire et du microbiote intestinal affecte les symptômes du SII (J. Tap et al., Gastroenterology 2017). Une nouvelle approche méthodologique combinant les données d'un journal alimentaire de 4 jours avec celles du microbiote intestinal obtenues par séquençage métagénomique pour des individus atteints de ce syndrome et des contrôles (individus sains) vient d’être publiée (J. Tap et al., Microbiome 2021). Cette étude est le fruit d’une collaboration entre Danone Nutricia Research (Palaiseau), l'Université de Göteborg (Suède) et la plateforme MetaGenoPolis (INRAE, Jouy-en-Josas). Retour sur cette étude… Les individus souffrant d'un SII sévère se caractérisent par une consommation plus importante d'aliments de moindre qualité au cours de leurs principaux repas (mesurée par un indice utilisé pour le calcul du Nutri-Score). Les niveaux de gaz exhalés (dihydrogène et méthane) et la gravité des symptômes peuvent être prédits à partir des données métagénomiques et alimentaires. Cette étude rapporte également que la sévérité du SII est associée à une altération dans la composition des hydrogénases du microbiote intestinal en corrélation avec les enzymes du microbiote impliquées dans le métabolisme des glucides associés à une alimentation carnée. Enfin, cette étude fournit une résolution sans précédent des interactions régime alimentaire-microbiote-symptôme et oriente en définitive vers de nouvelles études interventionnelles visant à identifier des recommandations nutritionnelles basées sur le microbiote intestinal pour la gestion des symptômes gastro-intestinaux. Contact : Julien Tap (julien.tap@danone.com) et Alexandre Cavezza, Florence Haimet (contact@mgps.eu) Plug In Labs Université Paris-Saclay : cliquer ICI Lire à nouveau les précédents FOCUS PLATEFORME de MetaGenoPolis ? Celui de mai 2020 et celui de septembre 2020 ! MetaGenoPolis (MGP) est un centre INRAE expert en recherche sur le microbiome intestinal appliquée à la santé et à la nutrition de l’homme et de l’animal. En collaboration avec les industries, les universités et les cliniques, MGP conçoit et met en œuvre des projets adaptés aux besoins de ses partenaires. Certifiés ISO 9001, les protocoles et procédures mis en œuvre sont constamment maintenus à la pointe de la technologie. MGP propose de la métagénomique quantitative et fonctionnelle pour explorer le lien entre le microbiome, nutrition et santé. MGP offre des services d’analyse du microbiome de bout en bout, y compris des recommandations personnalisées sur la collecte d’échantillons, la mise en banque d’échantillons, l’extraction d’ADN, la métagénomique quantitative et fonctionnelle, les grandes installations de stockage de données et de calcul, la bioinformatique, l’analyse statistique et l’interprétation des données. Une des ambitions de MGP est de constituer via un projet de science citoyenne une base de données publique regroupant les microbiomes de 100 000 individus français dont un des objectifs sera de mieux comprendre l’hétérogénéité des microbiomes intestinaux de français sains. A propos de Danone Nutricia Research. Entreprise multi-locale parmi les leaders de l’alimentation, Danone se développe sur des catégories tournées vers la santé et en forte croissance à travers ses 3 métiers : les Produits laitiers et d’origine végétale, les Eaux et la Nutrition spécialisée. Avec un cadre d’action unique, ‘One Planet. One Health’, qui affirme que la santé des personnes et de la planète sont intimement liées, Danone vise à inspirer des pratiques alimentaires plus saines et plus durables. Pour accélérer cette révolution de l’alimentation et créer de la valeur durable et rentable sur le long terme pour toutes ses parties prenantes, Danone a défini neuf Objectifs pour 2030 et est devenue la première société cotée à adopter le statut d'Entreprise à Mission en France. La R&I est au cœur de la mission du Groupe : « Apporter la santé par l’alimentation au plus grand nombre ». Danone Nutricia Research associe la recherche & l’innovation de Danone, avec 1800 chercheurs et développeurs, répartis dans 7 centres de recherche et dans 55 filiales locales. Grâce à son savoir scientifique et technologique et à sa compréhension des consommateurs, Danone Nutricia Research fournit des produits alimentaires bons et sains.
|



 Your new post is loading...
Your new post is loading...