Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
February 8, 4:20 PM
|
Aujourd’hui, un FOCUS PLATEFORME en lien avec le SESAME 2025, dont les résultats globaux pour Paris-Saclay vous ont été présentées le 1er décembre dernier. Le projet BioALTO vise à développer une plateforme expérimentale dédiée à la recherche préclinique de pointe dans le domaine de l’hadronthérapie et de la radiobiologie. Le nouvel ensemble expérimental sera développé sur l’installation ALTO (Accélérateur Linéaire et Tandem à Orsay) au Laboratoire de Physique des 2 Infinis Irène Joliot-Curie (IJCLab), pouvant fournir un ensemble d’ions d’intérêts en hadronthérapie avec notamment des protons, des ions héliums, carbones ou encore oxygènes. Cette large gamme d’ions permet à la plateforme BioALTO de se distinguer des autres plateformes nationales d’irradiation et d’étudier expérimentalement l’efficacité biologique relative pour tous les ions d’intérêts, sur une unique installation. La plateforme BioALTO va répondre à la demande croissante de faisceaux d’ions par la vaste communauté engagée dans la recherche de thérapies innovantes et dans la R&D pour le traitement du cancer en Île-de-France. Le projet porté par le laboratoire IJCLab, regroupant les pôles Physique Santé et Physique des accélérateurs va bénéficier de l’implication forte des équipes de Gustave Roussy dont l’expertise en oncologie, physique médicale et radiobiologie est reconnue, et de collaborations internationales avec, notamment, l’Institut de Microélectronique de Barcelone en Espagne. Le projet BioALTO est lauréat de l’appel à projets régional SESAME 2025 avec une subvention de la région Île-de-France à hauteur de 197 k€ qui va permettre l’optimisation d’une ligne de faisceau d’accélérateur et l’équipement d’une station d’irradiation pour mener des expériences de radiobiologie. Ce développement va être complété par l’installation d’une salle de culture cellulaire à proximité de la ligne d’irradiation pour la préparation, le stockage et l’analyse des échantillons biologiques. Cet ensemble expérimental sera unique en France, grâce aux nouveaux équipements qui seront installés et à la variété de faisceaux d’ions et d’énergies disponibles à ALTO, actuellement absents des autres plateformes d’irradiation. -> Contact : Amélia Maia Leite , Quentin Mouchard (bioalto@ijclab.in2p3.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Accélérateur Linéaire et Tandem à Orsay (ALTO). L'installation ALTO est principalement constituée de deux accélérateurs : une machine électrostatique, un tandem MP de 15MV et un accélérateur linéaire d'électrons de 50 MeV. ALTO offre ainsi la possibilité de produire, au même endroit, des faisceaux d'agrégats pour la physique interdisciplinaire, des faisceaux stables et radioactifs pour l'astrophysique et la physique nucléaire, ainsi que des faisceaux de neutrons mono énergétiques de quelques MeV. L'installation abrite aussi deux laboratoires de R&D et de fabrication de cibles de carbure d'Uranium (UCx) et de couches minces. Elle dispose d'une plateforme lasers, de séparateurs d'isotope en ligne et hors ligne, de banc de tests pour les sources stables, et de dix lignes expérimentales. Les faisceaux fournis sont dédiés à un large éventail de programmes de physique allant de la structure nucléaire à la physique atomique, la physique des clusters, la biologie, la nanotechnologie et des prestations industrielles. A propos de la plateforme BioALTO… La plateforme BioALTO en développement au sein de la plateforme de recherche ALTO (Accélérateur Linéaire et Tandem à Orsay) au Laboratoire de Physique des 2 Infinis Irène Joliot-Curie (IJCLab), vise à offrir au domaine de la radiobiologie un outil nouveau et unique, exploitant des faisceaux de protons et d'ions plus lourds de l'ordre de dizaines de MeV pour la recherche en hadronthérapie expérimentale à différents débits de dose. La ligne d’irradiation est actuellement en cours d’adaptation et d’amélioration permettant une interopérabilité avec l’installation ALTO et de garantir les conditions d’irradiations nécessaires pour les études radiobiologiques. En savoir plus sur BioALTO ? Amélia Maia Leite, Quentin Mouchard, Philippe Lanièce. BioALTO platform. Ecole Joliot Curie : Radiations for health, Sep 2024, Saint-Pierre d'Oléron, France. ⟨hal-04700410⟩.

|
Scooped by
Life Sciences UPSaclay
January 25, 11:51 AM
|
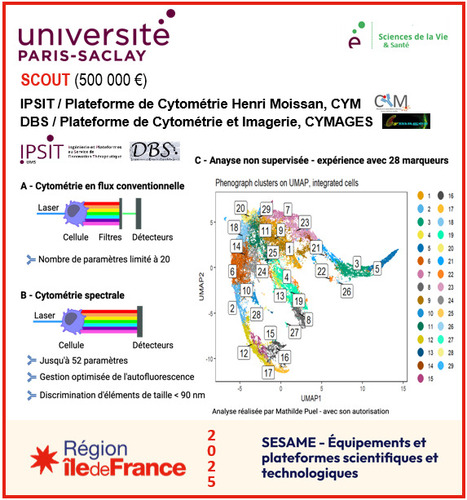
Aujourd’hui, un FOCUS PLATEFORME en lien avec le SESAME 2025, dont les résultats globaux pour Paris-Saclay vous ont été présentées le 1er décembre dernier. Les plateformes de cytométrie en flux et immuno-monitorage CYM (UMS Ingénierie et Plateformes au service de l’Innovation Thérapeutique (IPSIT), Faculté de Pharmacie Paris-Saclay) et CYMAGES (Département de Biotechnologies de la Santé, UFR Simone Veil - Santé à Montigny-le-Bretonneux, Université de Versailles Saint-Quentin-en-Yvelines) sont au service des projets ambitieux en recherche fondamentale, translationnelle et clinique des communautés de chercheurs du plateau de Saclay et de l’UFR Simone Veil – Santé. Afin de répondre aux besoins de leurs utilisateurs en termes d’accroissement du nombre de paramètres pour l’analyse et le tri d’éléments uniques, de gestion de l’auto-fluorescence cellulaire et d’accès à des éléments de très petite taille, CYM et CYMAGES ont conçu le projet SCOUT (Spectral Cytometry for Optimal characterization, Unsupervised analysis and sorting of single elemenTs), pour se doter, respectivement, d’un cytomètre trieur et d’un cytomètre analyseur à la pointe de la technologie spectrale. Ces cytomètres, équipés de 5 lasers, permettront de discriminer plus de 35 marqueurs fluorescents de façon simultanée, en dépit de l'autofluorescence. Ils assureront ainsi la caractérisation fine et le tri de cellules eucaryotes comme procaryotes, tout en rendant possible l’analyse de vésicules extracellulaires et de nanoparticules. De plus, la possibilité d’analyser le spectre de fluorescence complet facilitera l’utilisation de sondes fluorescentes non conventionnelles. SCOUT vient d’être lauréat de l’appel à projet régional SESAME 2025. La région Île-de-France apporte ainsi à ce projet une subvention de 500 k€ (50 % du montant des équipements souhaités) qui, ajoutée à des co-financements dont plusieurs doivent encore être obtenus, devrait permettre l’acquisition de ces instruments pour un budget total de 1 000 k€. Le trieur spectral sera placé dans un laboratoire de niveau 2 sur la plateforme CYM et l’analyseur spectral sur la plateforme CYMAGES, et les équipes de CYM et CYMAGES espèrent vous accueillir prochainement pour mettre en pleine lumière les caractéristiques de vos échantillons les plus précieux ! -> Contact : Géraldine SCHLECHT-LOUF (geraldine.schlecht-louf@universite-paris-saclay.fr), Marie-Laure AKNIN (marie-laure.aknin@inserm.fr), Simon Glatigny (simon.glatigny@uvsq.fr) Plug In Labs Université Paris-Saclay : CYM, cliquer ici, CYMAGES, cliquer ICI IPSIT / Plateforme de Cytométrie H.Moissan (CYM). La plateforme de cytométrie en flux (CYM) de l'Unité Mixte de Service, Ingénierie et Plateformes au Service de l'Innovation Thérapeutique (UMS-IPSIT), située au rez-de-chaussée du bâtiment Recherche Henri Moissan (HM1) offre ses services aux équipes de recherche académiques du territoire Paris-Saclay ainsi qu'aux industriels. Le personnel de la plateforme est à votre disposition pour vous aider à réaliser des projets de recherche fondamentale et préclinique, ainsi que des protocoles de recherche clinique. Son personnel est aussi à votre service pour la mise au point de nouvelles techniques utilisant la cytométrie en flux. Les équipements de cytométrie en flux de la plateforme permettent le phénotypage des cellules par la détection de molécules membranaires et intracellulaires (biomarqueurs) mais aussi des études fonctionnelles telles que la détection de phosphorylation des protéines, la prolifération cellulaire, la quantification de cytokines excrétées ou la détection d'ARN. Enfin, des tris cellulaires à haut débit sont également proposés par la plateforme (contact pour la cytométrie : marie-laure.aknin@universite-paris-saclay.fr). En parallèle, CYM met à votre disposition un appareil de mesure de haute technologie, le QuickPlex® SQ 120 (Meso Scale Discovery, MSD) permettant l’analyse multiplex de biomarqueurs par électrochemiluminescence (contact pour le MSD : sophie.viel@universite-paris-saclay.fr). Les activités de CYM peuvent être réalisées en collaboration avec celles d'autres plateformes de l’UMS-IPSIT, favorisant les analyses transcriptomiques sur cellules triées ou l'identification de nouveaux biomarqueurs. Département Biotechnologie Santé / Plateforme d'imagerie et cytométrie (CYMAGES). La plateforme d'imagerie et cytométrie CYMAGES, créée en septembre 2012 avec le soutien de la Région Ile-de-France, du Conseil Général des Yvelines, et de la Communauté d'agglomération de communes de Saint-Quentin-en-Yvelines, a pour objectif d'offrir à la communauté scientifique académique et industrielle des équipements d'imagerie et de cytométrie à la pointe de la technologie, ainsi que conseil et assistance. La plateforme CYMAGES propose un parc technologie composé de sept équipements de pointe en Imagerie et cytométrie photonique : Microscope confocal Leica SP8X (laser blanc) / Microscope confocal Leica SPE / Microscope plein champs HCS Olympus ScanR / Cyto-imageur Cytek Imagestream / Cytomètre analyseur Becton Dickinson Fortessa (18 paramètres) / Trieur cellulaire Becton Dickinson Aria III (13 Paramètres), lecteur multiplexe (MSD). La plateforme offre également des prestations d’analyse de données en imagerie et cytométrie.

|
Scooped by
Life Sciences UPSaclay
January 11, 11:16 AM
|
6 mois déjà… En effet, le 30 juin 2025, s’est déroulée l’inauguration officielle de la plateforme de radiochimie de MIRCen (CEA Centre de Fontenay-aux-Roses, DRF, Institut de biologie François Jacob), spécialisée dans la recherche et développement de radiotraceurs à visée préclinique dans le contexte de maladies neurodégénératives (mécanismes, thérapies, imageries). Plus de 120 invités se sont réunis au CEA de Fontenay-aux-Roses sur le site historique de la première pile atomique française Zoé. Parmi ces invités se trouvaient plusieurs partenaires de NeurATRIS, la composante française de l'infrastructure européenne de recherche translationnelle EATRIS-ERIC, spécialisée en neurosciences et en neurologie (hôpitaux Henri Mondor, Bicêtre, Robert Debré ; INRAE, ICM), plusieurs partenaires de PASREL-Imagerie, structure qui fédère les principaux acteurs de l’imagerie en Ile de France, plusieurs industriels (Lilly, Roche, Sanofi, Servier, UCB, Xing Imaging), des spécialistes de la radiochimie des universités françaises du territoire Paris-Saclay et de Lyon, mais aussi universités européennes (Bruxelles, Amsterdam). Doté d’un cyclotron biomédical, d’enceintes blindées et d’automates de radiosynthèse versatiles, la plateforme est capable d’utiliser des méthodologies de marquage isotopique très variées, maximisant ainsi les probabilités de succès du marquage. Elle a pour mission de développer et de fournir des radiotraceurs de nouvelle génération marqués au carbone-11 (demi-vie 20,4 min) ou au fluor-18 (demi-vie 109,8 min) pour les activités de recherche et développement du CEA et celles de ses partenaires, des acteurs académiques et des industriels. La plateforme répond également à des demandes de prestation et de collaboration ponctuelles. En étudiant le comportement in vivo de nos traceurs, les interactions ligand/cible, l’équipe TEP (MIRCEN / imagerie TEP, IRM et Optique) fournit des informations utiles pour le développement de nouveaux outils de diagnostic précoce des maladies neurodégénératives (Parkinson, Huntington, Alzheimer). Quand le marquage isotopique est réalisé sur une molécule candidat-médicament, nos études permettent d’accélérer le processus de développement de nouveaux médicaments : à titre d’exemples, l’étude de la biodistribution in vivo de la molécule permet d’évaluer sa spécificité, et de prédire d’éventuels effets secondaires ; la détermination de la pharmacodynamie et de la pharmacocinétique in vivo du traceur participe à estimer la dose efficace et la posologie du futur médicament. E> Contact : yann.bramoulle@cea.fr Plug In Labs Université Paris-Saclay : cliquer ICI MIRCEN / Radiochimie. La plateforme de radiochimie de MIRCEN produit des radioisotopes émetteurs de positons, le carbone-11 et le fluor-18, puis les incorpore dans des molécules pour en faire des radiotraceurs destinés à l’imagerie préclinique par Tomographie par Emission de Positons (TEP). Ces radiotraceurs peuvent également être distribués à l’extérieur du laboratoire.

|
Scooped by
Life Sciences UPSaclay
December 21, 2025 1:21 PM
|
L’unité mixte de service UMS-IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique, Faculté de Pharmacie, Bâtiment Henri Moissan, Orsay) s’est engagée depuis plusieurs années dans une démarche qualité ambitieuse visant à optimiser la performance de ses plateformes technologiques et à renforcer leur visibilité. Portée par deux référents qualité — Claudine Deloménie (responsable de la plateforme de transcriptomique ACTAGen) et Séverine Domenichini (responsable de la plateforme d’imagerie MIPSIT) — cette initiative structurante a bénéficié en 2020 d’un financement « Moyens de Recherche Mutualisés » (MRM) de 18 k€ accordé par l’Université Paris-Saclay. Ce soutien a permis d’intégrer à la démarche un accompagnement professionnel par un qualiticien expert. Grâce à une mobilisation collective, cette démarche a abouti à la formalisation d’un système de management de la qualité (SMQ) partagé, incluant des indicateurs annuels de performance et de suivi, et à l’identification de priorités stratégiques pour le développement des services de l’unité. Ces efforts ont porté leurs fruits : en 2024, le SMQ de l’UMS-IPSIT a passé avec succès un audit de pré-certification conduit par le Réseau Inserm Qualité. Ce résultat marque une étape clé vers l’objectif de certification ISO 9001, envisagée dès 2026. Ce succès collectif témoigne de l’engagement constant de l’équipe de l’IPSIT en faveur de l’excellence opérationnelle et de l’amélioration continue de l’UMS. -> Contacts : Valérie Domergue (valerie.domergue@universite-paris-saclay.fr) / Claudine.Delomenie (claudine.delomenie@universite-paris-saclay.fr) / Séverine Domenichini (severine.domenichini@universite-paris-saclay.fr) Plug-In-Labs Université Paris-Saclay : cliquer ICI A propos de l’IPSIT. L’IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes techniques, organisées en deux pôles technologiques (IMCELLF et OMICS) et quatre plateformes. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi ses FOCUS PLATEFORME décrivant toutes ses expertises !

|
Scooped by
Life Sciences UPSaclay
December 7, 2025 11:28 AM
|
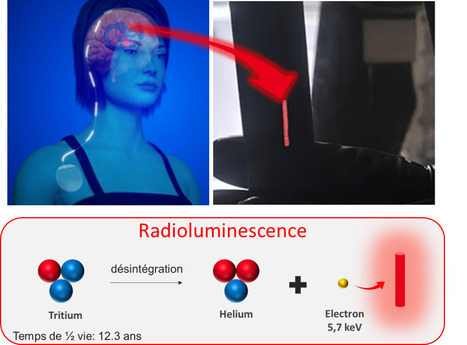
La radioluminescence ? Une R&D innovante aussi prise en charge par la plateforme de marquage isotopique du CEA (Service de Chimie Bioorganique et de Marquage (SCBM, Département Médicaments et technologies pour la Santé, Institut Joliot, CEA, Centre de Paris-Saclay). La radioluminescence, mais dans quel contexte ? La maladie de Parkinson est une pathologie neurodégénérative qui touche aujourd’hui plus de 8 millions de personnes dans le monde, avec un nombre de nouveaux cas en constante augmentation. En France, on dénombre environ 25 000 nouveaux cas chaque année. Les traitements actuellement disponibles permettent uniquement de soulager les symptômes, sans agir sur les causes profondes de la maladie. Dans ce contexte, la photobiomodulation représente un espoir prometteur pour le développement de traitements véritablement curatifs. Depuis plusieurs années, un projet de recherche innovant mené par les chercheurs de Clinatec (CEA, Grenoble) a mis en évidence l’effet de l’illumination de la substance noire sur l’évolution de la maladie dans différents modèles animaux. Actuellement, un essai clinique est en cours chez l’être humain et plusieurs patients ont déjà été implantés. Le dispositif utilisé (voir illustration, en haut à gauche, représentation schématique de l’implant actuel), se compose d’une batterie implantée sous la clavicule, d’un boîtier optique placé dans la boîte crânienne et d’une fibre optique insérée dans les ventricules cérébraux. Bien que cette approche soit prometteuse, l’implantation de la batterie et du câble la reliant à la fibre optique est invasive et donc lésionnelle pour le patient. Ainsi, le développement d’un implant autonome capable d’émettre de la lumière rouge, apparaît hautement souhaitable Et quel lien avec la plateforme de marquage isotopique ? C’est dans cette perspective qu’un projet financé par le CEA a été récemment conduit, réunissant les expertises complémentaires des chercheurs et ingénieurs de trois laboratoires du CEA de Grenoble (Clinatec, LITEN et LETI) et du laboratoire de marquage isotopique de l’Institut JOLIOT (CEA Centre de Paris-Saclay) et plus particulièrement sa plateforme de marquage isotopique. Ce projet a permis de démontrer qu’il était possible d’exploiter la radioluminescence pour concevoir de tels dispositifs (voir illustration, partie basse, principe de la radioluminescence). Les résultats obtenus ont montré que différents types de luminophores, dispersés dans des aérogels de silice ou de cellulose, peuvent être excités par les électrons issus de la désintégration du tritium (isotope radioactif de l’hydrogène, émetteur beta-moins), ouvrant ainsi la voie à la conception de nouveaux implants répondant aux exigences de compacité, d’autonomie et de puissance optique (voir illustration, en haut à droite, photographie d’un capillaire radioluminescent conçu sur la plateforme). De plus, toujours dans le cadre de ce projet, un dispositif expérimental unique a été développé au sein de la plateforme de marquage isotopique. Ce banc de mesure permet de mesurer en temps réel l’intensité lumineuse émise par un objet radioluminescent placé dans une atmosphère contrôlée. Ces travaux préliminaires très prometteurs qui ont fait l’objet d’un dépôt de brevet (FR 2113125) et d’une publication récente (Muñoz Velasco et al, 2025), seront approfondis dans le cadre du projet ANR Radiolight, dont les objectifs sont 1) d’optimiser les performances de ces implants tout en réduisant la quantité de tritium utilisée ; 2) d’étudier les effets du vieillissement sur ces matériaux ; 3) de développer de nouveaux concepts visant à générer des matériaux radioluminescents purement organique. -> Contact : Grégory Pieters (gregory. pieters@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI MTS / Plateforme de marquage isotopique. Cette plateforme possède une expertise (unique sur Paris-Saclay) en synthèse de composés marqués avec des isotopes stables (2H / deutérium et 13C / carbone-13) et par des isotopes radioactifs de type bêta (3H / tritium, 14C / carbone-14 et 125I / iode-125). Forte de son expertise dans la préparation (synthèse, contrôle de qualité) et formulation de molécules marquées, elle assure régulièrement des prestations et collaborations, académiques comme industrielles, dans le domaine du (radio)marquage moléculaire. Elle offre également à la demande, son expertise et environnement unique de travail (laboratoires « chauds », équipements dédiés) pour l’analyse et la caractérisation d’échantillons radioactifs : mesure de puretés chimique et radiochimique par HPLC, détermination d'enrichissements isotopiques et d'activités spécifiques par SM, analyse et détermination structurale par RMN liquide comme solide, mesure d’activités radioactives par comptage à scintillation. Enfin, la plateforme propose des solutions pour le traitement de déchets liquides radioactifs, en particulier ceux contenant du carbone-14 et du tritium. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
November 23, 2025 12:07 PM
|
Cette semaine, un FOCUS PLATEFORME dédié aux activités du SOERE-PRO, l’Observatoire de recherche en environnement rattaché à ECOSYS (UMR INRAE / AgroParisTech) ! Cet observatoire, unique, a pour objectif d’évaluer les effets agronomiques et les potentiels risques du retour au sol de divers produits résiduaires organiques (PRO) : effluents d’élevage, composts de déchets organiques urbains, digestats de méthanisation, etc. Créé au début des années 2000, il compte actuellement 8 sites expérimentaux. Depuis 2014, les sites QualiAgro, PROspective, EFELE, La Réunion et son système d’information sont intégrés à l’infrastructure de Recherche AnaEE-France. En 2018, les sites QualiAgro, PROspective et EFELE ont été labellisés en tant qu'Infrastructure Scientifique Collective INRAE (renouvellement en cours en 2025). L’originalité du SOERE-PRO tient dans la mise en place d’expérimentations de longue durée au champ dans différents contextes pédoclimatiques où l'évolution de la qualité des sols, des cultures, de l'eau et de l'air est mesurée après l'application répétée de PRO et la dynamique de ces évolutions est reliée aux caractéristiques des PRO appliqués (origine x traitement) ainsi qu’aux conditions pédoclimatiques. Le SOERE-PRO fournit des services d'Observation à la communauté scientifique : accès aux sites dans le cadre de programmes de recherche, fourniture d’échantillons (sol, PRO, plantes), fourniture de données (sol, PRO, plantes, eaux, GES). L’enjeu pour ce réseau de longue durée est de maintenir ses capacités d’expérimentation et de suivis (monitoring) ainsi que la motivation de son personnel scientifique et technique dans le temps. Pour cela, une coordination avec des responsables techniques et scientifiques a été mise en place dès le départ. Des temps d’échanges, de rencontres lors d’assemblées générales, de comités techniques, de groupes de travail notamment, rythment la vie du réseau. Des outils tels que des bulletins de veille presse et médias sur les PRO (publiés tous les deux mois), un site web dont la mise à jour complète a été réalisée de 2023 à 2025 (une version anglaise est en projet), un espace de partage institutionnel Nextcloud fournissent aux acteurs du réseau et aussi à la communauté scientifique, de précieuses informations scientifiques et techniques sur les dispositifs. N’hésitez pas à visiter le site web de cet observatoire pour mieux connaître ses objectifs, ses dispositifs, les suivis ainsi que les productions : https://valor-pro.hub.inrae.fr/. Aussi, si vous souhaitez en savoir plus et en quelques clics sur les travaux de recherche menés sur les dispositifs du SOERE PRO, n’hésitez pas… Michaud et al., Science of The Total Environment 2025 ; Jacquin et al., Agriculture, Ecosystems & Environment 2025; Didelot et al., Agriculture, Ecosystems & Environment 2025; Mora-Salguero et al., Front Microbiol 2025 ; Chen et al., Soil Use and Management 2024 -> Contact : Christophe Montagnier (christophe.montagnier@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI ECOSYS / SOERE PRO : Observatoire de recherche en environnement étudiant le recyclage agricole des Produits Résiduaires Organiques (PRO). Le SOERE PRO est un Observatoire de recherche en environnement composé de 8 dispositifs expérimentaux au champ de longue durée, étudiant les effets agronomiques et les potentiels risques du retour au sol de Produits Résiduaires Organiques (PRO). Cinq sites sont instrumentés pour suivre les PRO épandus, les compartiments de l'agrosystème, les émissions de gaz à effet de serre, le climat du sol, les eaux et la météo (Qualiagro en Ile de France, PRO'spective Grand-Est, EFELE en Bretagne, SOERE PRO - Réunion et SOERE PRO - Sénégal). Trois sites associés, sont peu ou pas instrumentés (La Bouzule Grand-Est, Couhins Nouvelle Aquitaine, MetaMetha Centre-Val de Loire). Le SOERE PRO a été labellisé en 2011 et 2015 par ALLENVI, 4 sites sont intégrés à ANAEE-France. En 2018, l'observatoire a été labellisé en tant qu'infrastructure scientifique collective (ISC) par INRAE. Les évolutions des agro-systèmes sont mesurées, à l'échelle de la parcelle agricole, au fur et à mesure d'apports de PRO issus d'activités urbaines et agricoles et de différentes filières de traitements. Les suivis de monitoring sont harmonisés entre les sites (PRO, sol, plante, eau, air) : éléments majeurs, rendements cultures, contaminants chimiques/organiques, pathogènes humains, reliquats azotés, propriétés physiques du sol. Les résultats du SOERE PRO sont intégrés dans des analyses multi-critères globales associant bénéfices et risques. Ils permettent de développer des modèles utilisés pour tester des scénarios d'insertion des PRO dans les systèmes de cultures pour optimiser leur usage. Les travaux contribuent également à l'accueil de nombreux projets de recherche (27 projets en cours en 2025) ainsi qu'à la formation d'étudiants (masters, doctorats).

|
Scooped by
Life Sciences UPSaclay
November 7, 2025 11:35 AM
|
Située au cœur du biocluster Genopole, la biobanque du Centre d'Étude des Cellules Souches (CECS) I-Stem est une plateforme essentielle pour la recherche basée sur l’utilisation des cellules souches humaines. Spécialisée dans la cryoconservation et la distribution de lignées cellulaires humaines, elle offre aux chercheurs un accès privilégié à une collection unique de cellules souches pluripotentes induites (CSPi) et de cellules souches embryonnaires humaines (CSEh), ainsi qu'à leurs dérivés. En intégrant StemCARE, une plateforme technologique labellisée IBISA, la Biobanque du CECS I-Stem bénéficie d'un cadre structurant favorisant l'innovation scientifique et le développement de nouvelles thérapies. La biobanque du CECS I-Stem met à disposition des ressources cellulaires de haute qualité pour les équipes académiques et industrielles du secteur des biotechnologies. Alexandra Benchoua, Directrice de recherche au CECS I-Stem, souligne : « Notre objectif est d'offrir aux chercheurs et industriels un accès à des lignées cellulaires répondant aux normes les plus exigeantes. Cette ressource unique contribue activement aux avancées en médecine personnalisée et de précision. » Les missions principales de la biobanque incluent : i) la collecte et conservation des échantillons : stockage sécurisé et conforme aux réglementations en vigueur ; ii) la préparation et distribution des lignées cellulaires : gestion rigoureuse assurant une traçabilité optimale ; iii) la garantie de qualité et conformité : procédures validées par le service Assurance Qualité d'I-Stem ; et iv) Une accessibilité et offre de collaboration : ouverture aux chercheurs scientifiques et industriels pour encourager les avancées scientifiques. Un levier pour le développement de nouvelles thérapies. Grâce à ses collections cellulaires et à son expertise en culture et conservation de cellules souches, la biobanque facilite le développement de nouvelles approches dans plusieurs domaines. Elle contribue à la modélisation des pathologies pour mieux comprendre les mécanismes des maladies génétiques. Elle joue un rôle clé dans le criblage pharmacologique afin d'identifier de nouvelles molécules thérapeutiques. Enfin, elle participe à l'optimisation des protocoles de transplantation cellulaire dans le cadre de la thérapie cellulaire. Un pilier central de StemCARE. En tant que composante essentielle de StemCARE, la biobanque joue un rôle clé dans l'écosystème. Sa capacité à fournir des ressources cellulaires de haute qualité, combinées à l'expertise scientifique d'I-Stem et au soutien technologique de StemCARE, permet d'accélérer la mise au point de nouvelles stratégies thérapeutiques. La biobanque est ouverte aux collaborations nationales et internationales pour les projets de recherche, de développement thérapeutique et d’innovation biomédicale. -> Contact : Julien Picot (julien.picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ICI A propos de Genopole / I-Stem / Plateforme Biobanque : Les cellules souches sont des ressources incontournables pour la recherche et développement. La Biobanque de cellules du CECS/I-Stem permet de conserver et de mettre à disposition des équipes de recherche le matériel biologique indispensable pour l’étude des maladies génétiques et le développement de thérapies cellulaires. La Biobanque du CECS / I-Stem conserve une large collection de lignées cellulaires humaines, notamment des lignées de cellules souches embryonnaires (CSEh), des lignées de cellules induites à la pluripotence (CSPi) ainsi que leurs dérivés. L’ensemble des lignées est en conservation miroir sur deux sites pour plus de sécurité. Après réception des échantillons initiaux fournis par les équipes d’I-Stem, des collaborateurs externes ou des partenaires industriels, les cellules sont cultivées et qualifiées en collaboration avec la Plateforme de Biologie cellulaire et la Plateforme de Bioproduction. Du fait de son organisation et de la gestion mise en place, la Plateforme Biobanque permet aux équipes de recherche de disposer à tout moment de nombreux modèles cellulaires de maladies génétiques ainsi que de cellules saines « contrôles » de haute qualité. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à la bioéconomie, Genopole réunit 78 entreprises, 19 laboratoires de recherche, 23 infrastructures, plateformes et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : favoriser l’émergence et la croissance de sociétés de biotechnologie, le transfert d’innovations vers le secteur industriel, le développement de la recherche et l’enseignement supérieur en sciences de la vie. Genopole est un Groupement d’intérêt public principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes technologiques labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, contactez Julien Picot (julien.picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
October 19, 2025 5:07 PM
|
Découvrez la technologie QuickPlex® SQ 120 sur la plateforme CYM : Sensibilité, polyvalence et performance. Depuis 2020 et grâce aux financements obtenus dans le cadre des dispositifs ERM – Équipements de recherche mutualisés (Université Paris-Saclay, 2019) et DIM – Domaines d’Intérêt Majeur (Région Île-de-France, 2020), la plateforme CYM a enrichi son offre technologique avec un équipement de pointe : le QuickPlex® SQ 120 (Meso Scale Discovery, MSD). Installé dans les locaux de l’UMS-IPSIT (bâtiment Henri Moissan - UFR Pharmacie, Orsay), cet appareil de haute technologie permet des analyses multiplexées sur échantillons biologiques, avec la capacité de quantifier simultanément jusqu’à 10 biomarqueurs (Hassler et al., PLOS Medicine 2020). Fondée sur le principe de l’ELISA, la détection repose sur une technologie brevetée par MSD : l’éléctrochimioluminescence™ ou ECLIA™ offrant : i) Une sensibilité exceptionnelle (jusqu’à l’ordre du fg/mL) ; ii) Une large gamme dynamique de 4-5 logs, et iii) Un large choix de biomarqueurs avec une flexibilité de créer son propre panel (custom). Cette technologie permet des dosages fiables dans des matrices variées, allant des plus classiques (sérum, plasma, surnageants de culture) aux plus complexes (sang total, homogénats cellulaires, liquide broncho-alvéolaire), en passant par des matrices plus rares (urines, larmes, LCR, Dry Spot ou buvard microvésicules). Le faible volume d’échantillon requis (entre 10 et 25 µL) est aussi un autre avantage non négligeable. C’est une technologie adaptée à des domaines de recherche de pointe. Rapide, flexible et générant peu de bruit de fond, le Meso QuickPlex® SQ 120 trouve des applications dans des champs variés tels que l’oncologie, l’inflammation, la neurodégénérescence, la toxicologie, le cardiovasculaire, l’immunogénécité. Notamment, cette technologie a été utilisée par deux équipes de l’UMR-S996 – Inflammation, Microbiome et Immunosurveillance, pour étudier les réponses immunitaires dans le cadre de projets innovants (Gallego et al., 2021; Nabhan et al., Eur J Pharm Sci, 2024). Il existe bien d’autres domaines d'applications. Par exemple, elle est recommandée par la FDA pour la détection des anticorps anti-médicaments (ADA) lors d’essais cliniques. Elle permet de limiter les interférences médicamenteuses, contrairement à d’autres approches (Morgan et al., Front. Immunol. 2019). L’UMR-S 996 a développé une expertise spécifique dans le dosage sur mesure des ADA. Ce lecteur est utilisable avec un large choix de kits proposés par la société MSD, chez l’animal ou chez l’homme. En savoir plus ?. La plateforme CYM vous propose, après discussion de votre projet, d’accéder à cette technologie à travers deux offres de service : i) Une prestation autonome : après conseils et formation, vous pouvez accéder en autonomie au lecteur pour analyser vos plaques, ou ii) Une prestation accompagnée : une ingénieure d’étude, Sophie Viel (IE Inserm), peut réaliser l’ensemble du processus (mise au point du test, lecture, analyse des résultats) à travers des projets collaboratifs ou des prestations de service. L’ensemble des prestations de la plateforme CYM s’inscrit dans une démarche qualité structurée (charte, fiches projet, suivi encadré). Vous avez des projets ? Venez les concrétiser avec l'aide de la plateforme CYM ! -> Contact : Sophie Viel (sophie.viel@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI CYM contribue régulièrement à des FOCUS PLATEFORME, n’hésitez-pas à les relire … IPSIT / Plateforme de Cytométrie H.Moissan (CYM). La plateforme de cytométrie en flux (CYM) de l'Unité Mixte de Service, Ingénierie et Plateformes au Service de l'Innovation Thérapeutique (UMS-IPSIT), située au rez-de-chaussée du bâtiment Recherche Henri Moissan (HM1) offre régulièrement ses services aux équipes de recherche académiques du territoire Paris-Saclay ainsi qu'aux industriels. Le personnel de la plateforme est à votre disposition pour vous aider à la réalisation de projets de recherche fondamentale, préclinique sur des modèles expérimentaux ainsi que pour des protocoles de recherche clinique. Son personnel est aussi à votre service pour la mise au point de nouvelles techniques utilisant la cytométrie en flux. Les équipements de cytométrie en flux de la plateforme permettent le phénotypage des cellules par la détection de molécules membranaires et intracellulaires (biomarqueurs) mais aussi des études fonctionnelles telles que la détection de phosphorylation des protéines, la prolifération cellulaire, la quantification de cytokines ou chimiokines excrétées ou la détection d'ARN. Enfin, des tris cellulaires à haut débit sont aussi proposés par la plateforme (contact cytométrie : marie-laure.aknin@universite-paris-saclay.fr). Et en parallèle, elle étend maintenant son offre de service en mettant à votre disposition un nouvel appareil de mesure de haute technologie, le QuickPlex® SQ 120 (Meso Scale Discovery, MSD) permettant l’analyse en multiplex de biomarqueurs par électrochemiluminescence (contact MSD : sophie.viel@universite-paris-saclay.fr). Nos activités qui peuvent être en relation avec celles d'autres plateformes permettent l'identification de nouveaux biomarqueurs pouvant être des cibles thérapeutiques. A propos de l’IPSIT. L’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes technologiques, organisées en deux pôles (IMCELLF et OMICS) et quatre plateformes. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
October 5, 2025 5:39 PM
|
La plateforme de BIOInformatique Intégrative (BIOI2) de l’Institut de Biologie Intégrative de la Cellule (I2BC CNRS/CEA/UPSaclay, Gif-sur-Yvette) accueille depuis fin 2024 un nouvel ingénieur, Baptiste Roelens. Le parcours de Baptiste, commençant par une formation à l'Ecole Polytechnique, combine l'informatique théorique et les sciences du vivant. Il a perfectionné ses compétences à l'Institut Curie, où il a obtenu son doctorat. Sa recherche doctorale s'est concentrée sur le développement d'approches quantitatives pour analyser le rôle du chaperon d'histone CAF-1 dans le maintien d'une structure chromatinienne d'ordre supérieur pendant la prophase méiotique chez la drosophile. Poursuivant son parcours de recherche, Baptiste a rejoint l'Université de Stanford pour développer des pipelines d'analyse d’images, explorant davantage les mécanismes régulant l'organisation des chromosomes et la progression du cycle cellulaire dans la lignée germinale de C. elegans. De retour en France, il a développé des approches d'apprentissage profond de pointe pour la conception de protéines au sein de la société de biotechnologie Viroxis. Grâce à sa double expérience, expérimentale et in silico, Baptiste sera un atout majeur pour la plateforme BIOI2 et est idéalement placé pour collaborer avec la communauté d'imagerie de l'I2BC, en particulier la plateforme Imagerie-Gif, afin de développer des pipelines qui rationalisent le traitement et l'analyse de données d'imagerie complexes. BIOI2 donne accès aux ressources et activités bioinformatiques développées et utilisées à l'I2BC, organise des sessions de formation ciblées sur les outils bioinformatiques, et est une plateforme contributive officielle de l'Institut Français de Bioinformatique (IFB). Pour plus d'informations, veuillez consulter https://bioi2.i2bc.paris-saclay.fr. Contact : contact-bioi2@i2bc.paris-saclay.fr. -> Contact : contact-bioi2@i2bc.paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en octobre 2020, la plateforme publiait son premier FOCUS PLATEFORME … Redécouvrez- le ! FOCUS PLATEFORME : La modélisation structurale pour la prédiction des interactions protéine-protéine: Comment les cellules humaines évitent les conflits entre réplication et transcription ? I2BC / Plateforme de bioinformatique intégrative (BIOI2). La plateforme BIOI2 offre un accès aux ressources et activités en bioinformatique développées et utilisées à l’Institut de Biologie Intégrative de la Cellule (I2BC). Elle regroupe plusieurs outils dans le domaine de l'analyse comparative des génomes, de l'étude des ARN, de la modélisation structurale des machineries cellulaires et de l’analyse de données d’imagerie. BIOI2 offre des services de support pour faciliter et améliorer le traitement des données de séquençage NGS, d’imagerie, de protéomique ou de biophysique et organise des formations en bioinformatique en étroite collaboration avec l'Institut Français de Bioinformatique (IFB). La plateforme est située au cœur des 17 plateformes technologiques de l'I2BC, qui sont membres d’autres Infrastructures Nationales en Biologie et Santé (FRISBI, FBI, France-Génomique, ProFI), afin de favoriser l'intégration et l'exploitation des données générées par les utilisateurs. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.

|
Scooped by
Life Sciences UPSaclay
September 21, 2025 4:28 PM
|
L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. Cet institut héberge quatre plateformes (En savoir plus ?) regroupant ainsi différentes installations et différents savoir-faire dédiés aux plantes. Dans le présent FOCUS PLATEFORME… c’est au tour de la plateforme POPS (transcriptomique) de nous faire découvrir son expertise. L’analyse de transcriptome est largement utilisée dans les projets de recherche. En offrant une vision exhaustive de l’activité des gènes d’un organisme, la transcriptomique permet d’explorer et d’identifier les mécanismes expliquant les phénotypes observés. Cette approche peut s’appliquer à n’importe quel échantillon (tissu ou espèce), cependant son utilisation reste souvent cantonnée à des questions relevant des designs expérimentaux relativement simples. Il y a probablement 2 raisons à cela. La première est la masse très importante de données générées qui nécessite une analyse complexe et experte. D’aucuns pensent que l’intelligence artificielle et les réseaux de neurones pourront lever ce verrou mais ces approches nécessitent une quantité énorme de mesures pour espérer identifier les structures explicatives et prédictives parmi les dizaines de milliers de transcrits analysés dans chaque échantillon. De même, les problématiques de l’agroécologie exigent des analyses faites au champ dans des conditions bien plus complexes (multiples espèces, multiples facteurs) et hétérogènes qu’au laboratoire. Cela nécessite là encore l’analyse d’un très grand nombre d’échantillons. Afin d’apporter la puissance et la sensibilité de la transcriptomique à ces problématiques majeures, il faut donc la rendre accessible au plus grand nombre et en réduire drastiquement les coûts. La plateforme transcriptomique POPS offre un service de screening de transcriptome végétal à très haut débit (plusieurs centaines d’échantillons par projet) et à prix très réduit grâce à la technologie BRB-Seq et à l’automatisation de toutes les étapes (extraction d’ARN, fabrication des banques, dosages et multiplexage). Nos équipements nous permettent également de réduire les volumes réactionnels. Par ce travail qui se poursuit encore actuellement avec l’étude du développement de ces espèces, la plateforme POPS démontre sa capacité à accompagner ses collaborateurs dans des projets « hors du laboratoire » depuis le design expérimental jusqu’à l’interprétation des données produites. -> Contact : pops.ips2@universite-paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en février 2020, juin 2023 et juillet 2024, POPS publiait ses premiers FOCUS PLATEFORME… Redécouvrez- les ! IPS2 / Plateforme de transcriptomique (POPS). La Plateforme de Transcriptomique des Plantes de Paris-Saclay (POPS) propose aux laboratoires académiques ou privés des outils d'analyse du transcriptome par séquençage à haut débit de l'ARN ou RNA-seq. Nous sommes spécialisés dans l'analyse des plantes qu'elles soient modèles ou de culture. Le service proposé inclut la génération des données de RNA-seq, les analyses bio-informatiques et statistiques ainsi que des conseils sur l'interprétation des résultats. Nous accompagnons donc nos collaborateurs depuis le design expérimental jusqu'à l'interprétation des données. La plateforme est certifiée pour l'ensemble de ses activités selon les exigences de la norme ISO 9001 depuis 2012. Afin d'offrir à chaque collaborateur l'analyse la plus adaptée à ses questions biologiques, nous disposons des équipements et des outils d'analyses bio-informatiques et statistiques permettant la construction de transcriptome de novo, l'analyse d'accumulation des ARNs poly-adenylés ou non, des petits ARNs (miRNAs, siRNAs, ribo-seq..) et des ARN poly-adénylés de très petite quantité de départ (jusqu'à 50 pg). Pour les analyses d'expression tissu-spécifiques, nous offrons également des analyses de transcriptome après micro-dissection laser ou en cellule unique grâce à la technologie Chromium (10x Genomics, Rhapsody et scaleBiosciences). Par ailleurs, nous pouvons offrir à nos collaborateurs de sessions de formation sur une semaine à l'analyse statistique des données RNA-seq. A propos de l’IPS2. L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées.

|
Scooped by
Life Sciences UPSaclay
September 7, 2025 4:38 PM
|
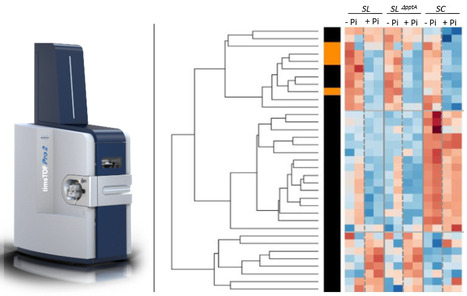
La platefome Protéomique-Gif (SICaPS) de l’Institut de Biologie Intégrative de la Cellule (I2BC, Gif-sur-Yvette, Institut Joliot, CEA, Saclay), propose un ensemble d’analyses protéomiques dédiées à l’analyse en profondeur et la comparaison de protéomes, d’interactomes et de modifications post-traductionnelles. Parmi ses équipements de spectrométrie de masse, le timsTOF Pro de Bruker (acquis grâce à des financements SESAME Santé IdF, Plan Cancer, IBiSA, Université Paris-Saclay et CNRS) permet d'identifier plusieurs milliers de protéines à partir de petites quantités d’extraits protéiques totaux (50 à 200 ng de protéines). Combinée à des méthodes de préparation des échantillons adaptées et à des analyses statistiques multivariées innovantes, la technologie timsTOF Pro offre une profondeur d’analyse qui permet d’étudier finement l'expression normale ou altérée des protéines de différentes souches en fonction de conditions biologiques telles que la composition du milieu de culture, la durée de la culture ou autre paramètre clé. Ces approches offrent des perspectives prometteuses pour accéder à une compréhension globale/systémique des processus moléculaires qui sous-tendent un fonctionnement cellulaire normal ou altéré/pathologique. Qu’est-ce que la technologie timsTOF Pro ? Dans le spectromètre de masse timsTOF Pro, la technologie « Trapped Ion Mobility Spectrometry » (TIMS) permet de séparer et concentrer par mobilité ionique les ions de même rapport masse-sur-charge mais de conformations différentes. Combiné à un analyseur hybride composé d’un quadripôle de sélection ultra-rapide et un analyseur à temps-de-vol, d’excellente résolution, il permet d’acquérir les spectres de fragmentation en mode PASEF (Parallel Accumulation & Serial Fragmentation) avec des vitesses d’acquisition MS/MS, sensibilités et sélectivités élevées. Alors qu’une analyse protéomique classique de type bottom-up, par nanoLC-MS/MS, permet de caractériser chaque peptide protéolytique par son temps de rétention en chromatographie liquide de phase inverse, son rapport masse-sur-charge et son spectre de fragmentation MS/MS, la séparation par mobilité ionique dans la cellule TIMS permet d’augmenter encore la sélectivité des analyses ce qui ajoute une quatrième dimension aux analyses protéomiques. Ainsi le timsTOF Pro permet de réaliser des analyses protéomiques 4D et d’identifier et quantifier un grand nombre de protéines, même peu abondantes, à partir d’échantillons complexes. Un exemple d’application ? Regard sur le protéome de Streptomyces et l’analyse en profondeur d’un très grand jeu de données aux multiples variables ! Dans le cadre d’un projet collaboratif avec l’équipe « Métabolisme Energétique des Streptomyces » (MES) de l’I2BC dirigée par Marie-Joelle Virolle (marie-joelle.virolle@i2bc.paris-saclay.fr), le timsTOF Pro a permis de comparer les protéomes de 3 souches de Streptomyces avec une combinaison de variables (souches sauvages ou mutantes, temps de culture, milieu de culture carencé ou non en phosphate). Chez les Streptomyces, la biosynthèse des antibiotiques est déclenchée en condition de limitation nutritionnelle en phosphate qui est généralement corrélée à un déficit en ATP (stress énergétique). Deux souches de Streptomyces, S. coelicolor et S. lividans, phylogénétiquement très proches, présentent des capacités très différentes à produire les mêmes métabolites spécialisés bio-actifs (antibiotiques). Dans cette étude, les différentes conditions et variables ont été analysées en quadruplicats pour un total de 48 échantillons. La profondeur de l’analyse protéomique réalisée en mode Data-Dependent Acquisition sur le système nanoLC-timsTOF Pro a permis d’identifier et quantifier plus de 4000 protéines de Streptomyces, soit plus de la moitié des protéines prédites in silico. Au-delà de l’analyse sensible par spectrométrie de masse, c’est l’analyse statistique de l’énorme jeu de données protéomiques comportant de nombreuses variables (type de souches, temps et milieu de culture, etc..) qui a été un des principaux défis pour aboutir à une analyse en profondeur, robuste et fiable, des données protéomiques de quantification relatives entre les réplicats de plusieurs conditions expérimentales. L’exploitation de ces données quantitatives multivariées et corrélées a ainsi été rendue possible par le développement d’une approche statistique reposant en partie sur l’utilisation d’un modèle linéaire généralisé à effets fixes (souches, temps et milieu de culture). D’une manière intéressante, la richesse des données générées et ainsi analysées a permis d’établir le profil métabolique et physiologique d’une souche fortement productrice d’antibiotiques (S. coelicolor), par rapport à une souche phylogénétiquement très proche mais faiblement productrice des mêmes antibiotiques (S. lividans). En savoir plus ? Cette étude protéomique combinant les performances du spectromètre de masse timsTOF Pro à une analyse statistique multivariée innovante des données a permis de générer une quantité inédite d’information et de mener une analyse protéomique comparative en profondeur de trois souches de Streptomyces, en fonction de la disponibilité en phosphate, en collaboration avec l’équipe de Marie-Joelle Virolle. L’ensemble des résultats a donné lieu à 3 publications : Lejeune et al., Production Int J Mol Sci 2022 ; Lejeune et al., Front Microbiol 2022 ; Lejeune et al., Res Microbiol 2024. Cette étude illustre l’intérêt et le potentiel des approches de protéomique ultra sensibles en profondeur sur des protéomes totaux et l’expertise de la plateforme Protéomique-Gif non seulement pour l’obtention de données de protéomiques de qualité mais aussi pour l’adaptation de l’analyse des données de protéomiques à des plans expérimentaux complexes. Les nouvelles performances du timsTOF Pro a ainsi permis d’accéder à une profondeur d’analyse des protéomes jamais atteinte et l’arrivée prochaine d’un spectromètre de masse encore plus sensible devrait permettre d'accroitre encore la profondeur des analyses. Si les analyses de protéomes ou d’interactomes complexes vous intéressent, n’hésitez pas à nous contacter et à nous soumettre vos demandes. Nous élaborerons ensemble la stratégie la plus adaptée à votre projet. -> Contacts : Laila Sago (laila.sago@i2bc.paris-saclay.fr), David Cornu (david.cornu@i2bc.paris-saclay.fr) et Virginie Redeker (virginie.redeker@cnrs.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, en 2021, la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? FOCUS PLATEFORME : Protéomique-Gif (SICaPS) : une plateforme dédiée à l'exploration des protéomes et de leurs modifications (27-sept.-21) Protéomique-Gif (SICaPS), La plateforme Protéomique-Gif (SICaPS), labellisée IBiSA, est ouverte à l'ensemble de la communauté scientifique. La plateforme propose des méthodologies de protéomique et des technologies de spectrométrie de masse de pointe pour identifier, caractériser et quantifier les protéines et leurs modifications à partir d'échantillons plus ou moins complexe. Une technologie de dernière génération permet l'identification et la quantification relative de protéines peu abondantes dans des échantillons protéiques très complexes. Des méthodes analytiques adaptées permettent de caractériser des modifications co- , post-traductionnelles ou chimiques des protéines (phosphorylations, lipides, pontages covalents, …). La plateforme dispose d'une solide expertise dans la préparation d'échantillons protéiques de nature variée, leur analyse par spectrométrie de masse et le traitement des données. Elle optimise et développe des méthodes analytiques pour réponse aux besoins des utilisateurs. La plateforme propose une variété de services sous forme de prestations : i) standards : préparations d'échantillons, mesures de masse exacte, identification de protéines, contrôle-qualités de protéines recombinantes ; ii) collaboratives : quantification relative des protéines, comparaisons approfondies de protéomes, interactomes, caractérisation de déterminants structuraux et de modifications des protéines. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.

|
Scooped by
Life Sciences UPSaclay
July 10, 2025 5:12 PM
|
L’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) compte à ce jour 10 plateformes dont AnimEx (Responsable : Valérie Domergue-Duplouy, IR, UPSaclay). AnimEx est située sur le site de la Faculté de Pharmacie de l’Université Paris-Saclay, et correspond au regroupement de 4 animaleries : la plateforme AnimEx1 et le secteur Gnotoxénique de l’UFR de Pharmacie de Châtenay-Malabry, la plateforme AnimEx2 de Clamart et l’animalerie de l’UFR Sciences d’Orsay. Sa mission principale est d’offrir à la communauté scientifique du secteur public ou privé, des structures d’hébergement et d’élevage d’animaux et des équipements pour l’exploration fonctionnelle du petit animal. AnimEx bénéficie dans le bâtiment Henri Moissan de locaux neufs et d’équipements modernes et automatisés. Elle est agréée pour héberger des souris (7100 cages), des rats (770 cages), des lapins, des hamsters et des cobayes. Elle est divisée en 4 secteurs : un secteur expérimental, un secteur élevage avec un statut EOPS, un secteur quarantaine et un secteur A2. Les animaux sont hébergés en portoirs ventilés (sauf lapins et cobayes) et les changes des animaux sont réalisés sous hotte de change. Elle gère l’élevage de souris, de rats et lapins transgéniques et met à disposition des expérimentateurs des équipements pour l’exploration fonctionnelle cardiovasculaire (échographes, télémétrie, pléthysmographe…), pour l’imagerie in vivo (IVIS Lumina) et pour réaliser des tests comportementaux automatisés (open field, labyrinthe en croix surélevée, piscine de Moris…). La plateforme peut réaliser des protocoles à façon (de la conception jusqu’au rendu des résultats) ou des modèles de pathologies (Infarctus du myocarde, Insuffisance cardiaque, hypertension artérielle pulmonaire, obésité, alcoolisation…). Elle dispose de 2 salles de chirurgie. Dans son secteur expérimental, la plateforme peut accueillir des protocoles utilisant de la radioactivité ou des protocoles sur animaux immunodéficients et dans le secteur A2, elle peut accueillir des projets avec des agents pathogènes de classe 2 ou des OGM de classe 2. Le secteur A2 comporte également une zone pour l’hébergement (isolateurs) et l’expérimentation avec des animaux de statut gnotoxénique. Enfin, la plateforme dispose d’une laverie automatisée et robotisée avec un premier robot qui vide les cages sales et un second qui remplit les cages propres. AnimEx fait naturellement partie du réseau CAPSud (Consortium des Animalerie Paris Sud), et la plateforme a déjà publié deux FOCUS PLATEFORME, n’hésitez-pas à les relire … -> Contact : Valérie Domergue-Duplouy (valerie.domergue@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IPSIT / Plateforme AnimEx. La plateforme AnimEx est située dans le bâtiment Henri Moissan, à l'Université Paris Saclay. Sa mission principale est d'offrir à la communauté scientifique du secteur public ou privé, des structures d'hébergement et d'élevage d'animaux et des équipements pour l'exploration fonctionnelle du petit animal. AnimEx est membre de CAPSud (Consortium des Animaleries Paris-Saclay) et est rattaché au Comité d'éthique CEEA n°26. La capacité d'hébergement de l'animalerie est de 7 800 cages (souris, rats, hamsters, cobayes, lapins) pour une superficie de 2 000 m². A propos de l’IPSIT. L’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes technologiques, organisées en deux pôles (IMCELLF et OMICS) et quatre plateformes. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
June 29, 2025 5:04 PM
|
Garder l’élevage au frais, pour ne rien perdre du vivant. Semences, embryons, tissus… Les CRB du réseau CRB-Anim conservent dans le froid le patrimoine génétique et génomique d'une multitude d'animaux de ferme et de compagnie de France. Cette mission encouragée par le GIS IBiSA est tout aussi indispensable pour la recherche que pour l'élevage. Lire l'article. Les INBS se sont réunies à Sète. Le 18 et 19 mars 2025, le domaine du Lazaret, à Sète, a accueilli les représentants de la plupart des infrastructures nationales en biologie et santé (INBS). Thème du séminaire : la communication. Cet évènement a été organisé par le Club des INBS avec le soutien financier du GIS IBiSA. Plus d’infos sur les INBS. Les INBS, bientôt sur le site d'IBiSA. A l'issue des journées de Sète, le GIS IBiSA a entrepris la création de nouvelles pages pour introduire les INBS sur son site internet. Ces pages comprendront une cartographie des plateformes de cœur à l'origine des infrastructures et des informations utiles pour bénéficier de leurs services. Plus d’infos sur les INBS. Formation sur l’empreinte écologique des plateformes et CRB. Le GIS IBiSA propose une nouvelle session de formation sur l'empreinte écologique des plateformes et des CRB, le 20 et 21 novembre 2025 à Paris. La date limite d’inscription a été repoussée au 30 juin 2025. A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Plus d'infos sur le GIS IBiSA. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME.
|

|
Scooped by
Life Sciences UPSaclay
February 1, 11:22 AM
|
Aujourd’hui, un FOCUS PLATEFORME en lien avec le SESAME 2025, dont les résultats globaux pour Paris-Saclay vous ont été présentées le 1er décembre dernier. La plateforme PICCEL (Plateforme d’Imagerie et de Criblage Cellulaire, anciennement plateforme PARi) du Département Radiobiologie Cellulaire et Moléculaire (DRCM, UMR SGCSR Université Paris-Saclay, Université Paris-Cité, INSERM, CEA Centre de Fontenay-aux-Roses (DRF, Institut Jacob)) accompagne depuis de nombreuses années les équipes académiques et industrielles françaises dans la mise en œuvre de criblages cellulaires à haut débit. Son expertise et ses technologies ont contribué à l’identification de nouveaux biomarqueurs, voies de signalisation et petites molécules impliqués dans divers processus biologiques physiologiques ou pathologiques En savoir plus sur les travaux du laboratoire ? Forissier V et al., Theranostics. 2025 ; Rouault CD et al., Nat Commun. 2025 ; Ariey-Bonnet J et al., EBioMedicine. 2023 ; Pina et al., Methods Cell Biol. 2022 ; Villa et al., Cell Death Dis. 2020 ; Lebraud E et al., Nucleic Acids Res. 2020. Grâce à un financement SESAME Île-de-France porté par Karine Dubrana et Guillaume Pinna, la plateforme se dotera en 2026 d’un microscope à haut contenu et à ultra-haut débit, unique en France, renforçant sa capacité à explorer les dynamiques cellulaires à grande échelle. Cet équipement positionnera la plateforme PICCEL parmi les infrastructures nationales de pointe en imagerie cellulaire multiparamétrique et ouvrira la voie à de nouvelles approches de caractérisation fonctionnelle pour la recherche biomédicale. Il permettra notamment la recherche de nouvelles molécules radio-sensibilisantes et/ou radio-protectrices des tissus sains utilisables en radiothérapie pour lutter contre les cancers. L’équipement sera un système d’imagerie par épifluorescence ultra-rapide conçu pour relever les défis émergents du criblage phénotypique multiparamétrique. Il permettra l’acquisition d’images en microplaques (jusqu’au format 1536 puits) dans quatre canaux de fluorescence ainsi qu’en lumière transmise. Le système sera entièrement automatisé grâce à son intégration avec un robot passeur de plaques, assurant la gestion complète du flux expérimental, depuis le stockage des microplaques jusqu’à leur acquisition et leur analyse. Associé à un logiciel avancé d’analyse d’images, il permettra le phénotypage cellulaire multiparamétrique. Dans cette configuration, le système pourra imager et quantifier jusqu’à 300 plaques 384 puits par jour, levant ainsi le principal verrou de débit des approches de crible par imagerie traditionnelles et permettant l’exploration de tests cellulaires complexes, multiplexés ou en cinétique multipoints. Cet équipement offrira à la plateforme PICCEL la capacité d’interroger une large variété de banques de dimensions et de natures très diverses (banques de petites molécules, banques CRISPR ou ARN interférentes) pour répondre à des questions biologiques jusque-là hors de portée. La plateforme PICCEL verra ses capacités techniques profondément transformées, donnant accès à ses partenaires à un outil unique en France pour l’imagerie cellulaire multiparamétrique à haut contenu. Au-delà des criblages, l’équipement soutiendra de nombreux projets en biologie cellulaire, radiobiologie, immunologie ou cancérologie, et permettra la formation de jeunes chercheurs à l’imagerie automatisée et à l’analyse quantitative. La plateforme PICCEL est ouverte à l’ensemble de la communauté scientifique académique et industrielle. Pour toute demande ou projet, n’hésitez pas à nous contacter. -> Contact : Guillaume Pinna (Responsable Plateforme PICCEL) — guillaume.pinna@cea.fr; Karine Dubrana (Directrice des plateformes DRCM) — karine.dubrana@cea.fr Plug In Labs Université Paris-Saclay : cliquer ICI DRCM / Plateforme d'Imagerie et Criblage Cellulaire (PICCEL). La Plateforme d'Imagerie et Criblage Cellulaire (PICCEL), située dans le Département de Radiobiologie Cellulaire et Moléculaire (DRCM, CEA), est dédiée au criblage à haut débit (HTS) et à haut contenu (HCS) sur modèles cellulaires. La plateforme dispose de l’expertise et de l’infrastructure nécessaires pour la conception, la miniaturisation et l’automatisation de tests cellulaires, le criblage systématique et parallèle de collections de perturbateurs biologiques (siARNs, CRISPRi/a, miRNAs, petites molécules, composés médicaments…), la quantification de systèmes rapporteurs simples ou multiplexés, ainsi que l’analyse statistique de jeux de données de grande dimension. L’imagerie à haut contenu constitue notre approche privilégiée pour le profilage phénotypique, incluant des mesures multiplexées à partir de marquages fluorescents ou label-free. En complément de ses activités de criblage, PICCEL met également à disposition ses équipements robotiques et d’imagerie pour des applications de caractérisation et d’exploration cellulaire de routine. La plateforme travaille en étroite collaboration avec les autres plateformes du DRCM pour proposer des solutions adaptées à chaque projet. L’équipe adopte une démarche qualité rigoureuse, garantissant la traçabilité et la confidentialité des résultats tout au long des projets de criblage.

|
Scooped by
Life Sciences UPSaclay
January 18, 11:17 AM
|
Un évènement à ne pas manquer ! Le 20 février 2026, le Service Hospitalier Frédéric Joliot (SHFJ, Institut des Sciences du Vivant Frédéric Joliot, Orsay) et le laboratoire de recherche BIOMAPS (CEA, CNRS, Inserm, Université Paris-Saclay) vous invite à l’inauguration de son IRM à bas champs (Magnetom Free.max 0,55T). Cet équipement unique sur Paris-Saclay, viendra compléter l’offre de PASREL-Imagerie avec un nouvel outil clinique de pointe, et renforcer le nœud Paris-Sud de l’infrastructure nationale France Life Imaging (FLI). Cette nouvelle IRM bas champs viendra accroître les capacités technologiques et scientifiques du SHFJ pour caractériser les solutions innovantes des acteurs industriels dans différentes aires thérapeutiques. Inscription gratuite, mais obligatoire : https://irm055t biomaps.sciencesconf.org Ci-dessous le programme de cette journée : 9:30 – 10:00: Accueil 10:00 – 10:10 : Le mot de France Life Imaging (Régine Trébossen) 10:10 – 10:20 : BioMaps vers les bas champs (Vincent Lebon / Nicolas Tournier) 10:20 – 10:40 : Champs magnétiques : des hauts et des bas (Marie Poirier Quinot) 10:40 – 11:00 : Bas champs : the come back (Angéline Nemeth) 11:00 – 11:10 : MAGNETOM Free.Max , une IRM à 0,55 T (Siemens Healthineers) 11:10 – 11:50: L’imagerie cardiovasculaire à 0,55T (Stanislas Rapacchi) 11:50 – 12:15 : Respirer à plus bas champs (Xavier Maître) 12:15 – 13:30 Buffet et visite -> Contact : Rose-Marie Dubuisson (rose-marie.dubuisson@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI BIOMAPS / PASREL / Imagerie préclinique et clinique, in vivo, IRM, 0.55 T (SHFJ). L’IRM 0.55T FreeMax permet l’imagerie anatomique de la tête et du cou, du rachis, du thorax, de l’abdomen, du genoux, coude, épaule. Le principal atout de cet IRM réside en ces contrastes propres au bas champ, rendant accessible l’exploration de régions habituellement difficiles à sonder telles que celles impactées par l’hétérogénéité due aux susceptibilités magnétiques (poumon, zone avec implants magnétiques). Sa grande ouverture de 80 cm de diamètre, contre 60 cm ou 70 cm habituellement, favorise l’accueil d’une population variée, incluant les personnes claustrophobes ou à forte corpulence. Le plateau IRM bas-champ 0.55T, réservée à la recherche clinique, présente l’avantage de pouvoir passer des cohortes en journée. Il appartient aux plateformes d’imagerie clinique du SHFJ, adossée au service de médecine nucléaire, permettant un accueil approprié à un examen clinique. La diversité d’imageurs de cette plateforme facilite les études multimodales (IRM, US, …). A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
January 3, 1:11 PM
|
Les INBS débarquent sur le site web du GIS IBiSA ! Les infrastructures nationales en biologie santé (INBS) sont au cœur de la stratégie française de recherche en science du vivant. La nouvelle page « IBiSA et les INBS » dévoile les missions du GIS pour l'organisation et la consolidation des 27 INBS inscrites sur la feuille de route du Ministère. IBiSA et les INBS. Des pages web dédiées à chacune des 27 INBS. 19 infrastructures sont déjà représentées sur le site web d'IBiSA. De nouvelles pages décrivent l'organisation de chaque INBS, précisent leurs conditions d'accès et offrent une cartographie de leurs plateformes, pour le moment limitée aux plateformes labellisées IBiSA. Les pages des 8 autres INBS seront peu à peu publiées. Explorer les INBS. Un annuaire spécial pour les plateformes des INBS. A partir des pages INBS et à l'instar de l'annuaire des plateformes IBiSA, le GIS lance un annuaire des plateformes INBS. Dans cette version encore en construction, vous retrouverez dans un premier temps les plateformes labellisées IBiSA. Étape suivante : créer des pages pour toutes les autres plateformes des INBS. Annuaire des plateformes INBS. Résultats de l'appel d’offres Plateformes IBiSA 2025. En 2025, le GIS IBiSA a reçu 77 candidatures de plateformes dans tous les domaines des sciences de la vie. Après 77 visites et 130 expertises, il a décerné 25 labels et distribué 2 210 000 € de financements à 45 plateformes technologiques en biologie, santé et agronomie. Consulter les résultats. Appel d’offres Plateformes IBiSA 2026. La prochaine édition de l'appel Plateformes IBiSA sera ouverte en janvier 2026. Les modalités de candidature seront mises à jour sur le site web. En attendant, retrouvez toute l'actualité du GIS sur les réseaux sociaux. Appel d’offres Plateformes IBiSA. A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Plus d'infos sur le GIS IBiSA. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME.

|
Scooped by
Life Sciences UPSaclay
December 14, 2025 5:00 PM
|
2025, année hautement radioactive ! Trois anniversaires se fêtent dans le calendrier scientifique : les 90 ans du prix Nobel des Joliot-Curie pour la découverte de la radioactivité artificielle, les 80 ans du CEA, et les 50 ans du premier cyclotron biomédical installé au Service Hospitalier Frédéric Joliot (SHFJ, Institut des Sciences du Vivant Frédéric Joliot, Orsay). Né en 1958 au cœur de l’hôpital d’Orsay, le SHFJ incarne depuis ses débuts une volonté affirmée de rapprocher la recherche sur les radioisotopes des patients et de la clinique. À l’origine, les isotopes provenaient des réacteurs de Saclay. Mais en 1975, tout change : le SHFJ s’équipe de son propre cyclotron biomédical, construit par CGR MeV (filiale de Thomson), une première en France. L’année suivante, l’inauguration est à la hauteur de l’événement, sous le regard du ministre de l’Industrie Michel d’Ornano et de la ministre de la Santé Simone Veil. Dès lors, le SHFJ entre dans une nouvelle ère : production autonome d’isotopes, maîtrise de la radiochimie, et innovation en imagerie TEP. En 1998, coup d’éclat : avec CisBio International, le SHFJ dépose la première autorisation de mise sur le marché d’un médicament radiopharmaceutique pour la TEP — le 18F-FDG, désormais incontournable dans le diagnostic du cancer. Le tournant du siècle marque un nouveau cycle : le premier cyclotron prend une retraite méritée (il sera complètement démantelé en 2009), remplacé par une machine IBA (réceptionnée fin 2001, pour une mise en service courant 2003), semblable à celles équipant aujourd’hui les sites industriels de production de radiopharmaceutiques. En 2017, un troisième cyclotron rejoint l’aventure, en partenariat avec PMB, pour créer une chaîne de fabrication entièrement automatisée, de la production de l’isotope au conditionnement final du produit. En un demi-siècle, le SHFJ a bâti une expertise unique dans la chimie du fluor-18 et du carbone-11, deux isotopes émetteurs beta+ à vie brève (109,8 min, et 20,38 min, respectivement), d’importance primordiale pour l’imagerie par Tomographie par Emission de Positions (TEP). Au travers de ses plateformes (BIOMAPS – CEA, CNRS, Inserm, Université Paris-Saclay) et ses partenaires, PASREL-Imagerie offre aujourd’hui à la communauté scientifique saclaysienne et au-delà un accès unique à des outils préclinique et clinique de pointe. Mais l’histoire ne s’arrête pas là. Aujourd’hui, l’imagerie devient « théranostique », le diagnostic accompagne la thérapie grâce à de nouveaux radioisotopes à visée thérapeutique, comme le lutécium-177 (émetteur beta-) ou l’actinium-225 (émetteur alpha). Le SHFJ, fidèle à son ADN d’innovation, explore ces voies prometteuses tout en consolidant son expertise autour du fluor-18 et du carbone-11, compagnons d’imagerie diagnostique idéaux de ces radiothérapies vectorisées émergentes. Prochaine étape, un nouveau cyclotron (?) pour BIOMAPS / PASREL Tracers (SHFJ), le déménagement du SHFJ vers l’infrastructure PASREL, à proximité de l’hôpital Paris Saclay, à l’horizon 2029. La plateforme a déjà publié plusieurs FOCUS PLATEFORME ces dernières années. Les relire ? -> Contact: Bertrand Kuhnast (bertrand.kuhnast@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI BIOMAPS / PASREL Tracers (SHFJ). La plateforme PASREL tracers fabrique à façon des radiotraceurs et radiopharmaceutiques expérimentaux marqués avec des isotopes émetteurs de positons (C-11, F-18, Cu-64, Zr-89) pour une utilisation en imagerie TEP préclinique ou clinique, ainsi qu’avec du Lu-177, émetteur béta- pour la thérapie en préclinique et bientôt avec des émetteurs alpha. Ces médicaments diagnostiques (petites molécules et biomolécules) aux indications variées (oncologie, neurologie, infectiologie, etc.) peuvent être utilisés au sein de nos plateformes d’imagerie ou distribués dans votre établissement (hors C-11). A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
November 30, 2025 5:48 PM
|
Les résultats sont tombés le 21 novembre 2025 : ce sont cinq projets d’équipements de l’Université Paris-Saclay qui sont lauréats de l’AAP SESAME 2025, dont deux « cœur de métier » du domaine Sciences de la Vie & Santé (SCOUT et ScreenFast), et deux « à ses interfaces » (BioALTO, BioESEM), le dernier projet (MasqLE) relevant du domaine Sciences & Ingénierie. Ces 5 projets seront subventionnés par la région Ile-de-France, dans le cadre de cet AAP, à hauteur de 66% du coût du projet. Vous avez dit SESAME ? Le dispositif SESAME (pour « Soutien aux Équipes Scientifiques pour l’Acquisition de Moyens Expérimentaux » vise à cofinancer des équipements scientifiques nécessaires au fonctionnement des laboratoires de recherche publics franciliens pour mener des projets d’envergure dans des champs thématiques autres que les Domaines de recherche et d’Innovation Majeurs (DIM) et les Questions d’Intérêt Majeur (QIM), et hors du périmètre d’intervention du Genopole et des projets inscrits au Contrat de Plan État-Région 2021-2027. SESAME 2025 : Demande de subventions versus montants alloués par la région ? Sur les 33 projets de recherche éligibles et évalués par la Région Ile-de-France (représentant un total de 30,5 millions d’euros HT de demandes de subvention), seuls 13 ont été sélectionnés via SESAME 2025, et ceci pour un montant de financement HT de 5,5 millions d’euros. Ces 13 projets recouvrent différentes thématiques scientifiques, dont 7 en biologie et santé, 2 en environnement et agrosciences, 2 en matériaux, 1 en physique et électronique, et 1 en informatique et sciences humaines et sociales. SESAME 2025 : Comment se positionne l’Université Paris-Saclay ? Sur ces 13 projets, 5 sont portés par l’Université Paris-Saclay, soit une petite moitié (38%) ! Un projet relève du domaine Sciences & Ingénierie : MasqLE (masqueur de lithographie électronique), C2N (Centre de Nanosciences et de Nanotechnologies, UMR 9001 - CNRS, UPSaclay, pour 1 M€ de subvention et un projet à 3,2 M €), et quatre relèvent directement ou indirectement du domaine Sciences de la Vie & Santé : SCOUT et Screen-Fast (SDV&S « cœur de métier »), et BioALTO et BioESEM (SDV&S « à ses interfaces ». Ci-dessous les premières informations concernant ces quatre projets SDV&S « cœur de métier » mais aussi « à ses interfaces », projets que vous découvrirez par ailleurs plus en détails dans les prochaines semaines via des FOCUS PLATEFORME dédiés. SCOUT | subvention = 500 000 € (IPSIT - Ingénierie et Plateformes au Service de l'Innovation Thérapeutique, UMS UPSaclay US31 Inserm, UAR3679 CNRS et 2I - Infection et Inflammation, UMR 1173 – INSERM, UVSQ, UPSaclay) – Cytométrie spectrale pour la caractérisation optimale, l’analyse non supervisée, et le tri d’éléments individuels. Screen-Fast | subvention = 260 000 € (SGCSR - Stabilité Génétique, Cellules Souches et Radiations, UMRE E008 - U1274 - INSERM, CEA, Univ. Paris, UPSaclay) – Technologie de rupture pour l’identification par criblage à haut contenu de nouveaux biomarqueurs et médicaments pour la santé. BioALTO | subvention = 197 183 € (IJCLab, UMR 912 - CNRS, Univ Paris-Cité, UPSaclay) – Plateforme expérimentale à ALTO pour la recherche préclinique en hadrothérapie. BioESEM | subvention = 260 000 € (LGPM - Laboratoire de Génie des Procédés et Matériaux, EA 4038 – CentraleSupelec, UPSaclay) - Observation en haute résolution des structures biologiques à conditions variables d’hydratation. A suivre donc ! -> Contact : Frédéric Dollé (frederic.dolle@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
November 16, 2025 5:53 PM
|
CATI est une plateforme de services universitaires française, désormais labellisée unité de service multi-institutions US52-UAR2031. CATI a apporté son soutien à la partie neuroimagerie d'environ 60 études multicentriques sur des troubles neurologiques et psychiatriques (maladie d'Alzheimer, maladie de Parkinson, maladie de Huntington, ataxie spinocérébelleuse, sclérose latérale amyotrophique, troubles bipolaires, schizophrénie, etc.), dont plusieurs essais interventionnels, et participe actuellement à des études multicentriques en néonatologie et en neuro-oncologie. Créée en 2011, la plateforme vise à rendre la gestion multicentrique de la neuroimagerie accessible à tout projet à un coût raisonnable. À ses débuts, la plateforme s'appuyait sur un large éventail d'expertises provenant de plusieurs équipes de recherche, au sein de NeuroSpin (Institut des Sciences du Vivant Frédéric Joliot, Centre CEA Paris-Saclay, Gif-sur-Yvette), telles que les équipes en place CEA et INRIA, mais aussi d’équipes de l'ICM (CNRS - SU - Inserm - SU - Inria) et du LIB (SU - Inserm - CNRS). S'appuyant sur un vaste réseau d'installations d'imagerie standardisées, la plateforme collecte et contrôle les acquisitions et effectue un traitement systématique des images à partir d'un large portefeuille couvrant la plupart des modalités IRM / TEP / SPECT. Des besoins spécifiques peuvent être pris en charge à la demande (nouvelle séquence IRM, radiotraceur TEP ou pipeline de traitement d'images) grâce à des liens étroits avec les laboratoires de recherche environnants et un réseau de collaborations (par exemple, le réseau REMI, le réseau FUN …). Les données brutes et les analyses peuvent être mises à la disposition du consortium de l'étude via un service web sécurisé, selon les demandes du chef de projet. Grâce aux efforts de standardisation entre les études et les pathologies, la plateforme favorise les collaborations afin d'accélérer l'émergence de projets de recherche sur le cerveau à l'échelle du big data et dans un cadre visant à l’émergence de solutions innovantes en santé numérique. -> Contact : Jean-François Mangin (jean-francois.mangin@cea.fr), Marie Chupin (marie.chupin@icm-institute.org) Plug In Labs Université Paris-Saclay : cliquer ICI NEUROSPIN / CATI. Créée en 2010, le CATI, Centre d'acquisition et de traitement des images, est une plateforme nationale dédiée aux études de neuro-imagerie multicentriques. Cette infrastructure a pour objectif de faire émerger des protocoles standardisés d'acquisition et d'analyse d'images, le tout pour obtenir un parc des données aux caractéristiques harmonisées sur les centres d'imagerie partenaires, avec un contrôle qualité systématique. À cette fin, ce centre est mis à disposition de l'ensemble de la communauté des cliniciens et des chercheurs étudiant la maladie d'Alzheimer et autres démences neuro-dégénératives. Il s'agit de promouvoir le travail en réseau et de parvenir à un gain de temps dans la détection et le suivi de la maladie. La qualité des équipements et des images ayant beaucoup progressé ces dernières années, le CATI a également pour mission d'assurer le transfert des technologies qu'il met au point vers le milieu hospitalier. Et, de fait, permettre à l'industrie pharmaceutique d'avoir accès à des cohortes de patients bien identifiées. À propos de NeuroSpin. NeuroSpin est une infrastructure de recherche sur le cerveau exploitant des grands instruments d'imagerie. NeuroSpin offre à la communauté scientifique publique et privée la possibilité de faire progresser la connaissance du cerveau, et particulièrement du cerveau humain, en proposant un accès à des méthodologies de pointes en imagerie cérébrale et en neuro-informatique. NeuroSpin développe et met à la disposition de la communauté des instruments uniques, notamment en imagerie très hauts champs et dans le domaine des big data. Cette offre s'inscrit dans le cadre des missions spécifiques de NeuroSpin qui sont : i) analyser les fonctions du cerveau humain, leur développement dans l'enfance, et l'impact de la culture et de l'éducation ; ii) identifier les marqueurs et les mécanismes de maladies neurologiques, psychiatriques et neurodéveloppementales ; iii) comparer le cerveau humain et celui d'autres espèces animales ; développer et tester des méthodes d'imagerie à toutes les échelles d'observation : par résonance magnétique (IRM), par électro- et magnétoencéphalographie (EEG et MEG), et par électrophysiologie massivement parallèle ou l'imagerie photonique et v) développer des logiciels spécialisés dans le traitement et la modélisation des grands jeux de données en neuroimagerie. À propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
October 22, 2025 4:56 PM
|
Les iPSC, des cellules reprogrammées pour explorer les maladies humaines. Encore peu connues du grand public, les cellules souches pluripotentes induites (iPSC) révolutionnent la recherche biomédicale : elles permettent de créer des modèles expérimentaux uniques et complexes pour étudier les maladies humaines. En France, plusieurs plateformes IBiSA, comme iPSC et stemCARE, s'organisent pour structurer une communauté sur ce thème. Lire l'article. Les nouvelles thérapies au sein du GIS IBiSA. Les plateformes spécialisées sur les iPSC font partie d'un ensemble de structures IBiSA tournées vers les nouvelles thérapies. Celles-ci couvrent un champ de recherche large comprenant les iPSC, les organoïdes, la bio-impression de tissus, les thérapies géniques vectorisées et la génération d'anticorps, dont les nanobodies. Les plateformes nouvelles thérapies. Des candidatures en hausse lors des appels IBiSA. Avec le temps, les plateformes impliquées dans les nouvelles thérapies sont de plus en plus nombreuses à candidater aux appels IBiSA. En 2025, elles sont 11 à avoir demandé un label ou un financement, ce qui représente 12% des candidatures reçues ! Le bureau d'IBiSA se réunira courant décembre pour délibérer. Les plateformes nouvelles thérapies. Appel d’offres Plateformes IBiSA 2026. La prochaine édition de l'appel d’offres Plateformes IBiSA sera ouverte en janvier 2026. Les modalités de candidature seront mises à jour sur la page dédiée du site web. Appel d’offres Plateformes IBiSA. A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Plus d'infos sur le GIS IBiSA. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
October 12, 2025 4:31 PM
|
Le Service Hospitalier Frédéric Joliot (SHFJ, Institut des Sciences du Vivant Frédéric Joliot, Orsay) et plus particulièrement son UMR BioMaps (UMR CEA/CNRS/Inserm/Université Paris-Saclay) opère plusieurs plateaux techniques d’imagerie, dont l’un d’entre eux équipé d’un système hybride combinant TEP (tomographie par émission de positons) et IRM (imagerie par résonance magnétique : BIOMAPS / PASREL / Imagerie préclinique et clinique, in vivo, TEP/IRM (SHFJ). Recherche biomédicale par imagerie et partenariat industriel ? C’est tout à fait possible à BioMaps ! La société JELLYNOV est à l’origine d’une solution innovante dans la lutte contre le surpoids et l’obésité, urgence de santé publique. Cette solution se présente comme un ballon gastrique transitoire ingéré sous forme de gel, capable d’induire une sensation de satiété et permettant ainsi de limiter la consommation alimentaire. Pour valider cliniquement la dynamique de cette solution unique, JELLYNOV a fait appel à l’expertise IRM de l’équipe de Développements Méthodologiques et Instrumentation (BioMaps, SHFJ). L’équipe a ainsi monté pour cette société un protocole IRM permettant le suivi temporel de l’occupation dans l'estomac du ballon intragastrique. Une première phase de preuve de concept a été nécessaire pour valider la possibilité de mesurer le volume du contenu de l’estomac sans produit de contraste, hors ingestion du gel, avec l’utilisation de séquences IRM adaptées pour l’occasion. Le défi à relever a été de combiner résolution la plus fine possible, rapidité d’acquisition pour minimiser les mouvements thoraciques (acquisition réalisée en apnée pour s’affranchir du mouvement respiratoire), tout en gardant un signal sur bruit correct pour identifier les différents tissus. Un protocole a pu être ainsi défini en vue de la seconde phase qui a consisté en l’étude clinique du gonflement du ballon gastrique et de sa résorption de l’estomac. Cette étude étant exploratoire, elle s’est déroulée sur une petite cohorte (une douzaine de volontaires). Elle a été déclinée en trois étapes sur une durée d’environ quatre heures, durant lesquelles une même séquence d’imagerie anatomique 2D est répétée. Après une première étape d’imagerie de référence à jeun, une deuxième étape d’imagerie post-ingestion du gel est exécutée à intervalles de temps réguliers sur une vingtaine de minutes pour évaluer son gonflement dans l’estomac. La dernière étape est l’évaluation de sa résorption de l’estomac en alternant des phases d’imageries, où le volontaire est couché, avec des phases pendant lesquelles celui-ci est soit debout soit assis afin de ne pas modifier la physiologie de vidange gastrique habituelle. Cette évaluation est effectuée entre 40 et 180 min post-ingestion. Les volumes d’air et de chyme de l’estomac sont ensuite mesurés à posteriori sur ces données. Le protocole d’imagerie est effectué sur l’imageur commercial hybride TEP/IRM 3T (SIGNATM PET/MR, GE Healthcare), associés à des antennes de réception dédiées à l’imagerie de l’abdomen/thorax (antennes CMA et GEMFlex- M, GE Healthcare). Les résultats ont montré un gonflement rapide du ballon gastrique dans l’estomac et son évacuation totale de l’estomac au bout de 90 à 180 minutes. -> Contact : Rose-Marie Dubuisson (rose-marie.dubuisson@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme a déjà publié plusieurs FOCUS PLATEFORME ces dernières années. Les relire ? BIOMAPS / PASREL / Imagerie préclinique et clinique, in vivo, TEP/IRM (SHFJ) : La plateforme d'imagerie TEP-IRM permet de réaliser des acquisitions simultanées TEP (tomographie par émission de positons) et IRM (imagerie par résonance magnétique, 3 Tesla) cliniques et précliniques, corps entier, synchronisées ou non à la respiration, statiques ou dynamiques. L'imagerie TEP se fait au moyen de radiotraceurs extérieurs ou produits sur site. Les compétences sont disponibles pour l'analyse et l'interprétation des données pharmacocinétiques en regard de données d'imagerie multimodale acquises au SHFJ ou dans d'autres centres. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
September 28, 2025 4:38 PM
|
L’UMS-IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique, Faculté de Pharmacie, Bâtiment Henri Moissan, Orsay) a le plaisir de vous annoncer l’arrivée de Pauline Tran, assistante ingénieure, qui œuvrera sur deux de nos plateformes : ACTAGen d’une part (IPSIT / Plateforme d'Analyse Transcriptomique et Génomique - ACTAGen) et BIOINFO d’autre part (IPSIT / BIOINFO). Diplômée de l’École de Biologie Industrielle (EBI) en 2021, Pauline a d’abord rejoint l’UMS-IPSIT en mai 2024 dans le cadre d’un CDD INSERM, avant de réussir le concours externe d’assistant ingénieur INSERM. Elle est en poste depuis janvier 2025. Dotée d’une double compétence en biologie moléculaire et en bioinformatique, Pauline vient renforcer les activités de nos plateformes, avec une attention particulière portée au soutien des utilisateurs, tant pour la mise en place de protocoles expérimentaux que pour l’analyse des données. Pauline s'intéresse particulièrement au développement de nouvelles approches en single-cell, visant à mieux décrypter la complexité et l’hétérogénéité des systèmes biologiques. Elle participe également activement à la mise en place de la PCR digitale, une technologie émergente qui ouvre de nouvelles perspectives en quantification absolue d’acides nucléiques. Avec son dynamisme et sa créativité, Pauline va donner un nouvel élan à nos projets et booster les ambitions scientifiques de toute la communauté. Bienvenue à bord ! -> Contact : Pauline Tran (pauline.tran@universite-paris-saclay.fr), Claudine.Delomenie (claudine.delomenie@universite-paris-saclay.fr), Florent Dumont (florent.dumont@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IPSIT / Plateforme d'Analyse Transcriptomique et Génomique (ACTAGen). La plateforme ACTAGen, située sur le campus de Pharmacie de l'Université Paris-Saclay, fait partie du réseau des Plateformes de GENOmique Paris-Saclay (GENOPS). Elle est l'une des 10 plateformes technologiques qui composent l'Unité Mixte de Service UMS-IPSIT (Ingénierie et Plateformes au Service de l'Innovation Thérapeutique). Nous offrons notre expertise aux différents acteurs de la recherche, de l'Université Paris-Saclay ou de laboratoires extérieurs, de statut public ou privé. Nous effectuons l'analyse qualitative et quantitative du transcriptome par séquençage haut débit (NGS) ou microarrays, et la mesure d'expression de gènes choisis par PCR quantitative en temps réel ou digitale, à partir de cellules et tissus d'origines variées. Le traitement statistique et bioinformatique des données est réalisé avec la plateforme IPSIT/BIOINFO. Nos activités contribuent à la compréhension de mécanismes physio-pathologiques, à l'identification de cibles thérapeutiques et de biomarqueurs, ou encore à l'évaluation de l'activité biologique de formulations de principes actifs, intéressant les secteurs académique et industriel. Nous nous appuyons sur nos partenariats au sein du réseau GENOPS pour orienter nos évolutions technologiques comme par exemple : i) Le séquençage du transcriptome (RNA-SEQ) et de régions génomiques (DNA-SEQ), en partenariat avec la plateforme de séquençage NGS de l'I2BC (CNRS, Gif-sur-Yvette), ii) La quantification par PCR digitale (Droplet Digital PCR, ddPCR) de cibles nucléiques rares ou à partir de petits échantillons, en partenariat avec la plateforme de microgénomique @BRIDGe (INRAe, Jouy-en-Josas). ACTAGen travaille au développement d’approches single cell pour mieux répondre aux besoins des équipes de recherche. IPSIT / BIOINFO. La plateforme de bioinformatique de l’UMS-IPSIT propose des prestations de services en bioinformatique et biostatistiques. Nous mettons l’accent sur le suivi des projets, la formation et l’autonomisation de nos utilisateurs. La création d’outils sous forme de package R accessible et portable permet de faciliter ce transfert de compétence. A propos d’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes techniques, organisées en deux pôles technologiques (IMCELLF et OMICS) et quatre plateformes. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi ses FOCUS PLATEFORME décrivant toutes ses expertises !

|
Scooped by
Life Sciences UPSaclay
September 13, 2025 6:47 AM
|
Ce jour, place à un FOCUS PLATEFORME sur ECOSYS (UMR INRAE/AgroParisTech/UPSaclay, Palaiseau) et son expertise « radiochimie vie longue », partie intégrante du plateau de chimie environnementale de recherche. Cette expertise au sein de ce plateau est centrale pour les activités du pôle Sol&Tox d’ECOSYS portant sur le transfert des contaminants organiques (pesticides, contaminants émergents), y compris les microplastiques, des sols vers les organismes vivants (faune et plante) et sur les biotransformations de ces composés. Le suivi de l'évolution des traceurs radioactifs du C est également utile à l'étude de la dynamique des matières organiques ou de l’assimilation microbienne. Les informations fournies par ces techniques ne peuvent pas être obtenues avec les techniques de chimie analytique classique, comme par exemple la formation des résidus non extractibles des contaminants organiques, permettant ainsi de faire des bilans de masse complets. Le plateau est installé sur le campus Paris-Saclay, et est largement ouvert à des sollicitations extérieures et mis à disposition pour d'autres unités. Il vient compléter un ensemble très complet d'équipements ce qui le rend assez unique dans le cadre des activités de chimie environnementale au sein du bâtiment Agroécologie du Campus Agro Paris-Saclay. RHIZOPharma, ou une adaptation récente du RHIZOtest®. L’élaboration de biotests tels que le RHIZOtest (cf illustration, partie supérieure) doit permettre d’estimer la phytodisponibilité de contaminants, comme les composés pharmaceutiques qui se retrouvent dans les produits résiduaires organiques (par ex. les boues d’épuration ou les composts), et leur bioaccumulation dans les plantes. Le projet RHIZOPharma a pour but d’adapter le RHIZOtest à l’étude du transfert sol-plante, ici le chou, de deux médicaments – deux antibiotiques : le Sulfaméthoxazole (SMX) et la Ciprofloxacine (CIP), en conditions contrôlées dans des laboratoires habilités à la manipulation de radioéléments. L’utilisation de contaminants radiomarqués au carbone-14 (isotope radioactif émetteur beta-moins et de période 5730 ans) permet d’accéder à des bilans précis de la distribution de ces derniers, par exemple dans le sol, les racines et les parties aériennes des plantes. Les plants de chou sont cultivés dans un premier temps en chambre de culture en hydroponie (cf. Schéma partie pré-incubation) pour permettre le développement d’un tissu racinaire plan et dense. Les plants sont ensuite mis en contact avec une galette de sol, cette galette étant préalablement contaminée par ajout d’une solution contenant la molécule radioactive étudiée. Cette seconde phase dite de contact permet donc de recréer les échanges réels d’eau et de nutriments tout en séparant physiquement le sol des racines. Durant ces 8 jours, les biotests sont placés dans des enceintes étanches avec en sortie un système de piégeage du 14CO2 par de la soude. Les mesures réalisées portent sur : i) la minéralisation de 14C-CTO via le suivi de la production de 14CO2 (piège CO2) (mesure de la radioactivité par scintillation liquide) ; ii) la formation de résidus de 14C-CTO plus ou moins fortement liés au sol (fraction disponible (extraction aqueuse) puis fraction adsorbée, maximum extractible (extraction avec un solvant organique)) ; iii) la mesure de la radioactivité par scintillation liquide (fraction de résidus non-extractibles par combustion totale (sol et plantes) à l’oxidizer) ; et iv) la mesure de la radioactivité par scintillation liquide. En savoir plus sur ces techniques ? Bravin et al., Environ. Pollut. 2020; Benoit et al., Encyclopedia of Soils in the Environment (Elsevier 2023), 470-482. N’hésitez pas à solliciter les responsables des plateaux pour monter des collaborations dans le cadre de vos projets de recherche. -> Contact : plateau radio-isotopes : Valérie Dumény (Valerie.Dumeny@inrae.fr) / plateau de chimie environnementale : Marjolaine Deschamps (Marjolaine.Deschamps@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en avril 2020, la plateforme publiait son premier FOCUS PLATEFORME … Redécouvrez- le ici ! ECOSYS / Plateaux de chimie environnementale de recherche : Les objectifs des plateaux de chimie environnementale de recherche sont de mettre à disposition des compétences et des outils d'analyse et d'expérimentation, pour supporter des recherches dans le domaine de l'environnement, avec le partage d'une problématique commune sur la caractérisation du fonctionnement de différents compartiments de l'environnement et sur l'évaluation des impacts liés à la dynamique des contaminants dans l'environnement. Ces plateaux, réunis dans un pôle de chimie environnementale, fonctionnent avec du personnel des unités constitutives (de techniciens aux ingénieurs de recherche), avec un temps partiel dédié, et un partage des compétences et des équipements analytiques. Ils constituent un support technique pour la construction de projets collaboratifs et pour structurer le partenariat externe. Les différents plateaux sont : i) radio-isotopes (expérimentations avec des molécules marquées au 14C), ii) isotopes stables (expérimentations avec utilisation de 13C et 15N), iii) chromatographie liquide - spectrométrie de masse (analyses de composés organiques dans des matrices complexes), iv) analyse des gaz à effets de serre (quantification de CO2, NH4, N2O et SF6), et v) mesure de composés organiques volatils (mesures en continu au champ ou en laboratoire).

|
Scooped by
Life Sciences UPSaclay
August 31, 2025 5:19 PM
|
Créée en 2017, la plateforme d’histologie – anatomocytopathologie de Genopole propose les équipements, le savoir-faire et l’expertise nécessaires permettant de répondre aux besoins croissants des entités du biocluster Genopole dont l’activité principale est la recherche et le développement d’innovations thérapeutiques. Les prestations de service proposées par cette plateforme certifiée ISO 9001:2015 sont tout naturellement la préparation des tissus (cryopréservation ou inclusion en paraffine), leur section au cryostat ou au microtome, leurs colorations histologiques ou la réalisation de marquages immunohistochimiques (DAB ou fluorescents) via des automates et enfin l’analyse morphométrique des tissus observés par microscopie. L’expertise sur lames réalisée par les médecins pathologistes du service anatomie et cytologie pathologiques du Centre Hospitalier Sud Francilien (CHSF, Corbeil-Essonnes) peut, dans certains cas, constituer l’étape finale. Parmi les nombreux équipements et automates utilisés en routine par les membres de la plateforme d’histologie – anatomocytopathologie, un nouvel équipement a fait son apparition en fin d’année dernière : le Merscope Ultra (Vizgen). Financée par Genopole et accessible à l’ensemble de la communauté scientifique francilienne, cette technologie de transcriptomique spatiale dite MERFISH (Multiplexed Error-Robust Fluorescence in situ Hybridization) est une méthode avancée d’imagerie à molécule unique permettant de mesurer simultanément le nombre de copies et la distribution spatiale de plusieurs centaines d’espèces d’ARN dans des cellules individuelles, et cela à partir d'une coupe histologique (ou d'un tapis cellulaire issu d'une culture). Cette technologie utilise un système de codage combinatoire et des cycles d’imagerie séquentiels pour augmenter considérablement la capacité de multiplexage, permettant ainsi une résolution spatiale subcellulaire (résolution de 200 nm environ et précision de la position de l'ARN de 50 nm environ) et une détection sensible des gènes faiblement exprimés. Ainsi, le Merscope Ultra (Vizgen) permet de réaliser de la génomique spatiale in situ à haute résolution en cartographiant les transcrits sur une surface de 3 cm² en une seule lecture dont la durée est comprise entre 24 et 72 heures. Cet équipement permet une analyse efficace des échantillons avec une capacité de profilage allant jusqu’à 1000 gènes personnalisés à la résolution cellulaire. Cet équipement propose également deux tailles de cellules de flux offrant ainsi une plus grande flexibilité expérimentale. Si le Merscope Ultra (Vizgen) possède un débit inférieur à ceux de ses concurrents, son point fort est l’édition personnalisée des gènes recherchés. Ceci permet son utilisation sur tous types de tissus, qu’ils soient d’origine végétale ou humaine en passant par les tissus provenant du poisson-zèbre. De plus le protocole MERFISH est flexible et permet l'adaptabilité aux tissus ayant des propriétés particulières comme des coupes de cerveau de souris (cf. bandeau de photos), de rein, d’un lobe du foie ou des biopsies musculaires par exemples. Par conséquent, les domaines d’application de la technologie MERFISH sont vastes et variés. Cette technologie est particulièrement utile dans la recherche biomédicale pour l’étude des tissus complexes, tels que les échantillons tumoraux, où la compréhension de l’organisation spatiale des cellules est cruciale. Elle permet également d’explorer les interactions cellulaires et les états cellulaires dans leur contexte tissulaire natif. Pour le moment, de nombreuses applications dans le cancer et les neurosciences sont décrites dans la littérature et les panels de gènes préfabriqués couvrent principalement ces applications. Néanmoins, la possibilité de création de panels sur mesure permet d'étendre le champ des applications à tous types de problématique biologique. Enfin, la capacité de MERFISH à détecter des populations de cellules rares et à analyser des échantillons de tissus intacts sans dissociation ou microdissection tissulaire en fait un outil puissant pour les études de développement d'approches thérapeutiques telles que celles développées par certaines des entités du biocluster Genopole. -> Contact : Julien Picot (julien.picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme a déjà publié plusieurs FOCUS PLATEFORME, n’hésitez-pas à les relire… GENOPOLE / Plateforme d'histologie-anatomocytopathologie. La plateforme d'histologie-anatomocytopathologie offre des équipements et le savoir-faire associé nécessaires à la préparation des tissus, leurs colorations histologiques et/ou immunohistochimiques en vue de leur exploration par imagerie photonique. Cette plateforme s'appuie sur l'expertise de médecins anatomocytopathologistes du Centre Hospitalier Sud-Francilien pour la bonne réalisation de projets précliniques et cliniques. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées aux innovations thérapeutiques, approches translationnelles et à la biologie synthétique, Genopole réunit 78 entreprises, 16 laboratoires académiques, 23 plateformes et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : favoriser l’émergence et la croissance de sociétés de biotechnologie, le transfert d’innovations vers le secteur industriel, le développement de la recherche et l’enseignement supérieur en sciences de la vie. Genopole est un Groupement d’intérêt public principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes technologiques labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, contactez Julien Picot (julien.picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
July 6, 2025 11:41 AM
|
La plateforme de cristallisation située dans l’Institut de Biologie intégrative de la Cellule (I2BC, Gif-sur-Yvette, Institut Joliot) a évolué vers une plateforme de cristallographie (I2BC / Plateforme de cristallographie) et propose de nouvelles prestations ainsi qu’un soutien scientifique aux clients pour tous projets en biologie structurale. La cristallographie des protéines est l’approche principale pour caractériser la structure 3D de protéines liées à des candidats médicaments et pour déterminer les bases moléculaires de complexes entre les protéines solubles ou membranaires et leur partenaire (ADN, ARN, autre protéines, peptides, lipides, co-facteurs, ligands divers dont métaux). Initialement, la plateforme de cristallisation permettait de réaliser le criblage, l'analyse et l'optimisation automatisés des conditions de cristallisation des macromolécules solubles et membranaires. Dorénavant, la plateforme de cristallographie propose des prestations étendues jusqu’à la détermination et/ou l’analyse de la structure des protéines. Les étapes successives à réaliser à partir d’un échantillon de protéine d’intérêt purifié sont l’obtention des cristaux, l’enregistrement des clichés de diffraction au Synchrotron Soleil, le calcul des cartes de densité électronique, l’affinement du modèle atomique de la protéine ou du complexe d’intérêt et son analyse structurale. L'accent est mis sur l'obtention de modèles les plus précis pour l'analyse structurale la plus fructueuse. Un service AlphaFold avec l'analyse des modèles prédits est aussi disponible, s’appuyant sur les services mis en place à l’I2BC par la plateforme de bioinformatique intégrative BIOI2. La plateforme est équipée de plusieurs robots de pipetage de nanovolume (y compris la phase cubique lipidique pour les protéines membranaires) et de visualisation, qui permettent de cribler à grande échelle les conditions de cristallisation à deux températures (4°C et 19°C). Les étapes de routine en amont d’un cristal de protéine sont l’obtention d’un gène d’intérêt synthétique optimisé pour une expression dans des hôtes procaryotes, l’expression et la purification de la protéine recombinante étudiée. La plateforme peut conseiller les clients sur ces différentes étapes et une collaboration/prestation peut être envisagée suivant la complexité du projet. La plateforme de cristallographie de l’I2BC s’adresse à tous les scientifiques (des novices aux cristallographes expérimentés) : il faut noter que la qualité de l’échantillon protéique est un critère important ! L’échantillon doit être pur et vérifié sur un gel acrylamide dénaturant avant d’être cristallisé. N’hésitez pas à nous contacter ! -> Contacts : Solange Morera (solange.morera@i2bc.paris-saclay.fr) ou cristallographie@i2bc.paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme a déjà publié plusieurs FOCUS PLATEFORME ces dernières années. Les relire ? I2BC / Plateforme de cristallographie. La cristallographie des protéines est l’approche principale pour caractériser la structure 3D de protéines liées à des candidats médicaments et pour déterminer les bases moléculaires de complexes entre les protéines solubles ou membranaires et leur partenaire (ADN, ARN, autre protéines, peptides, lipides, co-facteurs, ligands divers dont métaux). Initialement, la plateforme de cristallisation permettait de réaliser le criblage, l'analyse et l'optimisation automatisés des conditions de cristallisation des macromolécules solubles et membranaires. Dorénavant, la plateforme de cristallographie propose des prestations jusqu’à la détermination et/ou l’analyse de la structure des protéines. Les étapes successives à réaliser à partir d’un échantillon de protéine d’intérêt purifié sont l’obtention des cristaux, l’enregistrement des clichés de diffraction au Synchrotron Soleil, le calcul des cartes de densité électronique, l’affinement du modèle atomique de la protéine ou du complexe d’intérêt et son analyse structurale. L'accent est mis sur l'obtention de modèles les plus précis pour l'analyse structurale la plus fructueuse. Un service AlphaFold avec l'analyse des modèles prédits est aussi disponible. La plateforme est équipée de plusieurs robots de pipetage de nanovolume (y compris la phase cubique lipidique pour les protéines membranaires) et de visualisation, qui permettent de cribler à grande échelle les conditions de cristallisation à deux températures (4°C et 19°C). Les étapes de routine en amont d’un cristal de protéine sont l’obtention d’un gène d’intérêt synthétique optimisé pour une expression dans des hôtes procaryotes, l’expression et la purification de la protéine recombinante étudiée. La plateforme peut conseiller les clients sur ces différentes étapes et une collaboration/prestation peut être envisagée suivant la complexité du projet. Cette plateforme fait partie du pôle des plateformes de Biologie Structurale de l’I2BC qui comprend six plateformes: l’expression de protéines dans des hôtes eucaryotes (Prot-Ex), la cryo-microscopie électronique (cryoEM), la cristallographie, la résonance magnétique nucléaire (RMN), l’interactions macromoléculaires (PIM) et AlphaRep. Chaque plateforme offre un éventail complet de méthodes pour les études biochimiques, biophysiques et structurales au niveau moléculaire. Chacune bénéficie de l'expertise de scientifiques et d'ingénieurs qui conseillent, soutiennent et forment à l'utilisation d'instruments de pointe et à l'analyse des données. Le pôle de biologie structurale travaille en étroite collaboration avec le synchrotron SOLEIL, situé à proximité. La plateforme de cristallographie fait également partie de l'infrastructure nationale en Biologie Structurale FRISBI (Frisbi.eu). A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.
|



 Your new post is loading...
Your new post is loading...