Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
October 16, 2020 5:33 PM
|
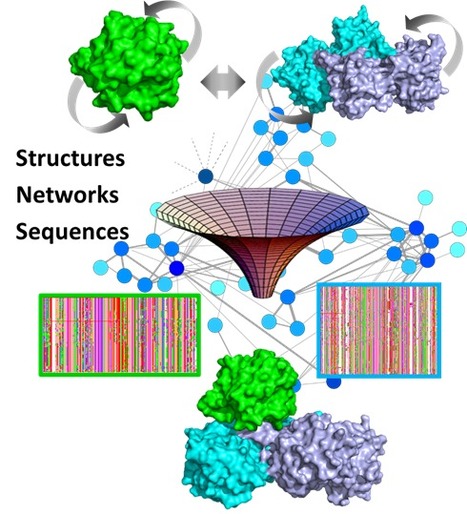
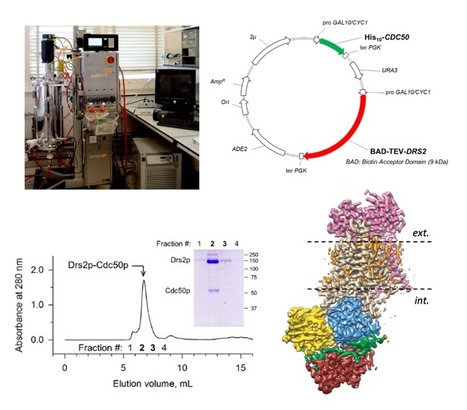
La réplication des génomes permet d’assurer la transmission fidèle de l’information génétique. Elle implique une orchestration fine des processus de réplication et de transcription des gènes pour éviter que des collisions entre machineries de réplication et de transcription ne provoquent des réarrangements chromosomiques, à l’origine de nombreux cancers et maladies génétiques. Récemment, une équipe du Centre de Recherche en Cancérologie de Marseille (CRCM, Inserm, CNRS, Aix-Marseille Université) a révélé que deux protéines, SLX4 et RTEL1, communiquaient entre elles pour éviter les conflits entre réplication et transcription, bien qu’aucun mécanisme moléculaire ne permette de comprendre leur mode d’action… Exemple choisi d’une collaboration fructueuse entre cette équipe marseillaise et le plateau technique de bioinformatique structurale (Institut de Biologie intégrative de la Cellule / Institut Joliot, CEA / Saclay, Gif-sur-Yvette), contacté pour aider à comprendre l’origine moléculaire de cette régulation stratégique ! Forte de son expertise, la plateforme a pris en charge l’analyse des séquences des deux protéines SLX4 et RTEL1, leur évolution dans différentes espèces de vertébrés et la prédiction de leur structure tridimensionnelle pour prédire leur mode d’assemblage. Un modèle structural 3D du complexe a ensuite été proposé et a permis de guider les expériences de mutagenèse et de biologie cellulaire qui ont, en retour, validé le modèle (Takedashi et al, Nat. Struct. Mol. Biol. 2020). De façon remarquable, des mutations de la protéine SLX4 retrouvées dans les cellules métastatiques de patients atteints de différents cancers, affectent l’interface du complexe formé par les deux protéines. De plus, le modèle 3D permet d’interpréter l’effet de mutations dans la protéine RTEL1 à l’origine du syndrome de Hoyeraal Hreidarsson, une pathologie rare apparentée aux maladies du vieillissement prématuré. Cette étude illustre l’intérêt des méthodes prédictives développées par la plateforme pour la modélisation des interactions protéine-protéine (Quignot et al, Nucl. Acids Res. 2018) dont les performances ont été soulignées à l’occasion du concours international de prédiction de structure 3D des interactions, CAPRI (équipe classée première en 2016 et 2019 (Lensink et al, Proteins 2020)). Contacts : Jessica Andreani (jessica.andreani@i2bc.paris-saclay.fr) & Raphaël Guerois (raphael.guerois@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
October 3, 2020 5:32 PM
|
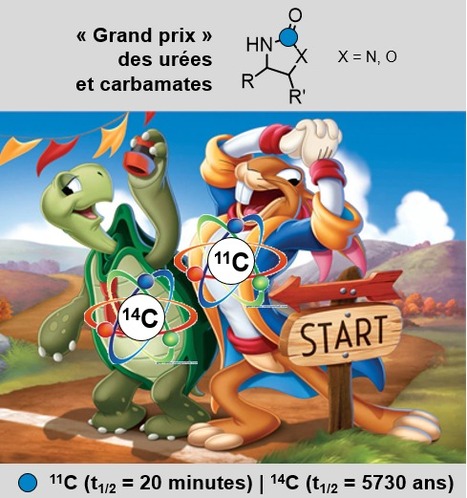
Ensemble, deux plateformes de l’Institut des Sciences du Vivant Frédéric Joliot (CEA, Centre de Saclay) – la plateforme de marquage isotopique (DMTS/SCBM, Saclay) et la plateforme de radiochimie (SHFJ/BIOMAPS, Orsay) – « unissent » des demi-vies extrêmes des radioisotopes du carbone qu’ils manipulent, et apportent la preuve de concept d’une stratégie « click » pour le marquage isotopique de molécules pharmaceutiques contenant des urées ou des carbamates ! Contexte et résultats : Le marquage de molécules organiques par des isotopes radioactifs du carbone est indispensable au développement de médicaments. D'un côté, le carbone-14 (14C), au temps de demi-vie long (5730 ans), est utilisé en préclinique pour étudier le métabolisme et la biodistribution des candidats médicaments. De l'autre, le carbone-11 (11C), au temps de demi-vie ultra-court (20 minutes) est exploité pour marquer des molécules utilisées pour l'imagerie moléculaire par tomographie par émission de positons. Bien que partageant des propriétés chimiques identiques, ces deux isotopes radioactifs présentent des propriétés physiques opposées, qui nécessitent généralement des stratégies différentes pour leur insertion dans des structures organiques. Or, étant donné la complémentarité de leur utilisation, des procédures de marquage communes seraient bénéfiques pour la R&D pharmaceutique. Dans les deux cas, la source de carbone est la même : le dioxyde de carbone (CO2). Le travail en synergie des deux plateformes a permis de réaliser le marquage par l'insertion directe du CO2 radioactif (11C ou 14C) dans des molécules organiques contenant des urées ou des carbamates cycliques, en une seule étape et en quelques minutes. Ces motifs moléculaires entrent dans la composition de nombreux produits pharmaceutiques, phytosanitaires ou encore de plastiques. En savoir plus ? Del Vecchio et al. Chem. Comm. 2020. La plateforme de marquage isotopique du CEA / Paris-Saclay (Institut Joliot, Département Médicaments et Technologies pour la Santé, Service de Chimie Bioorganique et de Marquage, Gif-sur-Yvette) est unique sur le territoire Paris-Saclay. Forte de son expertise dans la préparation (synthèse, contrôle de qualité) et formulation de molécules marqués, elle assure régulièrement des prestations et collaborations, académiques comme industrielles, dans le domaine du (radio)marquage moléculaire. Elle offre également à la demande, son expertise et environnement unique de travail (laboratoires « chauds », équipements dédiés) pour l’analyse et la caractérisation d’échantillons radioactifs : mesure de puretés chimique et radiochimique par HPLC, détermination d'enrichissements isotopiques et d'activités spécifiques par SM, analyse et détermination structurale par RMN liquide comme solide, mesure d’activités radioactives par comptage à scintillation. La plateforme de radiochimie du CEA / Paris-Saclay (Institut Joliot, SHFJ, BioMaps, Orsay) a pour vocation de synthétiser des radiotraceurs pour la recherche biomédicale, en particulier pour l’imagerie par tomographie d’émission de positons, à partir d’émetteurs de positons produits sur site (carbone-11, oxygène-15, -18) ou importés (par exemple -89). Contact : Davide Audisio (davide.audisio@cea.fr) et Fabien Caillé (fabien.caille@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ici et ici texte adapté de l’actualité scientifique parue sur le site web de l’Institut JOLIOT

|
Scooped by
Life Sciences UPSaclay
September 20, 2020 1:08 PM
|
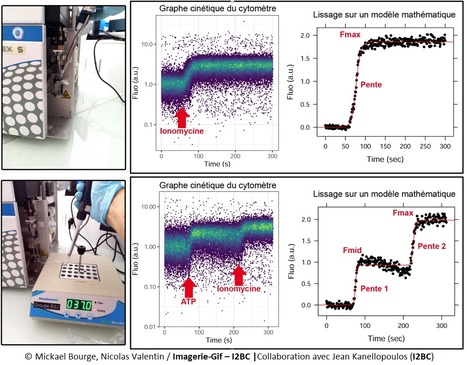
Les biosenseurs fluorescents sont des molécules qui, une fois introduites en cellules vivantes, rapportent un signal biologique spécifique mesurable par imagerie ou cytométrie. La sensibilité et le haut débit de la cytométrie en flux permettent de développer tout un panel d’analyses complémentaires essentielles à la compréhension du processus biologique observé. Les différences d’états physiologiques observées en imagerie alors peuvent être quantifiés en cytométrie. Plus encore, la cytométrie permet des analyses multiparamétriques offrant la possibilité de mesurer plusieurs activités simultanément cellule par cellule. Il est ainsi facile de comparer les résultats obtenus, d’en déduire leur interdépendance ou non, ce qui en fait un outil incontournable pour l’étude de la signalisation cellulaire. La plateforme de cytométrie en flux de l’I2BC (Institut de Biologie Intégrative de la Cellule) est localisée à Gif-sur-Yvette (campus CNRS) et réalise environ 300 prestations par an, pour des partenaires académiques comme privés. Récemment, la plateforme a utilisé son savoir-faire pour étudier en cellule unique de mammifère, les variations de flux calciques intracellulaires via le biosenseur fluorescent Fluo4. Cette sonde se compose d’un fluorophore de type fluorescéine combinée à une entité chélatante (BAPTA-like) spécifique de l’ion calcium ; son intensité de fluorescence est directement proportionnelle à la quantité de calcium présent dans le cytoplasme. Concrètement … Après acquisition de la ligne de base, l’opérateur ajoute directement dans l’échantillon étudié un ou plusieurs agonistes (ATP pour activer les récepteurs purinergiques et induire un flux calcique, ionomycine qui est un ionophore du calcium) et observe en temps réel les variations de fluorescence. Les données acquises sont ensuite analysées avec un script développé en langage R. Ce script permet : i) de faire les moyennes de fluorescence de la population de milliers de cellules chaque seconde, et ii) d’ajuster la courbe obtenue sur un modèle mathématique (dans le cas présent, une simple ou une double sigmoïde) afin d’en déterminer différentes constantes (fluorescences maximales de la première sigmoïde, Fmid ; fluorescence maximale de la seconde sigmoïde, Fmax ; pentes). Avec cette approche complète, il est alors possible de comparer des vitesses de variation de flux ioniques en fonction des agonistes/inhibiteurs utilisés, ou de comparer l’effet de différents environnements sur les réponses cellulaires étudiées. Cette méthode est utilisable avec tous types de cellules en suspension à température ambiante ou avec une plaque thermostatée (voir figure ci-dessus), et un biosenseur spécifique de l’espèce ionique à étudier. N’hésitez pas à nous contacter pour des développements d’approches utilisant des biosenseurs selon vos besoins et questions biologiques. I2BC / Plateforme de cytométrie (I2BC - plateformes IMAGERIE-GIF). Plus de 25 ans de service ! la plateforme réalise environ 300 prestations par an pour divers groupes de recherche appartenant à différents organismes de tutelles ou de sociétés privées. Les publications du service traduisent les nombreuses collaborations développées avec les différents instituts de la communauté Paris-Saclay ainsi qu’avec d’autres partenaires tels que l’IRD, le CIRAD, des universités françaises et étrangères. Son expérience polyvalente et son expertise en sondes fluorescentes permettent d’adapter la cytométrie à des projets très divers issus de laboratoires publics et privés. Son partenariat avec SPS (Labex Saclay Plant Sciences) contribue à une activité importante dans le domaine de la biologie végétale. La plateforme de cytométrie en flux propose la mesure de fluorescence d'un ou plusieurs (> 10) fluorochromes simultanément, cellule par cellule. Cette technologie permet d'étudier: i) le dosage de la quantité d’ADN nucléaire en vue de l’étude de cycles cellulaires et d’endoréplication, ii) le dosage d’ADN à des fins de recherche en écologie et systématique, et d’amélioration des variétés (analyse de ploïdies), iii) le suivi de l’activité génique par l’expression d’un ou plusieurs gènes rapporteurs (tels que celui de la "Green Fluorescent Protein"-GFP et autres protéines fluorescentes), iv) des mesures d’activités métaboliques de la cellule (biosenseurs): dosage de calcium, pH, potentiel membranaire, poussées oxydatives, glutathion..., v) des analyses immunologiques, vi) le tri de cellules animales, de levures, de bactéries, de protoplastes et d’organites cellulaires. Contact : Mickael Bourge (mickael.bourge@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
September 6, 2020 12:38 PM
|
Une nouvelle méthode spectroscopique, le tr-SRD (dichroïsme circulaire avec le rayonnement synchrotron résolu en temps) est maintenant disponible sur la ligne de lumière DISCO de SOLEIL (SYNCHROTRON SOLEIL/DISCO-SRCD) ! Grâce à la polarisation naturelle du rayonnement synchrotron, cette méthode permet l’acquisition en temps réel de la différence de l’absorption de la lumière polarisée circulairement (gauche et droite) via une mesure simultanée des deux faisceaux polarisés. Dorénavant, il est possible de suivre les dynamiques des macromolécules biologiques (protéines, ADN, ARN et leurs complexes respectifs) à des échelles de temps allant de la seconde à la micro seconde et dans une gamme spectrale (170-350 nm) inaccessibles, jusqu’à présent, en spectroscopie CD conventionnelle. Pourquoi ce nouveau développement ? Les méthodes biophysiques classiques telles que la fluorescence, l'absorbance, la spectroscopie infrarouge, la spectroscopie Raman et la résonance paramagnétique électronique sont toutes précieuses pour étudier la cinétique macromoléculaire telle que le repliement des protéines, la formation de liaisons avec des ligands et les réactions de polymérisation. Le tr-SRD qu’offre maintenant la plateforme SYNCHROTRON SOLEIL/DISCO-SRCD sera complémentaire à ces méthodes, mettant en évidence la dynamique des chromophores telle que les liens peptidiques ou plus complexes des nucléotides. Pour induire un changement structurel cinétique ou dynamique, un mélange rapide (« stopped-flow »), des élévations rapides de température (saut en T) ou des réactions photo-induites pour des échantillons photosensibles sont mis à la disposition des utilisateurs. Un exemple concret d’utilisation ? Le nouveau dispositif du tr-SRD a été testé pour la première fois en suivant l’isomérisation cis-trans induite par la lumière (photo-induction) sur un peptide FK-11-X composé d’un azobenzène lié par ses deux extrémités à la chaîne polypeptidique (illustration ci-dessus, avec la représentation d’un spectre toutes les 70 ms). Il a permis de suivre la formation de l'hélice a ainsi que son dépliement. Les résultats démontrent que l'hélice sous les contraintes du lieur azobenzène se replie en 2,5 secondes, tandis que son dépliement dure moins d'une seconde. Ces premiers résultats confirment la faisabilité de cette nouvelle méthode et ouvrent la voie à un large éventail d'investigations biophysiques des biomolécules. Ainsi, dans une approche multidisciplinaire, le tr-SRD servira parfaitement des techniques structurales à haute résolution telles que la diffraction et diffusion des rayons X ou encore la diffusion de neutrons et s’intègre dans le panel des techniques pour la biologie intégrative (Integrated Biology). En savoir plus ? Plug In Labs Université Paris-Saclay : cliquer ici Contact : Frank Wien (frank.wien@synchrotron-soleil.fr) SYNCHROTRON SOLEIL/DISCO-SRCD. DISCO est une ligne de lumière couvrant la gamme VUV-visible, dédiée à la biologie et la chimie. DISCO est composée de trois stations expérimentales, celle décrite ici (SRCD) concerne le dichroïsme circulaire (CD) utilisant le rayonnement synchrotron (SR), permettant de descendre jusqu’à 120 nm sur films. En solution, on peut travailler en tampons très absorbants. De plus le CD dispersif permet d’enregistrer des spectres CD à la ms.

|
Scooped by
Life Sciences UPSaclay
August 23, 2020 11:48 AM
|
Depuis décembre 2019, le virus SARS-CoV-2, responsable de l’infection COVID-19, génère une pandémie sans précédent et a mis notre planète à l’arrêt. Les réactions qu’il provoque chez les individus sont différentes. Certaines personnes ne se rendront pas compte qu’elles ont été ou qu’elles sont infectées, certaines présenteront des symptômes légers, alors que d’autres seront victimes de complications sévères d’infections allant du syndrome respiratoire aigu sévère au dysfonctionnement d’organes multiples conduisant à la mort. La plupart des individus souffrant de cas sévères d’infections présenteraient des comorbidités dont l’âge et certaines maladies chroniques. Néanmoins, il a aussi été observé la survenue de formes graves chez des individus plus jeunes sans facteur de risque. Le microbiote intestinal jouerait-il un rôle dans cette infection et la réaction immunitaire ? Pourrait-il, par exemple, être à l’origine de la différence de sévérité d’infections entre individus et les formes sévères chez des individus jeunes sans comorbidités ? Ou encore, serait-il possible de prédire les formes sévères de COVID-19 à partir du microbiote intestinal ? Joël Doré, directeur de recherche à l’Institut Micalis (Microbiologie de l'Alimentation au Service de la Santé) et directeur scientifique de la plateforme MetaGenoPolis au centre Île-de-France – Jouy-en-Josas de l’INRAE, et Harry Sokol, Professeur hépato-gastro-entérologue à l’hôpital Saint Antoine de Paris nous partagent le fruit de leurs réflexions dans une courte synthèse sur le microbiote intestinal et son implication potentielle dans cette crise sanitaire liée au COVID-19. S’il est en effet impossible de conclure à ce jour sur le rôle exact du microbiote intestinal dans le processus d’infection, une hypothèse serait qu’il pourrait être impliqué dans la différence de sévérité de l’infection COVID-19 entre individus et les formes sévères chez des individus jeunes sans comorbidités, notamment par la relation qu’il entretient avec son hôte en influençant l’homéostasie immunitaire. Le système immunitaire est en effet relié au microbiote intestinal. Lorsque celui-ci est équilibré, l’homéostasie immunitaire est favorisée. Dans le cas de l'obésité, hypertension et du diabète, qui sont des facteurs de risque reconnus pour les infections sévères COVID-19, cette relation symbiotique hôte-microbiote est altérée, engendrant une perte de la diversité bactérienne, une réduction de la perméabilité de la barrière intestinale, une augmentation de bactéries pathogènes et un état inflammatoire. Aussi, il est bien possible que certaines bactéries du microbiote intestinal jouent un rôle sur la susceptibilité à l'infection COVID-19, même si pour l'instant, aucune donnée solide n’existe pour incriminer une bactérie en particulier. Plusieurs initiatives nationales ont déjà vu le jour pour mieux comprendre l’implication du microbiote intestinal dans l’infection COVID-19. Elles devraient permettre prochainement d’apporter des éléments de réponses à ces questions. En savoir plus ? MetaGenoPolis (MGP) est un centre INRAE expert en recherche sur le microbiome intestinal appliquée à la santé et à la nutrition de l’homme et de l’animal. En collaboration avec les industries, les universités et les cliniques, MGP conçoit et met en œuvre des projets adaptés aux besoins de ses partenaires. Certifiés ISO 9001, les protocoles et procédures mis en œuvre sont constamment maintenus à la pointe de la technologie. MGP propose de la métagénomique quantitative et fonctionnelle pour explorer le lien entre le microbiome, nutrition et santé. MGP offre des services d’analyse du microbiome de bout en bout, y compris des recommandations personnalisées sur la collecte d’échantillons, la mise en banque d’échantillons, l’extraction d’ADN, la métagénomique quantitative et fonctionnelle, les grandes installations de stockage de données et de calcul, la bioinformatique, l’analyse statistique et l’interprétation des données. Une des ambitions de MGP est de constituer via un projet de science citoyenne une base de données publique regroupant les microbiomes de 100 000 individus français dont un des objectifs sera de mieux comprendre l’hétérogénéité des microbiomes intestinaux de français sains. Contact : Alexandre Cavezza, Florence Haimet (contact@mgps.eu) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
July 19, 2020 4:37 PM
|
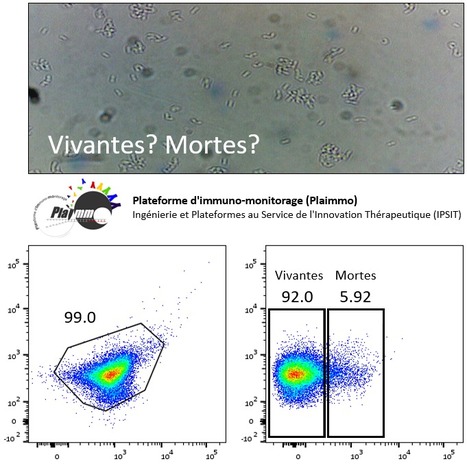
L’UMS IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) compte à ce jour 10 plateformes dont PLAIMMO (PLAteforme d'IMmuno-MOnitorage) ! Son personnel est votre disposition, équipe académique comme industrielle, pour vous aider à la mise au point de techniques utilisables en cytométrie en flux. Démonstration via trois exemples choisis ! Quantifier les bactéries par cytométrie en flux ? Dans le contexte d’un projet collaboratif sur le rôle plus ou moins délétère du microbiote dans les pathologies hépato-gastrointestinale, un enseignant-chercheur de l’Université de Paris Sud (Vanessa Liévin-Le Moal, UMR-S 996) nous a sollicité pour mettre au point la quantification de bactéries par cytométrie en flux. Première difficulté : comme le marquage bactérien nécessite une étape de fixation et de perméabilisation, les méthodes habituelles utilisant l'iodure de propidium ne peuvent être utilisées ; PLAIMMO innove et développe à façon une nouvelle méthode utilisant des marqueurs de viabilité pouvant être fixés. Deuxième difficulté : les bactéries (cellules de petite taille) se différencient très mal du bruit de fond électronique généré lors du passage des événements sur le cytomètre ; PLAIMMO met à profit son expertise et développe une méthode donnant des résultats satisfaisants, et actuellement en cours de validation pour être combiner avec d’autres marquages (illustration ci-dessus). La suite ? utiliser ces technologies pour confirmer, sur bactéries vivantes, et selon les traitements effectués, le niveau d’expression d’une protéine d’intérêt identifiée dans l’effet protecteur du foie en présence d’alcool. A suivre donc ! Doser des cytokines et chimiokines dans des volumes faibles ? Dans le cadre d’un second projet, industriel cette fois, la plateforme a été sollicitée par un chercheur (Francine Joly, SEPHRA PHARMA) pour développer le dosage de cytokines et chimiokines sur un volume récolté très limitant (maximum 250 µL) de ses échantillons. Dans ces conditions, il était impossible de faire des ELISA et l’utilisation de la technique LUMINEX™ n'était pas compatible avec le budget du projet. PLAIMMO utilise son savoir-faire et propose au porteur la technique des Cytokines Beads Assay ! PLAIMMO l’accompagnera aussi dans la mise en place et réalisation du protocole. En savoir plus sur ces techniques ? (Lehmann et al, J. Vis. Exp. 2017). Détecter des ARN par cytométrie de flux ? Plus récemment, la plateforme, fort de l’intérêt des techniques développées par INVITROGEN sur la détection des ARN par cytométrie en flux (PrimeFlow), a mis en place une stratégie d'étude et de validation en interne de ces techniques en commençant par des tests avec des gènes de ménage. Cela nous a permis de repérer les étapes problématiques et surtout de conseiller au mieux les utilisateurs de PLAIMMO désirant utiliser cette technique. Le principe du PrimeFlow est l’hybridation spécifique sur l’ARN d’intérêt d‘une paire d’oligonucléotides adjacente. Sur cette paire se fixe ensuite une molécule de pré amplification permettant la fixation de molécules d’amplification et ensuite de sondes marquées par fluorescence. Un des points les plus cruciaux est la température de fixation de ces sondes ! La plateforme de cytométrie en flux (PLAIMMO) de l'Unité Mixte de Service – Ingénierie et Plateformes au Service de l’Innovation Thérapeutique (UMS-IPSIT) située sur le site de Clamart offre régulièrement ses services aux équipes de recherche académiques du territoire Paris-Saclay ainsi qu'aux industriels. Le personnel de la plateforme est à votre disposition pour vous aider à pour la réalisation de projets de recherche fondamentale, préclinique sur des modèles expérimentaux ainsi que pour des protocoles de recherche clinique. Son personnel est aussi à votre service pour la mise au point de nouvelles techniques utilisant la cytométrie en flux. Les équipements de cytométrie en flux de la plateforme permettent le phénotypage des cellules par la détection de molécules membranaires et intracellulaires (biomarqueurs) mais aussi des études fonctionnelles telles que la détection de phosphorylation des protéines, la prolifération cellulaire, la quantification de cytokines ou chimiokines excrétées ou la détection d'ARN. Enfin, des tris cellulaires à haut débit sont aussi proposés par la plateforme. La plateforme est également équipée pour mesurer l'expression de gènes grâce à la PCR quantitative en temps réel. Nos activités qui peuvent être en relation avec celles d'autres plateformes, permettent l'identification de nouveaux biomarqueurs qui peuvent être des cibles thérapeutiques. Contact : Marie-Laure Aknin (marie-laure.aknin@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
July 5, 2020 1:00 PM
|
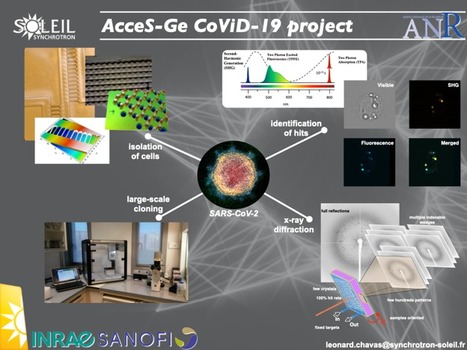
La plateforme de cristallographie des protéines PROXIMA-1 se trouve sur le site de Synchrotron SOLEIL sur le plateau de Saclay à Saint-Aubin. Elle met à disposition les technologies d’analyse structurale des macromolécules par diffraction aux rayons X. La plateforme fonctionne selon plusieurs modalités d'accès de manière à satisfaire l'ensemble des besoins des utilisateurs académiques et industriels, tel que dicté par le centre de recherche Synchrotron SOLEIL. Dans la lutte contre le SARS-Cov-2, la biologie structurale peut apporter des éléments d’études clés dans les développements pharmaceutiques. Dans ce cadre, PROXIMA-1 a initié un projet de génomique structurale accélérée sur le virus (AcceS-G CoViD-19). Ce projet collaboratif implique plusieurs équipes de Synchrotron SOLEIL (laboratoire de Biologie notamment) ainsi qu’une autre de ses plateformes (DISCO), mais aussi des équipes de l’UMR 0892 (VIM, Virologie et Immunologie Moléculaire, INRAe/UVSQ) et un partenaire industriel, SANOFI. En combinant des technologies de pointe dans un workflow unique, les chercheurs vont tenter d’appliquer les approches de croissances cristallines in vivo pour cribler toutes les protéines codées par le génome du pathogène responsable de CoViD-19. Les protéines étant exprimées dans leur environnement naturel, la méthodologie appliquée a le double avantage de permettre un gain de temps dans l’obtention éventuelle de cristaux sans avoir à passer par les étapes classiques de purification et de criblage in vitro, et de conserver les modifications post-translationnelles importantes dans la fonction de ces protéines. La plateforme ainsi développée pour ces criblages in vivo bénéficie de l’implémentation de technologies avancées telles que des automations de manipulations par le biais de robots et de systèmes microfluidiques ; le couplage à des systèmes de visualisation à microscopie bi-photonique ; ou encore des méthodes sérielles de collectes de données aux rayons X. Dès l’obtention de résultats, ceux-ci seront soumis pour publication dans des archives ouvertes afin que la communauté scientifique puisse rapidement progresser sur les traitements à développer pour contrer ce nouveau virus. Contact : Léonard Chavas (leonard.chavas@synchrotron-soleil.fr) Plug In Labs Université Paris-Saclay : cliquer ici SYNCHROTRON SOLEIL / PX1. PROXIMA-1 (PX1) participe à la biologie structurale intégrative installée à SOLEIL au sein de la section scientifique de biologie et santé, HelioBiology. Opérationnelle depuis Mars 2008, PROXIMA-1 est spécialisée dans les mesures de données bio-cristallographiques sur des macromolécules, au même titre que PROXIMA-2A. Elle offre un faisceau de lumière adapté et optimisé pour des études de diffractions aux rayons x de cristaux de molécules biologiques de grande taille, à hautes résolutions spectrales et pour des échantillons particulièrement sensibles aux dommages de radiations. La ligne est équipée d'un détecteur de rayons x à lecture rapide et de grande surface ; en combinaison avec un robot permettant des échanges automatisés des échantillons, cette configuration permet d'étudier un très grand nombre de cristaux dans un temps réduit. Des stratégies avancées de collectes de données sont disponibles de façon systématique en prenant avantage d'un goniomètre à géométrie 3-cercles de dernière génération. ____________________________________ https://www.synchrotron-soleil.fr/fr/lignes-de-lumiere/proxima-1 https://www.pluginlabs-universiteparissaclay.fr/fr/entity/915158-synchrotron-soleil-px1

|
Scooped by
Life Sciences UPSaclay
June 21, 2020 1:00 PM
|
La plateforme INRAE/Infectiologie Expérimentale des Rongeurs et Poissons (IERP) propose des services d’infectiologie in vivo sur les espèces poissons et rongeurs. Elle est rattachée à l’Institut des Sciences Animales Paris Saclay et est constituante de l’infrastructure nationale EMERG’IN. Le contrôle du statut sanitaire des animaleries est dans ce contexte un point clé pour assurer et optimiser l’hébergement des animaux d’élevage et d’expérimentation dans les meilleures conditions. Son maintien passe par l’établissement de barrières et le développement d’outils de diagnostic performants. Exemple choisi de développement : La plateforme, en collaboration avec l’unité VIM (INRAE) et l’institut Pasteur est aujourd’hui partenaire du projet Specific Pathogen Free zebrafish for Immunology and Inflammation Research (SPFI2R). Ce projet, soutenu par le département de Santé Animale de l’INRAE, l’infrastructure nationale EMERG’IN, le GIS IBISA et le DIM1health, a pour but le développement de poisson-zèbres, mais exempt d’organisme pathogène spécifique (EOPS +) ; ceci afin de (1) limiter les défauts de standardisation des modèles expérimentaux, infectieux ou non et (2) réduire l’impact des infections discrètes sur les processus biologiques étudiés (inflammation, infections, métabolisme, développement). Ce projet s’inscrit dans la continuité des activités de la plateforme visant à développer des modèles poisson « propre » pour l’étude de pathologies infectieuses humaines et animale, mais vise aussi à poursuivre l’amélioration des conditions d’élevage des poissons d’expérimentation pour la fourniture d’animaux plus standardisés et exempts de pathogènes environnementaux. Le défi zootechnique que se propose de relever la plateforme consiste en l’intégration des différentes étapes de production en conditions stériles puis contrôlées : de la stérilisation des œufs à la création de poisson à flore contrôlée. Le développement d’outils de suivi s’opère en parallèle par la création d’outils de diagnostics moléculaires, de tests microbiologiques et de nouvelles approches d’histopathologie. A court terme, le projet permettra de mesurer l’impact de ces conditions d’élevage standardisées sur la production de lignées d’intérêt (sauvages, mutantes, immuno-déficientes) et sur des modèles expérimentaux en infectiologie, microbiologie ou métabolisme. L’expertise en imagerie de la plateforme bénéficiera à ce projet pour évaluer l’implantation de populations bactériennes sélectionnées et mener des approches d’histopathologie en 3 dimensions, innovantes sur tissus intègres transparisés. La disponibilité de ce poisson-zèbre EOPS ? Si tout se passe comme prévu : Septembre 2021 ! En savoir plus ? La plateforme INRAE / Infectiologie Expérimentale des Rongeurs et Poissons (IERP) a pour principales missions la production d’animaux à statut sanitaire et génétique défini, la réalisation in vivo d’expérimentation en infectiologie et le phénotypage des animaux infectés par imagerie. Dans le cadre de projets collaboratifs ou de prestations de service, l'infrastructure mène des projets en infectiologie, immunologie, vaccinologie, génétique, physiologie et microbiologie. Distribuée dans plusieurs bâtiments du campus, notre infrastructure réalise des projets multi-espèces au sein d’une animalerie « rongeurs » pour les modèles souris et rat et d’une pisciculture expérimentale hébergeant les espèces aquacoles (truite et carpe) et poisson-zèbre. Chaque installation dispose de laboratoires de confinements (BSL2/3) équipés de technologies avancées (IVIS, bi-photon) pour le suivi par imagerie des animaux infectés. Contact : Dimitri Rigaudeau (dimitri.rigaudeau@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
June 7, 2020 11:24 AM
|
L’agriculture devient moteur de l’économie circulaire en utilisant de nouveaux produits organiques pour nourrir les cultures. La quantité et la diversité des produits organiques recyclés en agriculture augmentent avec le renforcement des pratiques de valorisation des déchets et la multiplication des procédés industriels et agro-industriels mis en œuvre. Ces pratiques cohabitent avec le recours aux engrais de synthèse depuis que le procédé Haber Bosh a été mis au point au milieu du XIXème siècle. Mais les consommateurs ne veulent plus être soumis aux adversités liées à leur utilisation. Il est primordial de préserver la qualité de l’air en réduisant les pertes vers l’environnement tout en conservant l’efficience des intrants azotés. Dans ce contexte, la maîtrise des émissions d’ammoniac (NH3) vers l’atmosphère est une préoccupation majeure à l’échelle internationale (Protocole de Göteborg) et nationale (PREPA) et un challenge que cherchent à relever aussi les industriels de la fertilisation, les coopératives agricoles ou des entreprises de biotechnologie. Ils rivalisent d’ingéniosité pour proposer des additifs ou formuler des engrais plus respectueux de l’environnement et ont besoin de quantifier les niveaux d’abattement obtenus. Ce défi, l’UMR ECOSYS a voulu les aider à le relever. Sur la base de compétences spécifiques développées dans la métrologie des émissions gazeuses au champ et en laboratoire, scientifiques, ingénieurs et techniciens de l’unité ont développé des outils et systèmes de mesure permettant de combler un déficit constaté à l’international. ECOSYS a, par exemple, développé la plateforme « ECOSYS / Dispositif de caractérisation du potentiel d'émission d'ammoniac » pour mesurer de manière semi-automatique la capacité des sols ou des engrais à émettre de l‘ammoniac. Des cellules de volatilisation ont été spécifiquement conçues et leur design a été prototypé par simulation numérique puis évalué expérimentalement pour optimiser les processus d’échange. Le dispositif se rapproche au plus près des conditions de la pratique agricole, tout en optimisant les conditions ambiantes (température, humidité de l’air et du sol…) et d’échange (débit, volume de tête, type de balayage…), sur une durée de mesure de quelques jours à quelques semaines selon les produits. En constante évolution vers plus de performances, plusieurs versions ont été mobilisées depuis 2002 (Le cadre et al, 2005) dans des projets variés, impliquant des organismes de développement agricole comme des industriels de la fertilisation ou du recyclage, ou des agro-industries. Ce dispositif a été utilisé pour affiner la mise au point industrielle de solutions azotées et de granules d’engrais formulés pour être moins émissifs. Les projets EMITEC (Espagnol et al, 2015), Efac-Raclage-V (Lagadec et al, 2019) et BTéP (Edouard et al, 2019) ont permis quant à eux d’évaluer les effets des inflexions des conduites des troupeaux et des aménagements des bâtiments d’élevage pour réduire la volatilisation sur l’ensemble de la filière de gestion des effluents, incluant les bâtiments d’élevage, différents procédés de stockage, et les épandages, tant pour les élevages porcins que pour les élevages bovins laitiers. Avec le projet ProLab, nous avons commencé à mettre au point la démarche du passage des mesures de laboratoire aux volatilisations en condition de la pratique agricole, pour des produits résiduaires organiques issus des différentes filières : élevage, agro-industrie, collectivités, assainissement. Par ailleurs, la demande croissante a conduit à externaliser et professionnaliser l’offre : la création d’une ligne de services, INRAE Transfert Environnement, adossée à l’UMR ECOSYS, permet de proposer ces mesures auprès des acteurs économiques et a conduit au recrutement de 2 ingénieurs en moins de 2 ans. La plateforme ECOSYS / Dispositif de caractérisation du potentiel d'émission d'ammoniac se trouve à Grignon dans l’UMR ECOSYS INRAE (Institut national de recherche pour l’agriculture, l’alimentation et l’environnement). En s’appuyant sur plus de 20 ans de compétences en métrologie des émissions gazeuses, l’UMR ECOSYS a mis en place ce dispositif et propose des mesures de potentiels d’émissions d’ammoniac (NH3) en conditions contrôlées au laboratoire. Il permet d’étudier et de quantifier la capacité des sols, des engrais minéraux commerciaux ou en développement et des produits résiduaires organiques de toutes origines à émettre de l'ammoniac. Ce dispositif a été conçu pour se rapprocher au plus près des conditions de terrain. Contact : Céline Décuq (Celine.Decuq@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
May 24, 2020 1:28 PM
|
Tout le monde en parle, études à l’appui : la consommation de fibres diverses est liée à une meilleure santé … Mais encore ? Il existe aujourd’hui un déficit en fibres dans notre alimentation aussi bien chez les adultes que les enfants, ce qui impacte négativement notre microbiote intestinal, pour qui ses fibres constituent un combustible important. La diversité bactérienne de notre microbiote diminue et nous sommes donc encourager à consommer plus de fibres et en plus grande variété. Contacté par la société Bridor en 2016, Joël Doré, directeur de recherche à l’unité Micalis (Microbiologie de l'Alimentation au Service de la Santé) et directeur scientifique de la plateforme MetaGenoPolis au centre Île-de-France – Jouy-en-Josas de l’INRAE, a mené avec ses équipes plusieurs années de recherche pour élaborer un mélange de 7 fibres végétales bénéfiques pour le microbiote. Ces recherches ont conduit au développement et d’un nouveau produit, « Amibiote », une baguette permettant d’enrichir l’apport en fibres des consommateurs en qualité comme en quantité. Outre la diversité en fibres apportées, ce produit contient 11 grammes de fibres, pour 100 grammes de pain (dont plus de 50% sous forme soluble), contre 4 grammes pour une baguette traditionnelle. Cette baguette est disponible depuis le 7 octobre 2019 dans près de 1000 points de vente et supermarchés ! De plus, une étude clinique dirigée par le CRNH (Centre de Recherche en Nutrition Humaine) Rhône-Alpes, sur 40 volontaires a montré que cette baguette était capable d’améliorer la composition du microbiote, de diminuer le taux de cholestérol et d’agir sur l’insulino-résistance. Un tel produit dont les effets sur la santé ont maintenant été prouvés permet d’espérer un réel impact sociétal ! Bonne dégustation ! MetaGenoPolis (MGP) est un centre INRAE expert en recherche sur le microbiome intestinal appliquée à la santé et à la nutrition de l’homme et de l’animal. En collaboration avec les industries, les universités et les cliniques, MGP conçoit et met en œuvre des projets adaptés aux besoins de ses partenaires. Certifiés ISO 9001, les protocoles et procédures mis en œuvre sont constamment maintenus à la pointe de la technologie. MGP propose de la métagénomique quantitative et fonctionnelle pour explorer le lien entre le microbiome, nutrition et santé. MGP offre des services d’analyse du microbiome de bout en bout, y compris des recommandations personnalisées sur la collecte d’échantillons, la mise en banque d’échantillons, l’extraction d’ADN, la métagénomique quantitative et fonctionnelle, les grandes installations de stockage de données et de calcul, la bioinformatique, l’analyse statistique et l’interprétation des données. Une des ambitions de MGP est de constituer via un projet de science citoyenne une base de données publique regroupant les microbiomes de 100 000 individus français dont un des objectifs sera de mieux comprendre l’hétérogénéité des microbiomes intestinaux de français sains. Contact : contact@mgps.eu Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
May 10, 2020 5:50 PM
|
La plateforme NGS (Next-Generation-Sequencing) et analyses génomiques est une plateforme collaborative ouverte aux équipes académiques d’Ile-de-France. Créée en 2014 au sein de l’Institut des cellules Souches pour le Traitement et l’Étude des Maladies monogéniques (I-Stem), cette plateforme a été récemment labellisée Genopole et offre un ensemble de techniques indispensables aux analyses transcriptomiques ou génomiques. A sa création, le choix du séquenceur Ion Proton (ThermoFisher) qui permettait de séquencer des panels de gènes ainsi que des petits génomes correspondait aux projets développés par les équipes d’I-Stem ou de collaborations déjà établies (Darville et al, EBioMedicine 2016 ; Cambon et al, Molecular therapy. Methods & clinical development 2017 ; Galvan et al, Brain 2018). Cette technologie a notamment permis d’identifier simultanément un grand nombre de gènes pouvant être modulés au cours d’un processus de différenciation cellulaire dans un contexte physiologique ou pathologique. Récemment, la plateforme a aussi participé à l’évaluation du potentiel thérapeutique d’un composé pour le traitement de la dystrophie musculaire des ceintures (LGMD2D) en collaboration avec l’équipe de Xavier Nissan (Hoch et al, Sci Rep. 2019). Identifié à l’issu d’un criblage à haut-débit, le Thiostrepton permet de restaurer la bonne localisation d’une protéine mutée, l’alpha-sarcoglycane, indispensable à l’intégrité des sarcomères des muscles striés. L’analyse du transcriptome de cellules traitées a permis d’étudier plus en détail le mécanisme d’action du Thiostrepton. En parallèle de projets de prestation, NGS (Next-Generation-Sequencing) et analyses génomiques développe également de nouveaux outils de séquençage ou d’analyses bio-informatiques, comme par exemple, une base de données d’expression génique qui permettra d’étudier l’expression d’un ou de plusieurs gènes dans différents modèles cellulaires dérivés de cellules souches pluripotentes humaines. Enfin, afin de pouvoir répondre aux besoins croissants des équipes académiques génopolitaines, NGS (Next-Generation-Sequencing) et analyses génomiques s’est récemment dotée d’un séquenceur NextSeq 550 (Illumina) grâce au soutien de Genopole, de l’Université d’Evry Paris-Saclay et de I-Stem. Ce nouveau séquenceur a été installé le 2 janvier dernier, est depuis pleinement opérationnel et permet de multiplier les capacités de séquençage de la plateforme par un facteur 4. Le NextSeq 550 (Illumina) est en effet le premier séquenceur de la gamme Illumina à permettre le séquençage 30X d’un génome humain (environ 3 Gb) ou de 65 génomes bactériens de type E. Coli (5 Mb) en à peine 24 heures. Proposant deux supports de séquençage, respectivement de 140 ou 400 millions de reads, le NextSeq 550 offre ainsi la possibilité d’adapter le débit de séquençage à chaque projet de recherche. La plateforme NGS (Next-Generation-Sequencing) et analyses génomiques propose aux équipes de recherche académique des prestations de séquençage à haut débit du génome et/ou du transcriptome ainsi qu’un accompagnement scientifique et technique en proposant des stratégies de séquençage les plus adaptées aux projets. A l’issue du séquençage, les données brutes sont analysées selon les applications puis transférées au responsable du projet. En plus de l’activité de prestation, la plate-forme conduit des programmes de R&D telles que la mise au point de nouvelles techniques de séquençage ainsi que la mise en place d’outils bio-informatiques (bases de données / pipelines d’analyse NGS). Contact : Margot JARRIGE (mjarrige@istem.fr) Plug In Labs Université Paris-Saclay : cliquer ici À propos de Genopole. Biocluster français dédié à la recherche en génétique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 83 entreprises de biotechnologies, 16 laboratoires de recherche, 25 plates-formes technologiques, ainsi que des formations universitaires (Université d’Évry Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologie et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon.

|
Scooped by
Life Sciences UPSaclay
April 26, 2020 5:21 PM
|
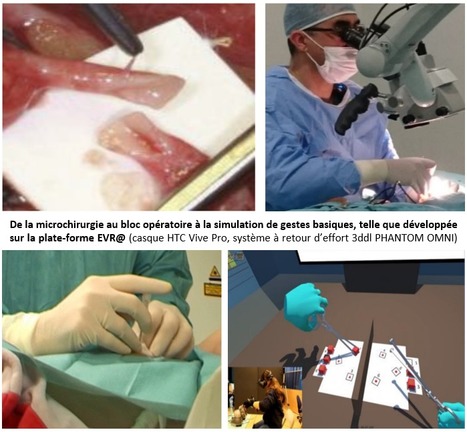
L’apprentissage des compétences chirurgicales à travers la simulation est une réelle nécessité afin de pouvoir répondre aux recommandations de la Haute Autorité de : « jamais la première fois sur un patient ». Dans ce contexte, les simulateurs virtuels ont déjà démontré leur efficacité pour la formation des chirurgiens ; cependant leur développement est délicat et relativement coûteux, ce qui empêche leur adoption comme outils standards de formation pour un grand nombre d’apprenants. Par conséquent, il semble primordial de centrer l’utilisation de ces simulateurs sur des pédagogies innovantes tout en impliquant les professionnels de santé tout au long du processus de conception. La littérature récente suggère que la formation médicale à travers la décomposition d’une procédure en microtâches basiques favoriserait l’apprentissage des gestes médicaux. Une telle approche simplifierait le développement de ces simulateurs virtuels et réduirait potentiellement leur coût. Dans ce contexte, la Plate-forme d’Environnement virtuel et de réalité augmentée (Evr@), labellisée Genopole et hébergée au sein du laboratoire Informatique, BioInformatique et Systèmes Complexes (IBISC) de l’Université d’Évry Paris-Saclay, développe depuis 2019 les bases matérielles et logicielles de deux simulateurs virtuels pour l’apprentissage de gestes médicaux, à partir des compétences accumulées à partir du projet VR Skills Lab (Laboratoire virtuel pour la formation des compétences médicales), initié en 2015 par Amine Chellali (MCF Univ. Evry, IBISC) [voir aussi Ricca et al, 2018; Ricca et al, 2017, Ricca et al, 2016; Benyahia et al, 2015]. Le premier simulateur concerne la formation aux gestes techniques en microchirurgie et le second la formation aux gestes d’insertion d’aiguille guidée par échographie en anesthésie. Les prochains mois seront ainsi consacrés à l’évaluation des deux simulateurs par les experts du Centre Hospitalier Sud Francilien (CHSF) avec des premiers prototypes fonctionnels attendus pour 2021. Ceci permettra de démontrer leur efficacité pour la formation des futurs médecins. Ces deux projets sont réalisés en partenariat avec Genopole et le CHSF. La plate-forme d'environnement virtuel et de réalité augmentée (Evr@) offre l'expertise nécessaire à la conception de techniques de visualisation et d'exploration de données massives en biologie, d'interfaces immersives centrées sur l'utilisateur pour l'étude des interactions moléculaires dans un environnement de réalité virtuelle et de réalité augmentée. Les principaux domaines d’activités de la plate-forme Evr@ sont (1) l’assistance numérique multimodale à la personne, (2) la capture et l’analyse de gestes de la personne, (3) la télé-opération précise de robots avec des gestes naturels et (4) l’aide à l’expérimentation d’hypothèses structurales en biologie. Contact : Amine Chellali (amine.chellali@univ-evry.fr); Frédéric Davesne (frederic.davesne@univ-evry.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
April 5, 2020 4:40 PM
|
Le plateau de chimie environnemental de recherche, rattaché à l’UMR INRAE / AgroParisTech ECOSYS, se situe à Thiverval-Grignon dans les Yvelines et est constitué de 5 sous-plateaux - voir descriptif ci-dessous - et met à disposition des outils et des compétences pour accompagner les recherches en environnement. On y trouve des dispositifs expérimentaux originaux mais aussi des équipements moyens et lourds, maîtrisés par des équipes de techniciens et ingénieurs motivés. Récemment, plusieurs matériels ont été acquis ou renouvelés pour permettre des analyses de plus en plus performantes. C’est notamment le cas d’un compteur à scintillation (Tri-Carb modèle 4910 TR, Perkin Elmer) sur le plateau « radioisotopes », équipement qui a pu être acheté en partie avec une subvention de l’Université Paris-Saclay – Département SdV en réponse à l’AAP "Petit Equipement de Laboratoire" 2019. Ce compteur devrait permettre de poursuivre la réalisation de bilans complets de la répartition de molécules polluantes marquées au carbone-14 dans des sols, eaux ou plantes, notamment dans le cadre des projets Rhizopharma (Pari Scientifique, INRAE) et Piegeachlore (R&D GESIPOL, Ademe). Plus d’information sur ce sujet : Goulas et al. Science of The Total Environment 2019. Le plateau « isotopes stables » a lui-aussi fait peau neuve avec l’acquisition d’un spectromètre de masse isotopique (IRMS, modèle Isoprime Precision, Elementar), couplé à un analyseur élémentaire CHN (modèle Vario isotope Select) et un analyseur Headspace (modèle Isoflow). Il va notamment permettre de quantifier la cinétique d'évolution du carbone de sols cultivés dans des essais de longue durée, nationaux ou européens, en utilisant l'abondance naturelle en carbone-13, cette fois dans le cadre du projet ANR StoreSoilC. Plus d’information sur ce sujet : Cardinael et al. European Journal of Soil Science 2019. L’appareil permet également d’analyser l’azote 15. N’hésitez pas à solliciter les responsables des plateaux pour monter des collaborations dans le cadre de projets de recherche. ECOSYS / Plateau de chimie environnementale de recherche : Les objectifs des plateaux de chimie environnementale de recherche sont de mettre à disposition des compétences et des outils d'analyse et d'expérimentation, pour accompagner des recherches dans le domaine de l'environnement, avec le partage d'une problématique commune sur la caractérisation du fonctionnement de différents compartiments de l'environnement et sur l'évaluation des impacts liés à la dynamique des contaminants dans l'environnement. Ces plateaux, réunis dans un pôle de chimie environnemental, fonctionnent avec du personnel des unités constitutives (de techniciens aux ingénieurs de recherche), avec un temps partiel dédié, et un partage des compétences et des équipements analytiques. Ils constituent un support technique pour la construction de projets collaboratifs et pour structurer le partenariat externe. Les différents plateaux sont : i) radioisotopes (expérimentations avec des molécules marquées au 14C), ii) isotopes stables (expérimentations avec utilisation 13C et 15N), iii) chromatographie liquide - spectrométrie de masse (analyses de composés organiques dans des matrices complexes), iv) analyse des gaz à effets de serre (quantification de CO2, NH4, N2O et SF6), et v) mesure de composés organiques volatils (mesures en continu au champ ou en laboratoire). Contact : Marjolaine.Deschamps@inrae.fr Plug In Labs Université Paris-Saclay : cliquer ici
|

|
Scooped by
Life Sciences UPSaclay
October 11, 2020 5:23 PM
|
La bioproduction de composés d’intérêt par fermentation est une des applications majeures de la biologie de synthèse. Genopole fait figure de pionnier en France et dans cette discipline, représente le plus important groupement de scientifiques et industriels sur un même site géographique (Évry-Courcouronnes). La création de la nouvelle plateforme de bioproduction en fermenteurs entretient cette dynamique et offre les conditions propices au développement de nouveaux projets sur le biocluster Genopole et plus largement en Ile-de-France. Financée par Genopole et localisée au sein de la Pépinière Genopole Entreprises CCI Essonne, cette nouvelle plateforme est gérée par la société Abolis Biotechnologies, qui apporte le savoir-faire et l’expertise des responsables de projets fermentation aux scientifiques qui souhaiteront accéder aux équipements. Une plateforme de bioproduction ? Pour quels projets ? Deux exemples choisis : le dernier projet en date, toujours en cours, est à destination d’un grand groupe industriel international fournisseur d’ingrédients dans le domaine alimentaire. Tous les développements à l’échelle laboratoire se feront sur la plateforme, l’industrialisation du procédé quant à lui (à partir du scale-up), se fera chez le client. Un précédent projet, achevé à ce jour, a consisté à évaluer pour un laboratoire pharmaceutique le potentiel de la fermentation comme nouvel axe stratégique de la production de son principe actif majeur. Dans ce cadre, la plateforme a été utilisée pour valider la faisabilité de cette approche et permettre un transfert de technologie. Que vous soyez une start-up, une PME, un laboratoire public ou privé ou bien même un grand groupe industriel, vous aussi, venez mettre en œuvre et réussir vos projets de fermentation. Quelles que soient vos compétences et expériences en la matière, vous bénéficierez d’un accompagnement adapté par des personnes expérimentées en fermentation. Ainsi vous rejoindrez la communauté de clients et/ou d’utilisateurs satisfaits qui ont mis en œuvre, tout ou partie de leur projet de fermentation. FLASH NEWS : La plateforme de bioproduction en fermenteurs s’est tout dernièrement enrichie de 4 mini-fermenteurs de dernière génération, de volume utile jusqu’à un litre et pilotables en parallèle ! Ils complètent les équipements de la plateforme, déjà forte de deux fermenteurs : un de 2 L ou 5 L et un autre de 20 L (pilote industriel). La plateforme de bioproduction en fermenteurs offre des équipements et l'expertise associée pour la bioproduction en fermenteurs de composés d'intérêt (médicaments, biocarburants, biomatériaux, etc.) par des levures ou des bactéries en conditions de culture aérobie ou anaérobie. La gamme de fermenteurs présents sur la plate-forme permet une montée en échelle progressive : les quatre mini-fermenteurs de 350 ml à 1 litre permettent de tester plusieurs conditions de culture en parallèle dans le but d’obtenir un rendement optimal du composé d’intérêt, avant l’échelle intermédiaire de 2 ou 5 litres permettant de s’assurer que les conditions de production sont stables, et enfin une montée en échelle jusqu’au pilote industriel de 20 litres. A propos de Genopole. Biocluster français dédié à la recherche en génétique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 83 entreprises de biotechnologies, 17 laboratoires de recherche, 26 plateformes technologiques, ainsi que des formations universitaires (université d’Évry Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Contact : Frédéric Austruy (frederic.austruy@abolis.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
September 27, 2020 1:01 PM
|
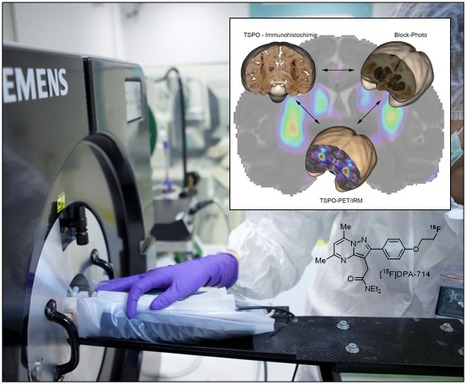
MIRCen (Molecular Imaging Center, Institut de Biologie François Jacob, CEA / Fontenay-aux-Roses) rassemble sur près de 8500 m2 des expertises et méthodologies complémentaires dédiées à l’exploration et la validation de concepts, ainsi qu’à l’évaluation préclinique de nouvelles stratégies thérapeutiques géniques, cellulaires ou médicamenteuses, principalement dans le domaine des maladies neurodégénératives. L’une de ses plateformes emblématiques est la plateforme d’imagerie TEP, IRM et optique (MIRCen / Imagerie TEP, IRM et optique), qui pour sa section relative à la Tomographie par Emission de Positons (TEP), est équipée de deux tomographes précliniques et dédiés au petit animal (FOCUS 220) et d’un laboratoire permettant des analyses métaboliques. Le versant clinique de ces recherches est quant à lui pris en charge par une équipe opérationnelle au Service Hospitalier Frédéric Joliot (SHFJ, Institut des sciences du vivant Frédéric Joliot, CEA / Saclay). Exemple choisi de recherche translationnelle impliquant la plateforme. La neuroinflammation joue un rôle clef dans l’ensemble des maladies neurologiques et neurodégénératives, comme la maladie de Parkinson et d’Alzheimer. Dans ce cadre, l’intérêt de l’imagerie de la neuroinflammation se trouve dans l’identification précoce et la quantification non-invasive de la cascade neuroinflammatoire, ceci afin de permettre le meilleur diagnostic possible pour le patient et son suivi dans le cadre de stratégies thérapeutiques. L’imagerie TEP repose sur l’identification de biomarqueurs moléculaires et, en ce qui concerne les phénomènes neuroinflammatoires, la cible moléculaire en vogue depuis plusieurs années est la protéine translocatrice 18kDa (TSPO 18kDa). Historiquement, le SHFJ a développé, marqué et évalué - en collaboration avec MIRCen - de nombreux radioligands spécifiques de cette cible dans des modèles animaux (rongeurs), au travers notamment de collaborations académiques internationales (FP7 INMiND) et industrielles, recherches qui ont notamment conduit au [18F]DPA-714, une pyrazolopyrimidine marquée par le fluor-18 (t1/2 : 109,8 minutes). L’utilisation de primates non-humains (PHN) – espèce intermédiaire et souvent obligatoire avant une application chez l’homme de radiotraceurs innovants – est un point fort de MIRCen, et dans ce cadre, a été développé un modèle animal dans lequel la surexpression cérébrale du TSPO est maîtrisée et localisée grâce à une stratégie de vectorisation virale (Van Camp et al, JCBFM 2020). Fort de ce modèle animal, la plateforme MIRCen / Imagerie TEP, IRM et optique a évalué la spécificité et sélectivité du [18F]DPA-714 au niveau cérébral, et les images tomographiques obtenues in vivo ont ensuite été corrélées avec des reconstructions tridimensionnelles du TSPO issues de coupes histologiques (in vitro) de cerveau. Cette étude a aussi permis de compléter (Lavisse et al, J. Neurosci. 2012 ; Peyronneau et al, Drug Metab Dispos 2013 ; Lavisse et al, EJNMMI 2014 ; Saba et al, Eur J Neurosci 2015) et de renforcer le positionnement unique de ce radiotraceur en tant qu’agent d’imagerie de la neuroinflammation, aujourd’hui engagé dans une étude clinique dans le cadre de la maladie de Parkinson (INFLAPARK), et visant à identifier chez le patient à quel stade de la maladie le phénomène inflammatoire apparait et dans quelles régions cérébrales en particulier. MIRCen / Imagerie TEP, IRM et optique. La plateforme propose des équipements et une expertise en imagerie du système nerveux central par Tomographie par Emission de Positons (TEP), Résonance Magnétique Nucléaire (RMN), et Imagerie Optique (IO). TEP : L'expertise de la plateforme se décline selon les trois axes stratégiques : i) validation de radiotraceurs sur des modèles animaux de maladies humaines, ii) validation de modèles animaux à l'aide de l'imagerie TEP et de radiotraceurs, iii) évaluation de nouvelles thérapies en vue du transfert vers la recherche clinique. La plateforme TEP interagit étroitement avec les plateformes RMN et Histologie pour la reconstruction 3D d'images multimodales (MIRCEN / Traitement d'Images). RMN : Des techniques d’imagerie haute résolution et de spectroscopie multinucléaires (13C et 31P) sont développées et validées: i) séquences RMN optimisées haute résolution du rongeur et du primate (pondérations T1, T2 et T2*), ii) séquences RMN optimisées pour la spectroscopie cérébrale 1H, 13C et 31P du rongeur et du primate, quantification automatisée de spectres à temps d’écho court et iii) imagerie CEST (« chemical exchange saturation transfer ») du glutamate chez le rongeur. L’optimisation de séquences complémentaires peut être réalisée dans le cadre des projets collaboratifs. IO : Les activités habituellement prise en charge sont : i) biodistribution in vivo de macromolécules (aptamères, anticorps, peptides, nanoparticules …), ii) suivi in vivo de la migration, de la différentiation et de l'amplification de cellules, iii) imagerie in vivo de l'expression de gènes. Contact : Nadia Van Camp (nadja.van-camp@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ici A propos de MIRCen. MIRCen - Molecular Imaging Research Center - est une installation de recherche préclinique développée par le CEA et l'INSERM. Cette installation, basée sur le site CEA de Fontenay-aux-Roses, est constituée d'un ensemble de plateformes dédiées au développement et à la validation de modèles animaux pertinents de pathologies humaines. Ces modèles sont utilisés pour le développement et la validation de nouvelles techniques permettant de détecter des déficits à un stade précoce de la maladie. Ils sont également utilisés pour évaluer des thérapies innovantes dans le domaine des maladies neurodégénératives. Ce centre fait partie intégrante de l’Infrastructure Nationale en Biologie et Santé (INBS) Neuratris, infrastructure dédiée aux thérapies innovantes en neurosciences.

|
Scooped by
Life Sciences UPSaclay
September 13, 2020 11:05 AM
|
Les interactions entre protéines sont des éléments essentiels des systèmes biologiques et leur analyse peut fournir des indications précieuses sur les fonctions des protéines. La plate-forme interactomique de l’IPS2 (SPOmics-Interactome) offre ses services aux équipes de recherche françaises et internationales. Exemple choisi : Récemment, une collaboration avec l’équipe de Gregory Vert (Signalisation Cellulaire et Ubiquitination, LRSV, Toulouse) nous a permis de développer plusieurs approches à grande échelle pour caractériser la machine d'ubiquitination E2-E3 entraînant la formation de la chaîne d'ubiquitine liée à K63 chez Arabidopsis (Arabidopsis thaliana). L'ubiquitination implique l'action séquentielle de trois classes d'enzymes : les Ub-activating enzymes, les Ub-conjugating enzymes (E2s) et les Ub ligases (E3s). Les activités de ces enzymes aboutissent à la fixation covalente de l'ubiquitine-Ub à un résidu de Lys (K) dans la protéine cible. Les chaînes PolyUb présentent des topologies différentes auxquelles sont associées des fonctions biologiques diverses. La liaison K48 des fragments d'Ub déclenche, par exemple, la dégradation des protéines cibles par le protéasome 26S. On en sait beaucoup moins sur les autres liaisons des chaînes polyUb. Les chaînes Ub liées au K63 n'induisent pas de dégradation dépendante du protéasome. Les rôles de la poly-ubiquitination impliquant le résidu K63 de l'Ub ont été largement étudiés chez la levure et les mammifères, y compris les rôles dans l'endocytose des protéines plasmamembranaires, les réponses aux dommages de l'ADN et, plus marginalement, l’autophagie et la signalisation. Les approches d’interactomique développées sur la plateforme SPOmics-Interactome ont permis d'identifier de nombreuses protéines qui interagissent avec les E2 UBC35/36 dédiées à la polyubiquitination K63 et leurs variantes E2 apparentées, dont plus d'une douzaine de ligases E3 et leurs cibles présumées. Les cibles de polyubiquitination K63 ont pu fournir une image plus complète des réseaux de polyubiquitine K63. Notre travail commun a permis d’identifier les enzymes conjuguant l'ubiquitine (E2) UBC35/36 comme les principaux moteurs de la formation de la chaîne de polyubiquitine K63 et mis en évidence le rôle majeur de ces protéines dans la croissance et le développement des plantes. En savoir plus ? Plug In Labs Université Paris-Saclay : cliquer ICI Contact : Dario Monachello (dario.monachello@inrae.fr) SPOmics-Interactome est la plateforme d’étude des interactions protéine-protéine de l’IPS2. Les technologies qu’elle maitrise sont basée sur la seule méthode permettant la détection de ces interactions à haut débit et in vivo - le système double-hybride chez la levure (Y2H) - aujourd’hui optimisé et automatisé. A ce titre, grâce à l’utilisation de plaques 384 puits et d’un système robotisé d’une part, mais aussi d’un protocole entièrement en milieu liquide, la plateforme est capable de cribler un pool de 50 protéines hybrides (DB- X) contre la banque de protéines hybrides AD-AIM (environ 12000 protéines d’Arabidopsis) en deux mois. Le protocole généralement utilisé est le suivant : Les plasmides portant les ORFs codant pour les protéines d’intérêt fournis par nos collaborateurs dans le vecteur pDEST - DB sont utilisés pour transformer des levures, puis les protéines DB-X hybrides sont testées pour éliminer celles capables d’auto-activation. Les levures exprimant les protéines DB-X hybrides sont alors cultivées individuellement dans des milieux sélectifs, puis combinées en pools de 50 clones et criblées contre la banque AD-AIM. Après identification des protéines candidates, une analyse matricielle en Y2H est effectuée par « déconvolution » des 50 DB-proies et par tests individuels contre les candidats AD-appâts. Une étape finale de séquençage des protéines AD- et DB- permet la validation de l’identité des protéines en interaction. Chaque nouvel ORF criblé est intégré dans la banque AD- permettant une constante implémentation du réseau.

|
Scooped by
Life Sciences UPSaclay
August 30, 2020 4:57 AM
|
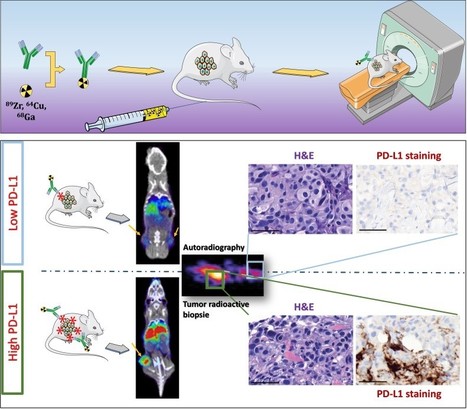
L’immunothérapie est devenue un grand espoir dans la lutte contre le cancer. Un point clé dans cette stratégie anti-tumorale est l’inhibition des points de contrôles immunitaires (tels que CTLA4, PD-1, PD-L1) au moyen d’anticorps afin d’entraver l’immunosuppression provoquée par la tumeur. Récemment, les divers succès des approches thérapeutiques utilisant par exemple l’ipilimumab (anti-CTLA4), le pembrolizumab et le nivolumab (anti-PD-1) ont vivement stimulé cet axe de recherche en oncologie. Dans le cas des thérapies anti-PD-L1, il est bien établi que le niveau d'expression pré-traitement de PD-L1 est prédictif de l’efficacité thérapeutique anti-PD-L1/PD-1. Au sein du Service Hospitalier Frédéric Joliot (SHFJ, Institut Joliot, CEA/Saclay, Orsay) et plus particulièrement de l’UMR BioMaps (CEA / CNRS / INSERM / UPSaclay) des chercheurs s’intéressent particulièrement à caractériser la distribution in vivo de cette protéine pour proposer un score prédictif thérapeutique. L’imagerie non invasive par tomographie par émission de positons (TEP) permet de suivre et de quantifier au cours du temps la distribution de l’expression de biomarqueurs tels que PD-L1, non seulement au niveau de la charge tumorale complète mais également au niveau du corps entier. L’utilisation de la TEP pour optimiser la translation et l'application des immunothérapies contre le cancer est aujourd’hui un concept très novateur allant vers une médecine de plus en plus personnalisée. À cet égard, il existe un besoin urgent de développer des stratégies qui mesurent la biologie de la charge tumorale complète d'un patient (tumeur et infiltrat) tout au long du développement tumoral notamment lors des traitements. L’objectif est ainsi de compléter, voire d’aiguiller les données obtenues en immunohistologie sur des biopsies. Notre expertise en immunoTEP - association d’un anticorps avec un radioisotope pour l’imagerie tomographique - permet de répondre à cet objectif. L’immunoTEP permet d’une part de caractériser la pharmacocinétique et la biodistribution d’un anticorps thérapeutique et d’autre part permet de développer des outils entièrement dédiés à l’imagerie pour le suivi de l’évolution de biomarqueurs d’intérêt [en savoir plus ?]. Dans ce cadre, une preuve de concept sur l’utilisation d’un anticorps (IgG) pour imager PD-L1 a été réalisée par la plateforme d’imagerie in vivo microTEP (SHFJ / imagerie préclinique, in vivo, microTEP) au sein de tumeurs présentant différents profils d’expression. Il a notamment été montré sur des modèles précliniques (souris) que l’IgG développée spécifiquement pour l’imagerie de PD-L1 détectait les changements aigus d’expression de PD-L1 induits par la chimiothérapie. Cet anticorps radiomarqué avec du Zirconium 89 (demi-vie de 3,3 jours) reconnait spécifiquement l’épitope extracellulaire PD-L1 murin et humain [Truillet et al, Bioconj. Chem. 2018]. Plusieurs stratégies sont aujourd’hui en cours d’évaluation sur la plateforme afin d’optimiser l’accumulation des ligands radiomarqués dans la tumeur, tout en favorisant leur élimination rapide dans les organes non ciblés. L’objectif est ainsi de proposer des ligands et une méthodologie associée pour un transfert vers la clinique. A ce titre, les chercheurs de BioMaps s’associent avec les plus grands experts locaux dans ce domaine, en interne CEA / JOLIOT (SiMOS ou Service d’ingénierie moléculaire pour la santé) pour l’ingénierie des ligands mais aussi avec l’écosystème clinique de Paris Saclay (Gustave Roussy). A suivre donc ! Le SHFJ compte plusieurs plateformes dédiées à l’imagerie clinique ou préclinique in vivo par TEP, dont SHFJ / imagerie préclinique, in vivo, microTEP, objet du présent FOCUS PLATEFORME. La plateforme d’imagerie TEP préclinique du SHFJ offre les équipements nécessaires pour préparer les animaux et réaliser de l’imagerie corps entier par tomographie par émission de positons (TEP) ou TEP couplée à la tomodensitométrie (CT) chez le rongeur, au moyen de radiotraceurs extérieurs ou produits sur site. Les services associées (hébergement des animaux, autoradiographie, immunohistochimie) peuvent compléter les prestations. Vous souhaitez consulter d’autres FOCUS PLATEFORME relatifs au Service Hospitalier Frédéric Joliot ? N’hésitez pas à cliquer sur SHFJ / Radiochimie et SHFJ / Imagerie préclinique et clinique, in vivo, TEP/IRM ! Contact : Alexandra Winkeler (alexandra.winkeler@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
August 16, 2020 4:51 PM
|
COVID-19 oblige, notre évènement initialement prévu le 28 avril 2020 a été reprogrammé au mardi 17 novembre 2020. Rien d’autre ne change, Genopole et l’Université Paris-Saclay organisent toujours cette deuxième édition de MeetMyPlatform | Sciences de la Vie, en partenariat avec le Crédit Agricole Consumer Finance qui nous accueillera dans ses locaux à Massy. MeetMyPlatform | Sciences de la Vie #2, sera à nouveau une série de temps forts scientifiques - une plénière, une table ronde industrielle, trois ateliers - des opportunités uniques de découverte et de partage avec plus de 110 plateformes académiques et près de 20 partenaires industriels présents, sans oublier des moments de convivialité et d’échange ... Les inscriptions déjà faites restent valables. Et vous ? y serez-vous aussi ? Cliquer sur le lien ci-dessous pour découvrir le programme complet de cette journée, mais aussi vous inscrire (inscription gratuite mais obligatoire) ! PROGRAMME | INSCRIPTION EN LIGNE MeetMyPlatform | Sciences de la Vie est une journée de rencontres entre plateformes et entreprises, organisée par Genopole et le Département Sciences de la Vie de l’Université Paris-Saclay. Son objectif est double : rassembler et donner de la visibilité à la communauté des plateformes de l’Université Paris-Saclay et permettre aux acteurs de la recherche mais aussi aux entreprises présentes lors de cet évènement, de les découvrir ou de renforcer des liens déjà existants. Pour mémoire, la première édition de MeetMyPlatform | Sciences de la Vie s’était tenue le 31 janvier 2019 - un jour de neige ! – et avait su rassembler sur le plateau de Saclay près de 300 participants : ingénieurs et techniciens de plateformes, acteurs de la recherche académique mais aussi représentants de grands groupes, de PME/ETI et de Start-up/TPE. Ces derniers ont particulièrement apprécié la convivialité de l’événement et la facilité d’obtention d’informations pertinentes dans leurs domaines d’activités. Les membres des plates-formes ont, quant à eux, apprécié ce mode de communication et de présentation de leurs équipements, expertises et savoir-faire, ainsi que les moments d’échanges privilégiés (ateliers) qui leur étaient consacrés en deuxième partie de journée pour notamment renforcer ou structurer de nouveaux réseaux thématiques au niveau sud-francilien. Rendez-vous le mardi 28 avril 2020 pour une deuxième édition ! Vous souhaitez en savoir plus sur MeetMyPlatfom, cliquer ici pour une présentation en 70 secondes chrono ! MeetMyPlatform | Sciences de la Vie s’inscrit naturellement dans la dynamique continue de mise en visibilité des compétences, expertises et équipements des plates-formes de l’Université Paris-Saclay et ceci notamment via son portail PlugInLabs Université Paris-Saclay. Comité d’organisation : Frédéric Dollé (frederic.dolle@cea.fr), Marion Le Devedec (marion.ledevedec@universite-paris-saclay.fr), Jessica Pericaud (jessica.pericaud@universite-paris-saclay.fr) et Julien Picot (julien.picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
July 12, 2020 1:18 PM
|
Le contexte. Le soleil, source d’énergie renouvelable la plus abondante, possède le potentiel de remplacer à plus ou moins long terme les énergies fossiles. Son caractère intermittent (alternance jour/nuit) nécessite par contre la capture de son énergie et sa conversion sous une forme chimique, facilement stockable et transportable. A Paris-Saclay, plusieurs communautés se sont regroupées pour travailler activement dans ce domaine des énergies alternatives, et notamment au développement de systèmes de conversion efficaces, robustes et économiquement viables. Ces recherches, multidisciplinaires par nature, s’intègrent aujourd’hui dans des programmes propres à certains organismes, comme la fermeture du cycle de carbone au CEA, mais également dans des programmes mobilisant des scientifiques de tout le territoire Paris-Saclay, citons ceux impliqués dans l’initiative de recherche stratégique (IRS) et objet transverse et interdisciplinaire (OTI) Momentom (MOlecules and Materials for the ENergy of TOMorrow). Un des procédés développés dans le cadre de ces programmes est la photocatalyse, considérée aujourd’hui comme une solution innovante et alternative pour la production d’un « carburant solaire » à partir de ressources abondantes et inépuisables que sont l’eau et le dioxyde de carbone. Aussi appelée « photosynthèse artificielle », cette approche s’inspire des mécanismes de la photosynthèse naturelle, source principale d’oxygène nécessaire à la vie sur terre et à l’origine de nos réserves énergétiques fossiles sous forme de biomasse. Des recherches innovantes, mais aussi des contributions clés de plateformes … Exemple choisi. Récemment, des équipes de recherche de l’Institut de biologie intégrative de la cellule / Institut des sciences du vivant Frédéric Joliot (I2BC, CNRS/CEA/Université Paris Saclay), de l’Institut de chimie physique (ICP, CNRS/Université Paris Saclay) et de l’Institut de chimie moléculaire et des matériaux d’Orsay (ICMMO, CNRS/Université Paris Saclay) viennent de faire un pas décisif dans le domaine de la photosynthèse artificielle : ensemble, elles ont utilisé de nouveaux polymères organiques nanostructurés, molécules complexes qui, en solution et sous l’action de la lumière, ont pu reproduire les fonctions du Photosystème II : oxyder l’eau en oxygène et stocker l’hydrogène produit au sein d’une molécule tierce (ici une quinone réduite en quinol). Ce travail n’aurait pu se faire sans la contribution majeure de la plateforme de spectroscopie IRTF de l’I2BC, ses équipements et les données générées ayant permis de mettre en évidence que les électrons et protons issus de la réaction d’oxydation de l’eau sont dans un premier temps stockés sur le polymère nanostructuré ! En savoir plus ? Voir aussi Patel et al., Chem. Sci. 2020. La contribution de la plateforme ne s’arrêtera pas là, son expertise est déjà engagée dans la compréhension fine du mode de fonctionnement de ce nanomatériau photocatalytique très prometteur ! Vous avez dit « IRTF », « IRTF différentielle » ? La spectroscopie vibrationnelle infrarouge à transformée de Fourier (IRTF), comparée à la spectroscopie électronique (UV-visible) par exemple, est applicable à toute molécule. Elle délivre une quantité d’information extrêmement riche et permet d’obtenir des empreintes caractéristiques de fonctions chimiques. La technique dite différentielle permet quant à elle de s’affranchir des contributions dominantes et non souhaitées de certains composants du mélange observé (par exemple, le solvant) et de détecter sélectivement des modifications spectrales liées aux perturbations induites par l’opérateur au système (changement pH, changement redox, ou irradiation par de la lumière, voir illustration ci-dessus). La plateforme de spectroscopie IRTF de l’I2BC est située au Laboratoire des Mécanismes Fondamentaux en Bioénergétique (Institut Joliot, CEA Paris-Saclay). Elle met à disposition des utilisateurs des spectromètres IRTF avancés et elle est équipée pour répondre à la plus grande partie des besoins des analyses IRTF. La plateforme comprend 4 spectromètres avec plusieurs accessoires : cellule à transmission, accessoires ATR, cellule électrochimique, thermostats, cryostats pour expériences à basse température... Elle permet l’étude d’échantillons sous différentes formes (liquide, solide, poudre...). Le laboratoire est aussi spécialisé dans la spectroscopie IRTF différentielle, résolue dans le temps, à basse température, et possède une bonne expertise dans l’étude de réactions biochimiques et photo-induites. La plateforme de spectroscopie IRTF fait partie du pôle des plateformes de Biophysique de l’I2BC qui comprend les plateformes de RPE, FTIR, résonance Raman, spectroscopies électroniques et microscopie de fluorescence à super-résolution. Contacts : Annamaria Quaranta (Annamaria.QUARANTA@cea.fr), Winfried Leibl (Winfried.LEIBL@cea.fr), Ally Aukauloo (ally.aukauloo@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
June 28, 2020 10:27 AM
|
A Gif-sur-Yvette en 1953, le CNRS se dote d’un « moyen de culture expérimentale des plantes à tous les ordres de grandeur et de précisions dans le contrôle des facteurs physiques et chimiques de l’environnement » ; le premier phytotron de France est né ! Avec cet outil de premier rang mondial, des découvertes majeures seront faites sur la vernalisation, le photopériodisme et l’organogénèse végétale. 60 ans plus tard au sein de l’Institut des Sciences Végétales (ISV), dans des proportions plus modestes, le CNRS poursuit son investissement dans la biologie végétale à Gif-sur-Yvette et permet l’installation de phytotrons modernes permettant le contrôle des paramètres du développement végétal que sont la température, l’hygrométrie, la durée, la qualité et l’intensité de la lumière avec les dernières technologies disponibles sur le marché. L’heure est à la génétique et à la compréhension des processus physiologiques des plantes, ainsi que les dialogues moléculaires que peuvent établir certaines plantes avec un pathogène (virus) ou un symbiote (bactérie). La plateforme d’expérimentation végétale de l’I2BC est composée de 3 espaces indépendants en confinement S2/S3 rénovés en 2013. Ainsi, nous pouvons accueillir des plantes génétiquement transformées et des pathogènes végétaux de classe 2 ou 3. Ces espaces représentent une surface au sol de 1200 m2 et sont gérés selon les règles de la prophylaxie et de la lutte biologique afin d’éviter toute utilisation de pesticides de synthèse. Ces espaces disposent en outre de la supervision climatique permettant de garantir un climat stable et reproductible aux chercheurs avec un système d’alarme à distance pour intervenir en astreinte le cas échéant. L’équipe associée à cette plateforme se compose d’un responsable et d’un agent technique principal pour l’entretien des cultures expérimentales. En partenariat avec le service technique de l’I2BC et l’entreprise de maintenance prestataire de service, ils coordonnent leurs efforts avec l’ensemble des utilisateurs scientifiques concernés (internes à l’I2BC, mais aussi externes : collaborations académiques et privées) pour assurer la qualité nécessaire à ce service de soutien à la recherche en biologie végétale fondamentale et appliquée au sein de l’Université Paris-Saclay. La plateforme est aussi rattachée aux initiatives de formation nationale « Ecole Serristes INRA » et « Réseau National Serristes CNRS » et à l’initiative de la création d’un nouveau réseau sur Paris-Saclay : RéSEPS - pour Réseau SErristes Paris-Saclay - avec pour objectif de maintenir et développer un haut niveau d’entraide et de développement des connaissances et compétences de ce corps de métier utile aux scientifiques dont l’objet d’étude est la plante. Avec les révolutions génétiques de ces dernières décennies et les nouveaux outils de biologie moléculaire de plus en plus performants, l’Institut de Biologie Intégrative de la Cellule (I2BC) hérite de ce patrimoine technique précieux (serre et chambres de culture) destiné à la poursuite de la compréhension des mécanismes moléculaires qui régissent les génomes, la physiologie végétale et les interactions plantes micro-organismes. Son accès se fait via sa plateforme d’expérimentation végétale. Si l’Université Paris-Saclay a considérablement modifié le paysage scientifique où se trouvait jadis le premier phytotron de France près des rives de la Mérantaise, la recherche en biologie végétale est toujours présente et la serre ou la chambre de culture sont toujours l’outil indispensable à cette recherche. Son existence est de plus en plus rare et maintenue par d’importants et constants investissements de quelques organismes majeurs de la recherche (CNRS, INRAE, CEA, CIRAD). C’est pourquoi la mutualisation de ces outils est primordiale pour assurer leur pérennité et permettre au plus grand nombre l’accès à ce matériel scientifique insolite mais essentiel. In fine, même si les infrastructures d’expérimentations et de productions végétales de l’I2BC ne se distinguent pas par une technologie de rupture, elles ont la capacité d’être modernes et accessibles à tous ceux qui en auraient besoin pour leur recherche et qui en sont privés actuellement. Alors n’hésitez pas à nous contacter pour que vos expériences végétales puissent se réaliser ! Contact : Guillaume Lucas (guillaume.lucas@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
June 14, 2020 5:11 PM
|
La plateforme MicroScope est développée depuis 2002 par le laboratoire LABGeM du Genoscope (UMR8030 Génomique Métabolique, CEA/CNRS/Université d’Evry Paris-Saclay) et offre à une communauté internationale de microbiologistes des outils bioinformatiques pour l’analyse et l'exploration de génomes procaryotes. Elle est régulièrement enrichie en données et en fonctionnalités nouvelles, et un article récent permet de découvrir les derniers développements de la plateforme (Vallenet D., Calteau A. et al, Nucl. Acids Res. 2020). Outre l’ajout de nouvelles fonctionnalités pour l’annotation de la fonction biologique des gènes et la caractérisation de régions génomiques d’intérêt (e.g. systèmes de sécrétion, clusters de biosynthèse de métabolites secondaires), d’importants efforts ont été réalisés pour faciliter la sélection de génomes, la navigation et l’exploration des données. Les auteurs décrivent aussi deux méthodes originales, nommées PPanGGOLiN et panRGP, qui utilisent une nouvelle représentation des données sous la forme d’un graphe de pangénome pour capturer l’intégralité du contenu génomique de milliers de souches d’une espèce donnée. Ces méthodes permettent de réaliser des comparaisons de génomes à large échelle et de prédire des îlots génomiques qui sont des régions génomiques variables impliquées dans la pathogénie ou dans l’adaptation à des environnements très diversifiés. MicroScope (Genopole / Genoscope / Plateforme Microscope - Annotation et analyses comparatives de génomes microbiens) est une plate-forme Web dédiée à l'annotation et à l'exploration des fonctions des gènes microbiens grâce à des analyses génomiques comparées, pangénomiques et métaboliques. La plateforme prend en charge l'intégration de génomes et de métagénomes nouvellement assemblés et fournit également des services d'analyse de données de séquençage à haut débit pour la transcriptomique et la recherche de variants. L'interface utilisateur de MicroScope permet un travail collaboratif au sein d'un contexte comparatif riche afin d'améliorer l'effort de curation par la communauté. MicroScope contient plus de 12 000 génomes microbiens qui sont analysés et expertisés par une large communauté d’utilisateurs (plus de 4800 comptes, dont 65% sont enregistrés à l’étranger). La plateforme MicroScope est membre de l’Institut Français de Bioinformatique et de France Génomique et est labellisée Genopole. Contact : David Vallenet (vallenet@genoscope.cns.fr) Plug In Labs Université Paris-Saclay : cliquer ici A propos de Genopole. Biocluster français dédié à la recherche en génétique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 83 entreprises de biotechnologies, 16 laboratoires de recherche, 25 plates-formes technologiques, ainsi que des formations universitaires (université d’Évry Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologie et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon.

|
Scooped by
Life Sciences UPSaclay
May 31, 2020 1:01 PM
|
Les protéines membranaires font l’objet d’une attention toute particulière ! et pour cause … Bien qu’elles ne constituent que 20 à 30% du protéome humain, elles représentent la majorité des cibles thérapeutiques actuelles. Or l’étude de ces protéines, à l’échelle moléculaire, passe par leur extraction à partir des membranes cellulaires, une étape qui s’avère souvent délétère pour leur structure et leur fonction. La plateforme I2BC/Expression de protéines solubles ou membranaires en levures développe des protocoles pour l’expression de protéines membranaires dans la levure S. cerevisiae, pour des volumes de cultures allant de quelques millilitres à plus de 20 litres, ainsi que pour leur purification. Elle est hébergée au sein du Laboratoire des Protéines et des Systèmes Membranaires (LPSM, I2BC/Institut Joliot, CEA/Saclay, Gif-sur-Yvette), et l’exemple choisi d’application ci-dessous - pour l’étude structurale des flippases - illustre ses capacités. La composition en lipides des deux feuillets des membranes biologiques est asymétrique, cette asymétrie permettant de contrôler de nombreux processus cellulaires (par exemple, le trafic membranaire, la signalisation cellulaire…). L’asymétrie lipidique est maintenue grâce à des protéines appelées flippases, qui catalysent le transport de lipides du feuillet externe vers le feuillet interne des membranes. Chez l’Homme, des mutations de flippases sont impliquées dans de nombreuses pathologies. Récemment, des équipes du LPSM, de l’Université d’Aarhus et du Max Planck Institute se sont associées pour déterminer la structure à haute résolution d’une flippase, le complexe Drs2/Cdc50. Dans ce contexte, la plateforme I2BC/Expression de protéines solubles ou membranaires en levures a mis tout son savoir-faire au service de la préparation de grandes quantités d’un complexe Drs2/Cdc50 pur, et sous forme stable et fonctionnelle, pour son étude structurale ultérieure par cryo-microcopie électronique. Les différentes structures obtenues révèlent le mécanisme d’auto-régulation auquel est soumis le complexe Drs2/Cdc50 ainsi que l’existence d’une cavité par laquelle il est proposé que le lipide substrat soit prise en charge lors de son cheminement d’un feuillet à l’autre des membranes cellulaires (Timcenko M et al, Nature 2019). Le plateau technique I2BC/Expression de protéines solubles ou membranaires en levures donne accès à différents équipements (fermenteurs, broyeurs …) permettant l’expression et la purification de cibles protéiques singulières, les protéines transmembranaires par l’utilisation de Saccharomyces cerevisiae ou la levure méthylotrophe Pichia pastoris pour la préparation de protéines secrétées et riche en ponts disulfures. Cette expertise est localisée sur deux sites, au Laboratoire des Protéines et des Systèmes Membranaires – I2BC/Institut Joliot, CEA Saclay et au laboratoire Fonction et Architecture des Assemblages Macromoléculaires – I2BC, Faculté des Sciences d’Orsay. Ce plateau fait partie du pôle des plateformes de Biologie Structurale de l’I2BC qui comprend : i) les plateformes de Cristallisation, RMN, CryoEM, Mesures d'Interactions Macromoléculaires, et ii) les plateaux techniques d'Expression de protéines solubles ou membranaires en levures, Expression des protéines en cellules d'insectes et Bioinformatique structurale. Contacts : Cédric Montigny (cedric.montigny@i2bc.paris-saclay.fr), Karine Blondeau (karine.blondeau@i2bc.paris-saclay.fr), Guillaume Lenoir (guillaume.lenoir@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI.

|
Scooped by
Life Sciences UPSaclay
May 17, 2020 5:37 PM
|
MetaboHUB est l'infrastructure nationale de métabolomique et fluxomique créée en 2013 dans le cadre du programme "Investissements d'Avenir" lancé par le Ministère de la Recherche et de l'Enseignement Supérieur et l'Agence Nationale pour la Recherche (ANR). Elle a pour objectif de fournir des outils technologiques de pointe et des services en métabolomique et fluxomique aux équipes de recherche académiques et à des partenaires industriels dans les domaines de la santé, de la nutrition, de l'agriculture, de l'environnement et des biotechnologies. MetaboHUB est réparti sur quatre clusters géographiques : Clermont-Ferrand, Bordeaux, Toulouse et Paris-Saclay, ce dernier aussi appelé Métabolome - IdF et objet du présent FOCUS PLATEFORME. La métabolomique au service de l’identification de biomarqueurs de diagnostic / pronostique dans le cadre de l’insuffisance hépatique chronique ? Retour sur une étude récente. Maladie irréversible du foie, la cirrhose se caractérise par une inflammation chronique, entrainant plus ou moins rapidement la destruction des cellules hépatiques et leur régénération anarchique sous forme de nodules, et donc la perte pure et simple des fonctions vitales de l’organe (synthèse, stockage et élimination). En France, environ 200 000 personnes sont atteintes de cirrhose, dont 30% ont atteint le stade sévère de la maladie, entrainant un nombre de décès par an relativement élevé (10 000-15 000). Le syndrome ACLF (Acute-on-Chronic Liver Failure) correspond à une décompensation aiguë (aggravation brutale) de la maladie. Il est caractérisé par une inflammation systémique intense et la défaillance d’un ou plusieurs organes ou systèmes (foie mais aussi reins, cerveau, système circulatoire et système respiratoire), touche 30 à 40% des patients hospitalisés pour une cirrhose décompensée et est associé à une mortalité élevée à court terme (30% à 28 jours). En collaboration avec la fondation EF-CLIF (European Foundation for the study of Chronic LIver Failure), la plateforme Saclaysienne de Métabolome - IdF (SPI / LEMM, voir ci-dessous) a analysé le métabolome de 831 personnes atteintes d’une cirrhose décompensée accompagnée ou non de complications aiguës (ACLF). Ce travail récemment publié (Moreau et al, J. Hepatol. 2019) montre qu’il existe une signature métabolomique constituée de 38 composés qui s’accumulent spécifiquement dans le sérum des patients atteints d’ACLF. La nature même de ces composés suggère que les mécanismes responsables de la défaillance d’organes dans l’ACLF seraient proches de ceux responsables du sepsis, avec notamment une inhibition forte de la production d’énergie dans les mitochondries. Les auteurs proposent également une séquence d’événements conduisant à l’ACLF : les modifications dans la réponse hormonale au stress et la génération d’espèces réactives de l’oxygène et du souffre initieraient le dysfonctionnement des mitochondries, conduisant à son tour à celui d’un ou plusieurs organes. Cette étude pionnière dans le domaine a permis au laboratoire et sa plateforme d’intégrer deux nouveaux consortiums européen dans le cadre du programme H2020 : Microb-Predict (2019-2024) et Decision (2020-2024), tous deux dédiés à une meilleure compréhension ou pronostic d’une cirrhose du foie avec défaillance d’organes. Métabolome - IdF est une plateforme bi-site (LEMM/SPI, Département Médicaments et Technologie pour la Santé, Institut des sciences du vivant Frédéric Joliot, CEA Paris-Saclay, Gif-sur-Yvette et Institut Parisien de Chimie Moléculaire, Sorbonne Université Campus Pierre et Marie, Paris), labélisée IBISA depuis 2010. Les prestations qu'elle offre s’articulent autour de l’analyse métabolomique par spectrométrie de masse. Plusieurs types de prestations peuvent être réalisés: i) production de données brutes - empreintes métabolomiques par spectrométrie de masse à basse, haute ou ultra-haute résolution de milieux biologiques - ou traitées sur la base des outils mathématiques et (bio)informatiques développés au laboratoire, ii) identification de métabolites (grâce notamment à une chimiothèque, > 1000 produits chimiques et interrogations automatiques de banques de données publiques) et iii) analyses statistiques et biologiques de données (positionnement de métabolites identifiés et leurs niveaux relatifs de concentrations au sein de voies métaboliques connues). Contact : François Fenaille (Francois.Fenaille@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
May 3, 2020 10:40 AM
|
La spectroscopie par résonnance magnétique nucléaire (RMN) peut fournir des indications précieuses sur les caractéristiques structurales et dynamiques des systèmes biomoléculaires ; elle est particulièrement bien adaptée à l’analyse à l’échelle atomique des états fonctionnels multiples, de la flexibilité interne et des processus d’interaction impliquant des molécules biologiques. La plateforme de spectroscopie RMN de l’I2BC (Département I2BC@Saclay, Institut Joliot, CEA/Saclay, Gif-sur-Yvette) est spécialisée dans la caractérisation des protéines, repliées et/ou désordonnées, impliquées dans la signalisation cellulaire : repliement, dynamique, interactions protéine-protéine et protéine-ligand. La plateforme s’appuie aussi sur une expertise dans la production de protéines recombinantes : protéines désordonnées, kinases, phosphatases produites en bactéries, cellules d’insectes ou en milieu « cell-free », et opère deux spectromètres à haut champ (600 et 700 MHz). Récemment, La plateforme a développé et exploité une nouvelle capacité : l’identification et le suivi cinétique de modifications post-traductionnelles (PTMs) à l’échelle du résidu. Cette approche permet de caractériser des modifications multiples voisines ou à motif dégénéré, et peut virtuellement être appliquée à tout type de PTM. Focus sur 2 études récentes, illustrant le potentiel de cette méthode ! 1 - La plateforme (en s’associant avec l’équipe de A. Carreira (Institut Curie)) a identifié les défauts de phosphorylation du suppresseur de tumeur BRCA2 causés par des mutations trouvées chez des patientes atteintes de cancer du sein. L’étude menée a permis i) d’identifier les résidus de BRCA2 phosphorylés par la kinase PLK1 en mitose, ii) d’observer que ces phosphorylations sont moins présentes dans les BRCA2 mutés, iii) de révéler que ces phosphorylations déclenchent la liaison de PLK1 à BRCA2 et permettent l'alignement des chromosomes au niveau de la plaque équatoriale pendant la mitose, iv) de montrer que ces évènements sont défectueux lorsque BRCA2 est muté. Ces résultats révèlent un nouveau mécanisme qui pourrait contribuer aux aberrations chromosomiques observées dans les tumeurs exprimant des variants de BRCA2 (en savoir plus, Ehlen et al, Nat. Commun. 2020). 2 - La plateforme a également identifié les sites phosphorylés sur Mdm2, l’ubiquitine-ligase régulant p53, à l’issue de dommages de l’ADN grâce à une nouvelle méthode utilisant la détection 13C directe. Celle-ci permet désormais d’étudier par RMN les protéines désordonnées dans des conditions physiologiques de pH et de température, un savoir-faire unique dans le monde (en savoir plus, Alik et al, Angew. Chem. Int. Ed. 2020). I2BC / Plateforme de spectroscopie RMN est spécialisée dans la caractérisation des protéines en solution, qu’elles soient repliées ou désordonnées. Elle s’est en particulier focalisée sur les protéines impliquées dans la signalisation cellulaire, la réparation des dommages de l’ADN et l’organisation du noyau : repliement, dynamique, interactions protéine-protéine et protéine-ligand, décrits à l’échelle atomique que permet d’atteindre cette spectroscopie. La plateforme a aussi développé et exploité une nouvelle approche : l’identification et le suivi cinétique résidu par résidu de modifications post-traductionnelles (PTMs). Cette approche permet d’identifier et de caractériser des modifications multiples voisines ou à motif dégénéré, et peut virtuellement être appliquée à tout type de PTM (plus d’informations : voir Ehlen et al, Nat. Commun. 2020 / Alik et al, Angew. Chem. Int. Ed. 2020). Enfin, la plateforme utilise la RMN pour cartographier des interfaces dans le cadre de la conception d’inhibiteurs d’interaction protéine-protéine (plus d’informations : voir Bakail et al. Cell. Chem. Biol. 2019). Coté équipements, la plateforme possède deux spectromètres à haut champ (600 et 700 MHz) équipés d’une cryo-sonde TCI 1H, 15N, 13C, et est rattachée à FRISBI, l’infrastructure nationale pour la biologie structurale intégrative. Enfin, cette plateforme fait partie du pôle des plateformes de Biologie Structurale de l’I2BC qui comprend : i) les plateformes de Cristallisation, RMN, CryoEM, Mesures d'Interactions Macromoléculaires, et ii) les plateaux techniques d'Expression de protéines solubles ou membranaires en levures, Expression des protéines en cellules d'insectes et Bioinformatique structurale. Contact : Sophie ZINN-JUSTIN (Sophie.ZINN@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
April 19, 2020 5:34 PM
|
Vous avez dit pharmacocinétique ? La pharmacocinétique (ou PK) étudie le devenir d'une substance active après son administration dans l'organisme. Elle comprend quatre grandes étapes : l’absorption (A), la distribution (D), le métabolisme (M), et l’excrétion du principe actif et de ses métabolites (E). La détermination des paramètres pharmacocinétiques d'un candidat médicament fournit les données qui permettront de choisir la voie d'administration, la forme galénique, la dose et la fréquence d’administration pour son utilisation future. Pour cela, il faut suivre au cours du temps la molécule d’intérêt et ses métabolites éventuels dans différentes matrices biologiques et pour ce faire la spectrométrie de masse est aujourd’hui la méthode de choix en matière de sensibilité, précision et spécificité. On obtient les premières informations lors de tests in vitro, liaison du composé aux protéines plasmatiques, aux hématies, stabilité métabolique du candidat dans le plasma de différentes espèces (rongeurs, humain) et également en présence des enzymes des hépatocytes (mesure de la clairance intrinsèque). La recherche et l’identification de métabolites produits par ces enzymes peut débuter à ce stade. L’étude du passage des barrières physiologiques comme par exemple la barrière hémato-encéphalique peut aussi être explorée précocement in vitro ou in vivo. Ensuite une première phase exploratoire du profil pharmacocinétique est obtenue in vivo chez une espèce animale modèle judicieusement choisie. Qu’est-ce que SMArt-MS ? Une plateforme spécialisée dans le développement et la validation de méthodes de bioanalyse quantitative par LC-MS/MS de petites molécules et d'anticorps thérapeutiques dans les fluides biologiques et leur utilisation pour la réalisation d'études de métabolisme et pharmacocinétique pilotes (early ADME-DMPK). Née le 1er février 2016, aujourd’hui certifiée ISO9001, cette plateforme est rattachée au Service de Pharmacologie et d’Immunoanalyse (SPI, Département Médicaments et Technologie pour la Santé, Institut Joliot, CEA/Saclay, Gif-sur-Yvette). Forte d’une expérience forgée au contact de l’industrie pharmaceutique internationale, SMArt-MS a été un contributeur actif dans plusieurs projets du LabEx LERMIT mais aussi auprès de nombreuses équipes académiques et biotechs du territoire Paris-Saclay. Son dynamisme, ces quatre dernières années, peut se résumer en deux chiffres : 50 projets réalisés et 22 publications signées (cliquer ci-contre pour consulter la dernière parue : Le Douce et al, Cell Metabolism 2020). SMArt-MS a aussi été récemment sollicitée pour faire partie de l’infrastructure de recherche ChemBioFrance. SMArt-MS et le projet NUTS : un exemple choisi de réussite et de développement ! Une autre de nos fiertés, est d’avoir été dès 2016 choisis par les porteurs du projet NUTS (LabEx LERMIT, lutte contre l’hypertension artérielle pulmonaire (HTAP)) en tant que partenaire pour l’entrée de ce projet en maturation à la SATT Paris Saclay (NUTS-MAT). Depuis, un travail considérable a été réalisé (chimie, PK mais aussi biologie), permettant de proposer une nouvelle cible thérapeutique, de sélectionner un candidat médicament avec un très bon profil de sécurité et d’obtenir des résultats in vivo avec une preuve de concept d’efficacité et de survie. La confiance de nos partenaires ainsi que celle de la SATT Paris Saclay a permis à SMArt-MS au nom de l’équipe projet de « pitcher » à la soirée Team-up for Startup #HealthTech (Institut Pasteur, 28 janvier 2020) dans un format 180 secondes et devant une centaine de participants. Plusieurs contacts ont été pris afin d’identifier un potentiel CEO et des investisseurs en vue de la création d’une Start-up en 2020. Des rendez-vous sont d’ores et déjà pris et l’aventure continue… La plateforme SMArt-MS possède des équipements de chromatographie ultra haute pression couplés à des spectromètres de masse quadripolaires permettant la mesure très sensible des composés d’intérêt et leurs métabolites. Ces appareils permettent la quantification de petites molécules mais aussi celle des « Biologics » comme les peptides, protéines et anticorps thérapeutiques. SMArt-MS fait partie intégrante de la plateforme Métabolome-IDF de l’infrastructure MetaboHUB, basée à Saclay et réalise la mesure quantitative absolue de métabolites endogènes en vue de leur validation comme biomarqueurs. SMArt-MS a ainsi accès aux spectromètres de masse de haute résolution de cette plateforme / infrastructure permettant les identifications délicates de certains métabolites. Contact : Alain Pruvost (alain.pruvost@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI.
|



 Your new post is loading...
Your new post is loading...