Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
October 8, 2023 5:15 PM
|
Des applications pour règlementer les échanges de ressources agronomiques. Soutenu par le GIS IBiSA, le projet ABS4BRCs a abouti à la création d'un site web et d'un logiciel qui facilitent les échanges de ressources agronomiques dans le respect de la règlementation internationale. Dédiées aux aspects règlementaires, ces applications s'adressent aux gestionnaires de collections, aux chercheurs et aux juristes des institutions. Lire l’article. Plus de 30 CRB français investis sur le développement de ces applications. Les applications du projet ABS4BRCs ont été mises au point avec la contribution d'une trentaine de CRB français du Cirad, d'INRAE et de l'IRD. Ces CRB sont tous intégrés à l'infrastructure nationale Ressources agronomiques pour la recherche (RARe) et pour la plupart référencés dans l'annuaire des CRB du GIS IBiSA. CRB du réseau RARe. Vous cherchez des ressources biologiques pour vos projets de recherche ? L'annuaire IBiSA recense les ressources et services proposés par les CRB labellisés IBiSA en agronomie, mais aussi en santé. Annuaire des CRB IBiSA. Votre CRB est labellisé IBiSA mais ne figure pas dans l’annuaire ? Contactez le GIS IBiSA par mail à l'adresse secretariat@ibisa.net pour connaître la procédure et obtenir le formulaire à remplir. Contacter le GIS IBiSA. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! Vous souhaitez découvrir le potentiel complet de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes du domaine Sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle du CEA, du CNRS, d'INRAE, de l'Inria, de l'Inserm, de l’INCa, de la CPU et du Ministère de l’enseignement supérieur, de la recherche et de l’innovation (MESRI), il est l’unique instrument de financement commun à l’ensemble des établissements en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité en vue de la structuration et certification des plateformes. Plus d’infos sur ibisa.net. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire ! Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaire académique comme entreprise, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
September 24, 2023 12:36 PM
|
L'Observatoire du Végétal (OV) est un ensemble unique de ressources dédiées au phénotypage multi-niveaux des plantes. Il est adossé à l’Institut Jean-Pierre Bourgin (IJPB, UMR 1318, INRAE-AgroParisTech-Université Paris-Saclay) et au réseau Saclay Plant Sciences (SPS). Ces infrastructures labellisées IBiSA sont situées sur le Centre INRAe de Versailles. L’objectif de l’OV est de mettre en place une analyse intégrée et à haut débit des plantes, alliant phénotypage macroscopique, biochimique, cytologique, chimique, métabolique. Il s’appuie à ce titre sur 6 composantes / plateformes : Biochimie (resp. Hakim MIREAU, Alexandre de St GERMAIN), Chimie / Métabolisme (resp. Grégory MOUILLE), Culture de plantes (resp. Hervé VAUCHERET), Cytologie / Imagerie (resp. Bertrand DUBREUCQ), Phenoscope (resp. Olivier LOUDET) et le CRB Arabidopsis (resp. Christine CAMILLERI, Christine HORLOW). Ce cluster, dédié au phénotypage des plantes, permet de réaliser une caractérisation à des niveaux d’analyse multiples d’échantillons végétaux produits in situ. Contacts : Christian Meyer (christian.meyer@inrae.fr) IJPB / OV : cliquer ICI Les plateformes de IJPB / Observatoire du Végétal prennent régulièrement RDV avec vous au travers de FOCUS PLATEFORME. Profitez de cette brève pour découvrir à nouveau un éclairage sur leurs expertises, leurs équipements et leurs offres de collaborations ou de prestations ! - IJPB / Observatoire du végétal - Plateforme de Biochimie
FOCUS PLATEFORME : Quand la biochimie se joint à la synthèse organique pour préserver la culture du chanvre industriel ! - IJPB / Observatoire du végétal - Plateforme Chimie-Métabolisme
FOCUS PLATEFORME : L'analyse des métabolites spécialisés des plantes : un outil pour l'étude des interactions plantes/microorganismes, une spécificité de la plateforme Chimie-Métabolisme-Métabolome de l'Observatoire du Végétal - IJPB / Observatoire du végétal - Plateforme de culture de plantes
FOCUS PLATEFORME : La culture sous tous les climats : une spécificité de la plateforme Observatoire du Végétal - Culture de Plantes à l'INRAE de Versailles ! - IJPB / Observatoire du végétal - Plateforme de Cytologie et Imagerie Végétale
FOCUS PLATEFORME : La microdissection assistée par laser, un apport « tranchant » dans l'analyse à très petite échelle des transcriptomes végétaux !
FOCUS PLATEFORME : AAP SESAME 2021 - Vers de nouveaux fronts de science avec un microscope à super résolution à technologie STED dès 2022 ! - IJPB / Observatoire du végétal – Phenoscope
FOCUS PLATEFORME : Changement d'échelle avec le développement d'un nouveau robot de phénotypage des plantes de type 'PHENOSCOPE'... en taille XL ! - IJPB / Observatoire du végétal - Centre de ressources biologiques Arabidopsis
FOCUS PLATEFORME : Le Centre de Ressources Biologiques (CRB) Arabidopsis de l’Observatoire du Végétal à l’INRAE de Versailles : des ressources génétiques uniques

|
Scooped by
Life Sciences UPSaclay
September 10, 2023 5:47 PM
|
FOCUS PLATEFORME : Plateforme MicroScope : 20 ans de contribution à l’analyse des génomes bactériens MicroScope est une plateforme bioinformatique de premier plan qui offre aux biologistes des outils puissants et des ressources pour approfondir leurs recherches en génomique microbienne, notamment pour comprendre le métabolisme des bactéries et en explorer le potentiel. Issue des recherches de l’équipe LABGeM (UMR 8030 - Génomique Métabolique, CEA/CNRS/Université d’Evry Paris-Saclay, Genoscope, Institut Jacob, CEA, Évry-Courcouronnes), elle fait partie des 24 plateformes et plateaux techniques mutualisés du biocluster Genopole, qui soutient son activité par des investissements réguliers notamment en serveurs informatiques. MicroScope est aussi membre de l’Institut Français de Bioinformatique et associée à l'infrastructure France Génomique. Le LABGeM (pour Laboratoire d’analyses bio-informatiques pour la génomique et le métabolisme) a été créé en 2001 par Claudine Médigue, au sein du centre national de séquençage Genoscope, grâce au dispositif ATIGE* de Genopole dont la chercheuse était lauréate. Portée par la dynamique des activités de séquençage du Genoscope et de nombreuses collaborations avec des microbiologistes désirant analyser le génome de leurs bactéries modèles, l’équipe développe à partir de 2002 une plateforme d’annotation collaborative de génomes microbiens, nommée initialement MaGe pour « Magnifying Genomes » puis MicroScope à partir de 2007. Depuis, la plateforme a régulièrement été enrichie de données et de fonctionnalités nouvelles. Les évolutions de la plateforme MicroScope ont fait l’objet de 5 publications (plus de 1600 citations depuis 2006) et ont été présentées dans de nombreuses conférences en France et à l’étranger. Les membres du laboratoire ont également été impliqués dans plus de 150 publications collaboratives dans divers domaines d’applications comme la génomique environnementale, l’épidémiologie et la santé avec l’étude de bactéries pathogènes de l’homme ou celles de nos microbiotes, ou encore les biotechnologies. Ainsi, la découverte de nouvelles fonctions et activités enzymatiques bactériennes peut donner lieu au développement de procédés, notamment de dépollution ou de production biologique de composés d’intérêt. Aujourd’hui, la plateforme MicroScope est dirigée par David Vallenet et Alexandra Calteau. « MicroScope est utilisée par une large communauté internationale de microbiologistes académiques : plus de 6700 comptes sont créés, dont 65% sont enregistrés à l’étranger » mentionne Alexandra Calteau. « Elle a contribué à l’analyse de plus de 25 000 génomes microbiens » précise-t-elle. Plusieurs entreprises privées y ont également recours dans le cadre de prestations de service ou de projets de recherche collaboratifs. Les 20 ans de MicroScope seront fêtés le 26 septembre 2023 dans la salle Elyseum de Genopole autour d’orateurs partageant leur expérience d’utilisateur et de la présentation par le LABGeM des nouvelles méthodes développées. Il sera retransmis en direct. Vous souhaitez participer à cet évènement ? inscription gratuite mais obligatoire - places limitées ! *ATIGE : « Action thématique incitative de Genopole » offrant à des chercheurs les moyens d’intégrer un laboratoire académique du biocluster et d’y créer une nouvelle équipe de recherche. La majorité des bénéficiaires d’Atige, devenus leaders scientifiques, sont restés à Genopole et ont enrichi le biocluster de nouvelles thématiques scientifiques. Contact : david.vallenet@cea.fr / Julien.Picot@genopole.fr Plug In Labs Université Paris-Saclay : cliquer ICI Envie de (re)lire leur précédent FOCUS PLATEFORME (15 juin 2020) ? FOCUS PLATEFORME : Annoter et explorer la fonction biologique des gènes de milliers de microorganismes dans un environnement bioinformatique intégré GENOSCOPE / Plate-forme MicroScope - Annotation et analyses comparatives de génomes microbiens – Genopole. MicroScope est une plateforme web dédiée à l'annotation et à l'exploration des fonctions des gènes microbiens grâce à des analyses comparatives génomiques, pangénomiques et métaboliques. La plateforme prend en charge l'intégration de génomes et de métagénomes nouvellement assemblés et fournit également des services d'analyse de données de séquençage à haut débit pour la transcriptomique et la recherche de variants. L'interface utilisateur de MicroScope permet un travail collaboratif au sein d'un contexte comparatif riche afin d'améliorer l'effort de curation par la communauté. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 65 entreprises de biotechnologies, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : créer et soutenir des entreprises de biotechnologies et le transfert de technologies vers le secteur industriel, favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
July 15, 2023 12:58 PM
|
La plateforme d'analyse du mouvement du CIAMS (Complexité, Innovation, Activités Motrices et Sportives) est située dans les locaux de l'hôpital Paul-Brousse à Villejuif, et a pour objectif de développer des travaux de recherche appliqués au champ de la santé, en utilisant des outils de mesures et des concepts relevant de la biomécanique et de la neurophysiologie. Les espaces rénovés de plus de 150 m² accueillent une vaste salle d'expérimentation, divisée en 5 espaces modulaires dédiés à différents types d'expérimentations: i) un espace posture équipé d'une plateforme de force de 60 cm x 60 cm et d'un dispositif d'inclinaison de la surface d'appui au sol ; ii) un espace mouvement équipe d'une plateforme de 60 cm x 120 cm et dédié à l'étude de tâches simples (transfert assis/debout, pointage) ; iii) un espace locomotion destiné à l'étude de la marche et équipé d'une plateforme de très grande dimension (180 cm x 90 cm) ; iv) un espace musique équipé d'un piano numérise et d'un système de sonorisation, et qui vise à étudier l'effet stimulation sonores sur le mouvement ; et v) un espace clinique géré par le service de gérontologie de l'hôpital Paul Brousse. Une thèse de sciences et deux stages de master sont réalisés sur site, auxquels s'ajoutent deux projets mobilisant conjointement les compétences du laboratoire CIAMS et des services de l'APHP. Le premier vise à évaluer l'effet du traitement par prothèse discale du patient lombalgique chronique sur la capacité posturo-cinétique (collaboration Hôpital Béclère, Pr JC Auregan), tandis que le second s'intéresse à l'effet des surfaces inclinées sur le contrôle postural des patients seniors (collaboration Hôpital Paul Brousse, Pr E Duron). Pour ces projets relevant d'une demande d'agrément auprès du comité de protection des personnes (CPP), l'université Paris-Saclay agira pour la première fois en tant que promoteur (catégorie RIPH3). Il est attendu de ces projets conjoints qu'ils permettent à la fois la validation de modèles du contrôle moteur dans un contexte clinique, et l'émergence de techniques innovantes au service du patient. Contacts : Jean Jeuvrey (jean.jeuvrey@universite-paris-saclay.fr), Alain Hamaoui (alain.hamaoui@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI CIAMS / Plateforme Paul Brousse : Fruit d’un partenariat entre l’AP-HP et l’Université Paris-Saclay, cette plateforme associée au CIAMS est localisée à l’hôpital Paul Brousse (Villejuif), avec pour objectif de développer l’analyse du mouvement appliquée à la santé, notamment la marche et la posture chez des sujets sains et pathologiques. Pour se faire, de multiples équipements sont utilisés dont 3 plateformes de forces AMTI ainsi que des électromyographes (EMG) sans fil. Les plateformes forces permettent des analyses fines des forces de réaction sur un espace au sol dépendant de la taille de la plateforme. La plus grande plateforme occupe un espace de 180 cm de long et de 90 cm de large. La plus petite occupe un espace de 60 cm sur 60 cm et est plutôt réservée à l’étude de la posturologie (i.e. les mécanismes de gestion de l’équilibre). A propos du CIAMS. Le CIAMS est un laboratoire multidisciplinaire (SDV et SHS) en sciences du mouvement et facteurs humains. Il contribue notamment au réseau Neurosciences, Mouvement, Handicap des unités de recherche en Sciences de la Vie de l'Université Paris-Saclay. Le CIAMS est un membre fondateur de la Fédération Demenÿ-Vaucanson (FéDeV), une Structure Fédérative de Recherche impliquant une quinzaine d’unités de recherche, dont la mission est de structurer la communauté scientifique de Paris-Saclay dans le domaine des sciences du mouvement.

|
Scooped by
Life Sciences UPSaclay
June 22, 2023 5:08 PM
|
Alors que les analyses en cellule unique arrivent seulement dans le domaine végétal, POPS, la plateforme de Transcriptomique des Plantes de Paris-Saclay (Institute of Plant Sciences Paris-Saclay, Univ Evry, INRAE, CNRS, Université Paris-Saclay, Université de Paris) est largement impliquée dans une étude visant à détailler le comportement individuel des cellules d’une feuille d’Arabidopsis thaliana lors d’une infection par la bactérie phytopathogène Pseudomonas syringae. En analysant le transcriptome de milliers de cellules par la technologie 10X genomics, nous avons pu mettre en évidence l’hétérogénéité des réponses végétales au sein de différentes populations cellulaires, permettant ainsi le dessin d’un nouvel et original champ de bataille entre plante et pathogène. Un résultat remarquable est notamment que les réponses de différents types cellulaires comme celles des cellules de l’épiderme et du mésophylle peuvent suivre deux voies distinctes. La première leur est propre et se caractérise par la mise en place de processus spécifiques guidées par l’activation de facteurs de transcription particuliers et contribuant à l’échelle de la feuille à des réponses coordonnées. La seconde voie de réponse est commune et permet à différents types cellulaires de converger vers un état « immunitaire » caractérisé par la synthèse de lignine, de phytoalexines et de l’acide salicylique, une hormone bien connue du stress. Plus généralement cette approche single-cell fournit une richesse inédite d’informations biologiques, apportant des éléments innovants à des questions fondamentales ayant trait à la totipotence immunitaire des cellules végétales, à la distinction entre réponses autonomes et non autonomes à un stress biotique ou encore à la prédiction du degré de résistance d’une plante à un pathogène. En savoir plus ? Delannoy et al., bioRxiv 2023. Par ce travail qui se poursuit encore actuellement avec l’analyse single-cell de mutants de plante immunodéficients, la plateforme POPS démontre sa capacité à accompagner ses collaborateurs dans ces projets hautement techniques depuis le design expérimental jusqu’à l’interprétation des données produites. Contact : pops.ips2@universite-paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI IPS2 / Plateforme de transcriptomique (POPS). La Plateforme de Transcriptomique des Plantes de Paris-Saclay (POPS) propose aux laboratoires académiques ou privés des outils d'analyse du transcriptome par séquençage à haut débit de l'ARN ou RNA-seq. Nous sommes spécialisés dans l'analyse des plantes qu'elles soient modèles ou de culture. Le service proposé inclue la génération des données de RNA-seq, les analyses bio-informatiques et statistiques ainsi que des conseils sur l'interprétation des résultats. Nous accompagnons donc nos collaborateurs depuis le design expérimental jusqu'à l'interprétation des données. La plateforme est certifiée pour l'ensemble de ses activités selon les exigences de la norme ISO 9001 depuis 2012. Afin d'offrir à chaque collaborateur l'analyse la plus adaptée à ses questions biologiques, nous disposons des équipements et des outils d'analyses bio-informatiques et statistiques permettant la construction de transcriptome de novo, l'analyse d'accumulation des ARNs poly-adenylés ou non, des petits ARNs (miRNAs, siRNAs, ribo-seq..) et des ARN poly-adénylés de de très petite quantité de départ (jusqu'à 50pg). Pour les analyses d'expression tissu-spécifiques, nous offrons également des analyses de transcriptome après micro-dissection laser ou en cellule unique grâce à la technologie Chromium (10x Genomics). Par ailleurs, nous pouvons offrir à nos collaborateurs de sessions de formation sur une semaine à l'analyse statistique des données RNA-seq.

|
Scooped by
Life Sciences UPSaclay
June 10, 2023 6:39 AM
|
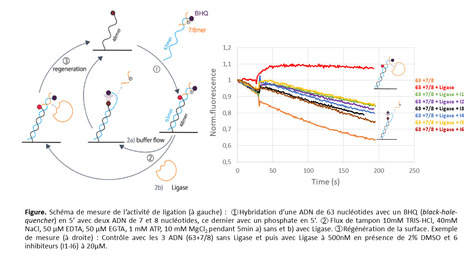
La plateforme I2BC / Plateforme de mesures d'Interactions Macromoléculaires (PIM) et l’équipe du département B3S, Intégrité du génome (IntGen) (Institut de Biologie Intégrative de la Cellule (I2BC), Gif-sur-Yvette, Institut Joliot, CEA, Saclay) proposaient déjà à ses utilisateurs la technologie switchSENSE. Cette technique, qui utilise des nanoleviers d'ADN fixés sur une surface d'or, permet de mesurer les interactions macromoléculaires, notamment protéine-ADN et protéine-ARN. Elle est complémentaire à deux autres méthodes de mesures en surface aussi disponibles sur la plateforme, la BioLayer interférométrie (BLI) et la résonance plasmonique de surface (SPR). En janvier 2023, l’I2BC a effectué une mise à jour de l'instrument switchSENSE (DRX2, Dynamic Biosensors) de première génération (Muller-Landau and Varela, Eur Biophys J 2021) par un instrument heliX+ de nouvelle génération (Dynamic Biosensors). Une valeur sûre validée et utilisée régulièrement par l’équipe IntGen. Vous avez dit HeliX+ (Dynamic Biosensors) ? une première sur le territoire français ! Ce nouvel instrument possède des caractéristiques uniques en particulier pour les protéines de liaison à l'ADN et à l'ARN (Marcelot et al, Nucl Acids Res 2021). Il peut résoudre les cinétiques rapides en toute confiance grâce à une microfluidique avancée avec deux circuits séparés (tampon, lavage), et à la collecte de données toutes les 10 ms (Nemoz et al, Nat Struct Mol Biol 2018 ; Velours et al, Eur Biophys J 2021). Les interactions moléculaires peuvent être mesurées jusqu'à des affinités femto-molaires (fM). Il permet le criblage en plaques (96 et 384 puits) et l'analyse des changements conformationnels induits par les petites molécules. Il mesure les activités enzymatiques dont celles des polymérases, des ligases et des nucléases. L'équipe a récemment mis au point un test d'activité de la Ligase4 humaine qui est impliquée dans la réparation des cassures double-brin de l'ADN. Nous avons pu analyser l'activité inhibitrice de premières molécules identifiées au laboratoire (Figure 1). Le schéma expérimental porte sur l'hybridation de deux oligonucléotides de 7 et 8 nucléotides sur un brin complémentaire de 63 nucléotides qui porte un quencheur (BHQ). Si ces oligos sont liés par la Ligase4, la sonde de fluorescence est éloignée du quencheur et fluoresce. Lorsqu'un flux passe sur le senseur, si les deux oligos ne sont pas liés, ils se dissocient progressivement du brin complémentaire, on a alors une extinction de la fluorescence. Quand la Ligase4 a lié les deux oligos, la sonde fluoresce et le signal est stable. Nous avons pu étudier une dizaine d'inhibiteurs et valider l'action de certains d’entre eux. Des expériences en FRET sont également possibles via cet équipement et grâce à la présence d'un double détecteur de fluorescence. Les biopuces du heliX+ ont été optimisées avec un gain en stabilité et en spécificité grâce à des nouveaux adaptateurs (type origami, structure Y). La densité du ligand sur la surface est facilement ajustable selon le type d’interaction à mesurer. Jusqu’à cinq puces peuvent être chargées en même temps permettant de programmer des expériences en série. Le logiciel a été aussi fortement amélioré. Il est plus intuitif et efficace pour programmer et analyser les expériences. Le Helix+ a été obtenu grâce au soutien de l’INBS FRISBI, du comité plateforme de l’I2BC et de son département B3S. Contact : Paloma Fernández Varela (paloma.fernandez-varela@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI I2BC / Plateforme de mesures d'Interactions Macromoléculaires (PIM). La Plateforme de mesures d'Interactions Macromoléculaires (PIM) de l'I2BC offre un large éventail d'équipements et d'expertises en biochimie et biophysique pour réaliser des mesures de contrôles qualités ainsi que des analyses fonctionnelles d'échantillons de protéines : microcalorimétrie, ultracentrifugation analytique (UCA), MST, SEC-MALS, SPR, switchSENSE. Les prestations offertes par la plateforme concernent 1) La caractérisation biophysique de protéines ou de complexes biologiques (SEC-MALS, UCA), 2) L'analyse d'interactions biologiques (ITC, SPR, BLI, switchSENSE, MST), 3) L'analyse de la stabilité des macromolécules et des complexes biologiques pour notamment aider à la formulation (DSC, DSF). En complément, 4) Une nouvelle activité est proposée sur la plateforme : La génération et production de protéines artificielles (αRep) qui fixent avec une affinité et une spécificité élevée n'importe quelle protéine cible choisie. Les appareils et prestations sont mis à disposition de l'ensemble des laboratoires de l'Université Paris-Saclay, des autres laboratoires académiques ainsi qu'aux industriels. Pour plus d'informations concernant ces prestations : stephanie.marsin@i2bc.paris-saclay.fr. Cette plateforme fait partie du pôle des plateformes de Biologie Structurale de l'I2BC qui comprend : i) les plateformes de Cristallisation, RMN, CryoEM, Mesures d'Interactions Macromoléculaires, et ii) les plateaux techniques d'Expression de protéines solubles ou membranaires en levures, Expression des protéines en cellules d'insectes. Envie de (re)lire ses précédents FOCUS PLATEFORME ? A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 60 équipes de recherches et 15 plateformes technologiques, provenant de 8 unités de recherches (CGM, IBBMC, IGM, ISV, LEBS, VMS, SB2SM, SBiGeM). L’institut est réparti sur 3 sites de recherche (Campus d’Orsay Vallée de l’Université Paris-Saclay, Campus du CNRS de Gif sur Yvette et Campus du CEA / Centre de Saclay) au sein de 14 bâtiments jusqu’au rassemblement programmé sur le campus du CNRS de Gif-sur-Yvette.

|
Scooped by
Life Sciences UPSaclay
May 28, 2023 5:17 PM
|
PAPPSO est une plateforme experte dans l'analyse des protéomes. Implantée sur deux sites, à l'institut MICALIS (UMR INRAE, AgroParisTech, UPSaclay - Jouy-Josas) et à l'UMR Génétique Quantitative et Evolution-le Moulon (INRAE, AgroParisTech, CNRS, UPSaclay - Gif-sur-Yvette), elle est largement ouverte à la communauté scientifique Paris-Saclay. La carte de visite de PAPPSO est aujourd’hui sa capacité unique à analyser de grandes cohortes (>100 échantillons) et des échantillons très complexes (e.g. métaprotéomique du microbiote intestinal). Elle est également fortement engagée dans le développement de méthodes analytiques et d'outils bioinformatiques. PAPPSO débute l'année 2023 sur un nouvel élan, après une année 2022 particulièrement riche en évènements et opportunités. Un nouveau binôme de responsables à la tête de PAPPSO. Suite aux départs à la retraite des deux fondateurs de PAPPSO, Michel Zivy et Véronique Monnet, le management de la plateforme a été récemment repris par deux ingénieures de recherche de PAPPSO. Il s’agit de Mélisande Blein-Nicolas en tant que responsable scientifique localisée sur le site de Gif-sur-Yvette, et de Céline Henry en tant que responsable technique localisée sur le site de Jouy-en-Josas. Mélisande et Céline se sont préparées pour prendre en main leurs nouvelles responsabilités et insufflent déjà une nouvelle dynamique dans PAPPSO. Elles auront à cœur de maintenir PAPPSO à un haut niveau de compétences et de qualité en tant qu’infrastructure scientifique collective bien visible dans le paysage de la protéomique française et internationale. De nouveaux labos pour le site de Gif-sur-Yvette. Le 15 février 2022, le site de Gif-sur-Yvette quittait la Ferme du Moulon pour investir les locaux flambant neufs de l'IDEEV (Institut Diversité Ecologie et Evolution du Vivant) situés à quelques centaines de mètres (voir illustration – haut). Le déménagement des deux spectromètres de masse, instruments extrêmement sensibles, a été une véritable aventure qui s'est déroulée dans les meilleures conditions. Les appareils sont à présent bien installés dans une pièce spacieuse et lumineuse qui leur fournit d'excellentes conditions de fonctionnement. Une nouvelle ingénieure en poste fixe sur le site de Jouy-en-Josas. Fin 2022, une nouvelle ingénieure analyste en spectrométrie de masse, Carine Machado Rodrigues, a rejoint l'équipe du site Jouy-en-Josas sur un poste fixe. Carine était en CDD sur la plateforme depuis novembre 2021. La pérennisation de son poste est une excellente nouvelle pour PAPPSO qui attendait depuis 2017 de remplacer un ancien analyste parti en mobilité. PAPPSO entame donc l'année 2023 avec une équipe renforcée (voir illustration – bas – de gauche à droite M. Blein-Nicolas, C. Henry, T. Balliau, M. Davanture, L. Oliveira Correia, W. Bienvenut, C. Machado Rodrigues, O. Langella et F. Rusconi). De nouveaux projets de développement pour PAPPSO. La plateforme a obtenu pour 2023 un financement de l'institut Carnot Plant2Pro pour développer la protéomique végétale à haut débit. Depuis une dizaine d'années, la plateforme reçoit de plus en plus fréquemment des demandes d'analyse de grandes cohortes d'échantillons végétaux, toutes liées à des projets de recherche en amélioration végétale ou en agro-écologie. Par le passé, PAPPSO s'est déjà illustrée pour sa capacité à traiter un grand nombre d'échantillons dans deux projets phares : le PIA Amaizing avec plus de 1000 échantillons de maïs traités et l'ANR Proteocardis avec près de 500 échantillons du microbiote intestinal humain analysés. Cependant, l'accroissement des demandes d'analyse de grandes cohortes nécessite que la plateforme augmente fortement ses débits d'analyse pour éviter l'engorgement. Le financement obtenu permettra le recrutement d'un ingénieur d'étude pendant un an et de deux stagiaires afin de mener les développements analytiques nécessaires. Ces dernières années, on note également un engouement croissant pour les études sur le rôle des vésicules extracellulaires (VE). Longtemps négligées car préposées au transport des débris cellulaires, ces VE ont été décrites comme jouant un rôle dans le transport de matériel biologique entre cellules. PAPPSO s'est engagée dans des développements analytiques pour l'étude des VE, dont les protéines en très faibles quantités sont difficiles à détecter par spectrométrie de masse. Ces développements ont été initiés dans le cadre de projets sur l’étude des protéines de vésicules plasmatiques (Laurent Galio, UMR BREED, Jouy-en-Josas) et de vésicules utérines (Laurent Galio et Esther Dos Santos, UVSQ). Des études se poursuivent dans le cadre d'une collaboration avec les chercheurs de l'équipe MuSE (Mariane DePaepe, UMR Micalis, Jouy-en-Josas) visant à étudier la composition protéique de vésicules des bactéries du microbiote intestinal chez des patients atteints de la maladie Crohn. Aussi, le 25 novembre 2019, la plateforme publiait son premier FOCUS PLATEFORME ! Plateforme d'Analyses Protéomiques de Paris Sud-Ouest (PAPPSO) : de la Protéomique à la Métaprotéomique, à la recherche de biomarqueurs ! Le relire ? Contact : Céline Henry (celine.henry@inrae.fr) ; Mélisande Blein-Nicolas (melisande.blein-nicolas@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Plateforme d’Analyse Protéomique de Paris Sud-Ouest (PAPPSO). L'objectif de la Plateforme d'Analyse Protéomique de Paris Sud-Ouest (PAPPSO) est de mettre à disposition des équipes qui s'adressent à elle un équipement, une compétence scientifique et un savoir-faire adaptés aux questions posées, depuis les plus simples (identification de protéines provenant d'un organisme entièrement séquencé) jusqu'aux plus complexes (quantification relative ou semi-absolue des variations, dynamique des modifications post-traductionnelles,...), dans le cadre de collaborations et de prestations. PAPPSO réunit deux plateaux techniques complémentaires. L'un, adossé à l'UMR Génétique Quantitative et Evolution ? Le Moulon (Gif-sur-Yvette), est spécialisé dans la biologie végétale (espèces modèles et cultivées), et l'autre, adossé à l'UMR Microbiologie de l'Alimentation au service de la Santé (MICALIS, Jouy-en-Josas), est spécialisé en microbiologie, biologie animale (modèles bovin, murin) et, plus ponctuellement, en biologie humaine. Ses équipements de toute dernière génération permettent à PAPPSO de proposer la réalisation d'analyses par spectrométrie de masse, avec ou sans pré-fractionnement des protéines ou des peptides, et avec ou sans marquage isotopique. PAPPSO s'est spécialisée dans le haut débit (analyse quantitative de cohortes de grande taille), dans l'analyse d'échantillons très complexes (métaprotéomique), et dans la peptidomique. Elle développe les outils de bioinformatique et d'analyse de données qui permettent de traiter ce type d'expérience, et accompagne les utilisateurs dans l'interprétation de leurs données.

|
Scooped by
Life Sciences UPSaclay
May 13, 2023 5:11 PM
|
L’IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT se compose de 11 plateformes technologiques, organisées en trois pôles (OMICS, INTERACTIONS et IMCELLF) et trois plateformes transverses (Animex, Bio-info et Verre-Scien-Tech). Résolument à l’interface de la chimie, de la biologie et de la clinique, IPSIT est le lien entre la cible pathologique et le médicament ! En effet, le processus de découverte d’un médicament requiert une approche multidisciplinaire associant des savoir-faire complémentaires, notamment dans les domaines de la chimie, pharmacotechnie, immunologie, biologie structurale, biologie moléculaire et cellulaire, sans oublier la génétique et la pharmacologie. L’IPSIT, par ses plateformes, apporte aux équipes une aide scientifique essentielle dans ce processus. IPSIT participe aussi à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Aussi, depuis la rentrée 2022, IPSIT a pris possession de ses nouveaux locaux, et toutes ses plateformes sont maintenant réunies dans le bâtiment Henri Moissan du pôle Biologie-Pharmacie-Chimie de l’Université Paris-Saclay, situé sur le plateau de Saclay à Orsay. Enfin, IPSIT et ses plateformes prennent régulièrement RDV avec vous au travers de FOCUS PLATEFORME. Profitez de cette brève pour découvrir à nouveau un éclairage sur leurs expertises, leurs équipements et leurs offres de collaborations ou de prestations ! - i) Pôle OMICS
Plateforme Transcriptomique et génomique (ACTAGen) : Du développement technologique d'une puce dédiée à l'exploration du transcriptome d'un champignon, à un contrat industriel : retour sur une success story ; ACTAGen, plateforme de transcriptomique et génomique et vecteur de la démarche qualité à l’UMS-IPSIT Plateforme de spectrométrie de masse & lipidomique (SAMM) : SM@BPC : une plateforme de spectrométrie de masse sur Paris-Saclay à la hauteur des enjeux du pôle Biologie – Pharmacie – Chimie (BPC) de l'Université Paris-Saclay ! ; AAP SESAME 2021 : Un premier succès pour la communauté Paris-Saclay et le consortium SMaCS « Spectrométrie de Masse pour la Chimie et la Santé » ! Plateforme de protéomique (PROTEOMIC) : Détournement d'antibiotique à doses sub-inhibitrices par Clostridium difficile : une enquête conjointe de l'équipe BaPS / MICALIS et de la plateforme de protéomique de l'IPSIT ! - ii) Pôle INTERACTIONS
Plateforme de criblage moléculaire (CIBLOT) : CIBLOT - Une plateforme proposant la recherche d'inhibiteurs d'interaction Protéine-Protéine à haut débit Plateforme Interactions moléculaires (INTERMOL) : INTERMOL - Une plateforme dédiée à l'étude des interactions moléculaires pour le développement de thérapies innovantes iii) Pôle IMCELLF Plateforme imagerie cellulaire (MIPSIT) : La microscopie photonique Super-Résolue : Une nouvelle dimension au service de l'innovation thérapeutique Plateforme Pathologie expérimentale & télé-expertise (PHIC) : De la lame virtuelle à la caractérisation de mécanismes pathologiques et l'identification de pistes thérapeutiques ! Plateforme Immunomonitorage par cytométrie de flux (PLAIMMO) : PLAIMMO : une plateforme spécialisée en cytométrie de flux, à votre écoute ! ; PLAIMMO met en service un nouveau lecteur haute performance : Un QuickPlex® SQ 120 (Meso Scale Discovery, MSD) ! ; Le tri de cellules infectées, humaines ou transfectées : de nouvelles possibilités offertes aujourd'hui par PLAIMMO ! - iv) plateformes transverses
Plateforme Bio-informatique (BIO-INFO) : La reproduction de résultats publiés, une méthode incontournable en bioinformatique ! Plateforme Animalerie & exploration fonctionnelle (ANIMEX-1 et ANIMEX-2) : Quand les rats de la plateforme AnimEx se mettent au service des athlètes ; Des souris, des hommes et leurs microbiotes… une expertise forte à AnimEx ! Plateforme Verrerie scientifique & technique (VERRE SCIEN-TECH) : Le soufflage de verre et la science, une histoire d’alchimie et une plateforme vraiment unique sur le territoire Paris-Saclay ! Contacts : Valerie Domergue (valerie.domergue@universite-paris-saclay.fr) ; Anne Garnier (anne.garnier@universite-paris-saclay.fr) IPSIT : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
May 5, 2023 6:07 PM
|
La plateforme de phénotypage de l'Observatoire du Végétal à l'Institut Jean-Pierre Bourgin (IJPB, INRAE-Versailles) est fondée sur l'utilisation de robots « Phenoscope » conçus pour cultiver et phénotyper des plantes de petite taille (typiquement Arabidopsis thaliana) au stade végétatif. Cet outil permet en particulier de suivre de façon dynamique la croissance de la partie aérienne de la plante tout en maitrisant l'environnement abiotique (température, hygrométrie, lumière, CO2, arrosage, nutriments --en particulier azotés--...) et de minimiser les effets de ses variations sur le développement des plantes. Ceci permet de réaliser des expériences combinant haut-débit, homogénéité et reproductibilité, qui ne sont pas faisables « à la main ». Ces outils sont particulièrement utiles pour les plans expérimentaux de grandes tailles ou complexes, comme ceux permettant le phénotypage de nombreux génotypes et/ou de multiples conditions environnementales (en particulier contrôlées via l'arrosage), mais aussi pour la production de matériel végétal homogène en conditions contrôlées, par exemple pour assurer la qualité d'analyses omiques ultérieures (transcriptomique, métabolomique, protéomique...) avec un nombre raisonnable de réplicats. Ces dernières années, la plateforme IJPB / Observatoire du Végétal – Phenoscope a travaillé avec une société privée extérieure pour concevoir un nouveau Phenoscope (appelé « Phenoscope XL ») qui permettra de cultiver des plantes de petite taille au-delà du stade végétatif et en particulier sur un cycle reproducteur complet (de la graine à la graine). Il s'agit d'une demande importante de notre communauté qui désire aussi produire des graines dans des conditions plus reproductibles. Aussi, si la partie mécanique du robot est maintenant fonctionnelle, nous avons choisi de reprendre totalement le programme informatique de supervision afin de le moderniser, le sécuriser et le rendre adaptable aux 2 types de Phenoscope de la plateforme : en effet les Phenoscopes « classiques » bénéficieront également de ces avancées informatiques qui sécuriseront grandement notre fonctionnement et la sauvegarde des données associées dans notre base de données. Le Phenoscope XL devrait être totalement opérationnel d'ici la fin de l’année 2023, mais n’hésitez pas dès à présent à nous contacter pour profiter de notre expérience en matière d'interactions plante - environnement abiotique ! Contacts : Olivier Loudet (olivier.loudet@inrae.fr); Christian Meyer (christian.meyer@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IJPB / Observatoire du Végétal – Phenoscope. Le Phenoscope est un outil de phénotypage haut-débit dédié à la culture et à l'observation de centaines de plantes, cultivées dans des pots individuels. Il est typiquement exploité pour le suivi de la croissance végétative d'un grand nombre de génotypes sous différents régimes d'arrosage. Les pots tournent en continu (cycle typique de 4 heures) le long d'un circuit fermé consistant en une succession d'allers-retours, ce qui permet d'exposer plus de 700 plantes à des conditions très homogènes. Des stations de pesée/arrosage sont utilisées pour suivre et ajuster la disponibilité en eau et en éléments nutritifs selon des scénarios précis définis par l'utilisateur pour chaque pot et chaque cycle. Des paramètres phénotypiques sont acquis au niveau des stations d'imagerie (visible et infra-rouge) de façon automatisée et non-destructive. Le Phenoscope est particulièrement étudié pour gérer des plantes de petite taille (Arabidopsis) au long de leur phase végétative (mais le Phenoscope XL -en développement- permettra bientôt de poursuivre au-delà de l'initiation florale). Nous avons montré que le gain moyen permis par l'utilisation des Phenoscopes était d'un facteur 3 environ (en terme de réduction de l'hétérogénéité environnementale ou du nombre de répétitions nécessaires). Les Phenoscopes sont idéalement exploités dans des projets de génétique quantitative (QTL, GWAS), des screens de mutants, ou toute autre expérience à grande échelle nécessitant la comparaison de centaines d'individus dans différents environnements. Ils peuvent aussi être utiles pour une caractérisation détaillée d'un petit nombre de génotypes (en particulier en comparaison avec d'autres jeux de données disponibles dans notre base de données) et pour l'établissement de courbes de réponse au traitement, où plusieurs niveaux graduels de stress sont appliqués. Il n'existe que 3 de ces robots dans la communauté, tous situés à l'IJPB (INRAE site de Versailles) au sein de l'Observatoire du Végétal. A propos de l’Institut Jean-Pierre Bourgin (IJPB). L'Institut Jean-Pierre Bourgin est un des plus grands centres de recherche européens dans le domaine de la biologie des plantes. Il regroupe un ensemble de ressources et de compétences pluridisciplinaires en biologie, chimie et mathématiques uniques en France. L'IJPB est une unité mixte de recherche (UMR1318) sous la tutelle conjointe INRAE et AgroParisTech et appartenant à l’Université Paris-Saclay. L'IJPB est également membre fondateur de l’Ecole Universitaire de Recherche (EUR) Saclay Plant Sciences (SPS). Son but : Développer une recherche pluridisciplinaire sur le végétal pour produire des connaissances fondamentales en biologie, les inscrire dans un processus d’innovation et répondre ainsi aux défis scientifiques et sociétaux à venir. L'IJPB s'intéresse notamment à : i) l'évolution et le fonctionnement des génomes végétaux ; ii) la réponse des plantes aux contraintes de leur environnement et la biodiversité associée ; iii) les mécanismes régissant le développement, la signalisation et la communication des plantes à différentes échelles, de la cellule à la plante entière jusqu’à la graine ; iv) la modélisation des phénomènes biologiques complexes dans un but prédictif et v) la caractérisation du métabolisme végétal et de ses produits (celluloses, lignines, lipides et métabolites spécialisés) pour l’agroécologie et une bioéconomie durable.

|
Scooped by
Life Sciences UPSaclay
April 7, 2023 8:15 AM
|
Avec l’augmentation des demandes à l’interface biologie et matériaux, et le développement de nouvelles sondes permettant l’imagerie dans le proche infra-rouge (au-delà de 850 nm), le Centre de Photonique pour la Biologie et les Matériaux (CPBM, ISMO, UMR8214, CNRS - Université Paris-Saclay) n’était plus en mesure de répondre à toutes les demandes qui lui étaient faites ! En effet, que ce soit les mesures de diffusion (FCS, FRAP) ou l’imagerie de durée de vie (FLIM), la détection dans le proche infra-rouge demeure actuellement un frein à ces recherches. La fenêtre proche infrarouge (NIR), également appelée fenêtre optique ou fenêtre thérapeutique, définit la plage de longueurs d'onde de 650 à 1350 nm où la lumière a une profondeur de pénétration maximale dans les tissus biologiques. Ce type de détection et d’analyse en microscopie n’est pas accessible en plateforme en France et n’est disponible que dans un nombre restreint d’équipes de recherche. CoCom : un projet d’équipement en réponse à ce déficit ! CoCoM (pour Confocal Correlatif Multimodal) doit permettre une ouverture à la communauté de ces techniques, en s’appuyant sur l’expertise présente sur la plateforme CPBM et à l’ISMO dans ce domaine. Le couplage de ce système avec un module de super résolution dSTORM 3D, grâce au partenariat avec la société Olympus/Evident sera un puissant levier permettant de répondre aux problématiques en santé ou énergie de la communauté, intégré dans un environnement scientifique complet. Il est à noter qu’il existe aussi une forte demande en science des matériaux, que ce soit des équipes de recherche universitaire mais aussi pour nos utilisateurs du secteur privé. CoCoM peut aussi être considéré comme un équipement complémentaire des autres dispositifs présents sur le périmètre de l’Université Paris-Saclay, par exemple dans le cas du développement de l’Imagerie Photo Acoustique qui combine excitation lumineuse et ondes sonores pour, notamment, suivre, in vivo, l'injection de médicaments encapsulés dans des nanoparticules. A l’aide de CoCoM, l’analyse de la distribution tissulaire et cellulaire sur des coupes histologiques de tissus deviendra possible pour valider les images photoacoustiques obtenues grâce au système Vevo LAZRX acquis par l'UMR-S 1180 (INSERM/UPSaclay, Orsay) en collaboration avec l’Institut Galien Paris-Saclay - IGPS (équipement également acquis grâce à une dotation SESAME de la région, 2019), réalisant de l’imagerie photoacoustique. Soumis à l’appel à projets régional SESAME 2022 pour un budget total de 445 k€, CoCoM fait aujourd’hui partie des 4 projets UPSaclay sélectionnés par la région Île-de-France, et recevra à ce titre une subvention de 245 k€. CoCoM a déjà obtenu les compléments de financement par différents AAP et partenaires (le GIS-IBiSA, l’Université Paris-Saclay, l’ISMO, le PPSM, l’IGPS, l’ICMMO). Ces financements vont donc permettre de finaliser l’acquisition d’un microscope confocal i) multimodal permettant l’imagerie spectrale de fluorescence confocale (ISC), l’imagerie de durée de vie de fluorescence (FLIM) et la spectroscopie de corrélation de fluorescence (FCS), ii) disposant d’un large domaine spectral dans le proche Infra-Rouge (IR), et iii) évolutif, permettant, d’intégrer des outils de super-résolution et de réaliser des développements technologiques pour l’acquisition de spectre d’excitation et une détection élargie. Cette combinaison de caractéristiques en fera un équipement unique en France et en Europe, accessible à une large communauté de scientifiques, et permettra par exemple de répondre aux demandes grandissantes dans le domaine de la santé incluant notamment les matériaux biomimétiques, la cartographie super-résolue de sondes moléculaires en milieux biologiques, le suivi de nanoparticules et nano-médicaments, ou l’imagerie cellulaire. Le microscope répondra aussi aux besoins de caractérisation d’interfaces ou de surfaces très présents chez les utilisateurs de la plateforme, notamment dans le domaine de l’énergie et du développement durable : photocatalyse, photosynthèse artificielle, photovoltaïque et nouveaux catalyseurs. Aussi, le 23 janvier dernier, le Centre de Photonique pour la Biologie et les Matériaux publiait son premier un Scoop-it® / FOCUS PLATEFORME : Le relire ? Contact : Ludivine Houel-Renault (ludivine.houel-renault@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI ISMO UMR 8214 / Centre de Photonique pour la Biologie et les Matériaux (CPBM). Le Centre de Photonique pour la Biologie et les Matériaux (CPBM) est une plateforme de l'Institut des Sciences Moléculaires d'Orsay (ISMO - UMR8214). C'est une plate-forme technologique d'imagerie de la dynamique de fluorescence ouvertes à des utilisateurs tant académiques qu'industriels. L'ensemble de l'activité de recherche du centre est structuré en 2 thèmes principaux : i) imagerie de fluorescence multimodale pour des applications biomédicales, environnementales et en chimie et ii) développements en instrumentation optique pour des études de surfaces en chimie et biologie.

|
Scooped by
Life Sciences UPSaclay
March 24, 2023 1:08 PM
|
La plateforme « épigénomique et recherche translationnelle » (EPITRANS) de l’IPS2 (Institute of Plant Sciences Paris-Saclay, UEVE/INRAE/CNRS/UPSaclay/Université de Paris, Gif-sur-Yvette) est une infrastructure scientifique collective (ISC) de l’INRAE, labellisée IBISA et certifiée Iso9001. Elle a pour mission de développer et de mettre à disposition de la communauté scientifique des outils de génétique haut-débit, directe et inverse, chez les espèces cultivées. Elle propose d’identifier les gènes et les régulations épigénomiques majeurs contrôlant les caractères d’intérêts agronomiques puis à créer des allèles qui permettent d’améliorer la performance des plantes. Elle se positionne comme pierre angulaire entre la recherche fondamentale et la recherche appliquée, sur le territoire Paris-Saclay, mais aussi au niveau national et international. Depuis sa création, la plateforme EPITRANS exploite des collections de mutants chez le pois (Dalmais et al., Genome Biol. 2008) et plus récemment chez le lupin (ANR MicroLUP, projet-ANR-19-CE13-0029), deux espèces de légumineuses. Étant donné leur capacité à fixer l'azote atmosphérique, le développement de ces cultures est essentiel à la transition agro-écologique. Parmi les légumineuses, le soja est la première source de protéines par hectare pour l’alimentation animale et humaine. La France en importe massivement car elle n’est pas auto-suffisante en graines produites localement. Pour réduire cette dépendance et assurer les prévisions de croissance en surface de culture, il est nécessaire de créer des variétés non génétiquement modifiées adaptées aux climats européens qui favoriseront l’expansion vers le nord tout en répondant aux futurs besoins alimentaires. C’est pourquoi, la plateforme s’engage, au travers de deux projets de recherche, SOYADAPT et SOYSTAINABLE, respectivement démarrés en 2022 et 2023, à créer deux collections de mutants induites. Ce travail est réalisé en collaboration avec des partenaires privés (dont RAGT et LIDEA) et publics (dont l’Institut de Recherche en Horticulture et Semences). L’objectif de la plateforme est d’obtenir 10 000 lignées M2 mutées, induites pour moitié par un agent chimique (l’EMS) et pour l’autre par un agent physique (les fast-neutrons). Parmi ces collections, la plateforme réalisera à la demande de ces partenaires de la détection de mutations dans des régions ciblées du génome (criblages TILLING). En savoir plus sur ces deux projets ? - SOYADAPT (2022-2025) - développement de variétés de soja adaptées au semis précoce par la création de nouvelles ressources génétiques et l'identification de nouveaux allèles de gènes cibles ; projet financé par le consortium PlantAlliance, coordonné par Julia Buitink, INRAE IRHS Angers.
- SOYSTAINABLE (2023-2029) - vers une source de protéines de soja local et durable en alimentation humaine résiliente au changement climatique ; projet financé par l’ANR (ANR-22-PLEG-0003), AAP Plan Investissement d’Avenir (PIA4) « Développer les protéines végétales et diversifier les sources de protéine » Volet 1 – protéines de légumineuses (Plan France 2030, France Relance), coordonné par Jean-Malo Couzigou, Université Toulouse Paul Sabatier III.
Contact : marion.dalmais@inrae.fr Plug In Labs Université Paris-Saclay : cliquer ICI IPS2 / Epitrans. La plateforme EPIgénomique et recherche TRANSlationnelle (EPITRANS), Infrastructure Scientifique Collective (ISC) de l'INRAE, a pour missions de valoriser la recherche fondamentale en transformant les idées en produits et de faire le lien entre le chercheur et le secteur économique. Elle s'intéresse aux allèles et épi-allèles qui améliorent la performance des plantes dans un environnement de plus en plus contraint et plus particulièrement chez les espèces d'intérêt agronomique. Son activité consiste à développer et à mettre à disposition de la communauté scientifique des outils de génétique haut-débit, forward et reverse, chez les végétaux, notamment lorsque les études traditionnelles de génétiques ne peuvent s'appliquer (transformation génétique). Cinq outils sont ainsi proposés : le clonage positionnel par NGS, le TILLING (« targeted Induced Local Lesion IN Genome ») et l'ECO-TILLING, l'Epigénomique et le CRISPR. Le premier permet de cloner des gènes d'intérêts agronomiques pour ensuite étudier leur(s) fonction(s) par TILLING, par recherche de modifications épigénomiques, ou encore par édition du génome via le système CRISPR (selon les espèces). Le TILLING s'appuie sur la production de larges collections (<5000 lignées) de plantes mutées ou de collections de germoplasmes (ECOTILLING) combinée à une identification rapide et systématique des mutations dans les séquences cibles. Ces allèles, non génétiquement modifiés, sont proposés aux sélectionneurs pour améliorer leurs lignées élites en leur offrant une alternative aux collections limitées de germoplasmes. C'est pourquoi, la plateforme a des liens étroits et durables avec le secteur industriel dans différents domaines d'applications (Limagrain, Gautier Semence, Rijk Zwaan, Symrise). EPITRANS est leader en Europe dans le domaine de la recherche translationnelle de par la richesse de ses collections de mutants (260 000 lignées) et par la diversité des espèces cultivées disponibles (13 au total). La plateforme est labellisée par le GIS-IBISA (Infrastructures en Biologie Santé et Agronomie) et est certifiée Iso9001.

|
Scooped by
Life Sciences UPSaclay
March 9, 2023 6:00 PM
|
L'Institut de Biologie Intégrative de la Cellule (I2BC CEA-Joliot/CNRS/UPSaclay) est une unité mixte de recherche soutenue par l'Université Paris-Saclay, le CNRS et le CEA, accueillant une soixantaine d’équipes de recherche et hébergeant 15 plateformes technologiques et 2 plateaux techniques, répartis en 6 pôles. Aujourd’hui, FOCUS PLATEFORME s’intéresse à une toute nouvelle modalité d’imagerie – SIM (Structured Illumination Microscopy) – récemment acquise par la plateforme d’imagerie photonique (Imagerie-Gif). Cette approche permet de combiner des acquisitions multi-couleurs super-résolues en 3D et une observation optimale de processus cellulaires dynamiques sans préparation spécifique des échantillons ; l’acquisition de ce nouvel équipement (ZEISS, Elyra 7 avec Lattice SIM²), d’une résolution sans précédent, s’inscrit dans un continuum de modalités proposées sur les plateformes Imagerie-Gif. SIM, lattice SIM … mais de quoi parle-t-on ? SIM – Structured Illumination Microscopy : Effet de Moiré et transformée de Fourier pour une meilleure résolution. Un des enjeux majeurs en microscopie photonique est l’amélioration de la résolution dans les trois dimensions de l’espace (XY et Z) afin d’accéder à la localisation de structures la plus précise possible à l’échelle cellulaire, sub-cellulaire et moléculaire. Engendrée par l’interaction lumière-matière (sources lumineuses - éléments optiques du microscope), l’imagerie photonique se confronte au phénomène de diffraction qui vient entacher l’image et donc son contraste. Parmi les stratégies possibles pour dépasser cette limite, une solution est de passer dans le domaine fréquentiel (Transformée de Fourier). C’est le principe de l’illumination structurée conventionnelle ou SIM (Structured Illumination Microscopy) qui est basée sur l’utilisation d’un pattern d’illumination pour visualiser un échantillon biologique. L’interaction de deux éléments structurés (pattern d’illumination +échantillon biologique) produit un effet de moiré (cf. figure) qui a pour conséquence de rendre observable des détails (hautes fréquences) qui étaient indiscernables en microscopie conventionnelle. Afin de rendre homogène l’apparition de ces hautes fréquences sur l’intégralité du champ observé, il est nécessaire de déplacer le pattern d’illumination selon 9 à 15 positions différentes (rotations ou translations) appelées phases. L’image finale super-résolue est obtenue en intégrant les hautes fréquences révélées par toutes les phases d’illumination (9 à 15 acquisitions) grâce à un algorithme de reconstruction. Lattice SIM : Une nouvelle stratégie d’illumination pour une meilleure résolution et une meilleure préservation de l’échantillon. Le Lattice SIM est une nouvelle technologie développée par la société ZEISS qui consiste à moduler spatialement l’illumination pour obtenir un pattern en points (maillage ou treillis : lattice en anglais). Comme dans le cas du SIM conventionnel, le motif d’illumination est également déplacé afin d’obtenir les images nécessaires à l’étape de reconstruction mathématique. Ce nouveau motif de points en treillis procure plusieurs avantages. Outre le fait que le sectionnement optique (observation de l’échantillon dans l’épaisseur, selon l’axe Z) est possible avec les deux modes SIM et Lattice SIM, ce dernier permet d’améliorer sensiblement le contraste et la résolution en XY (120nm). Il est également plus robuste dans la dimension Z car il se propage mieux dans la profondeur de l’échantillon. La résolution axiale en est augmentée et ce à plusieurs microns de la lamelle. Enfin, l’échantillonnage de l'éclairage en Lattice SIM est deux fois plus élevé que celle du SIM classique mais beaucoup moins redondant. La dose de laser nécessaire pour éclairer l'échantillon est par conséquent réduite. Ce qui est optimal notamment pour le suivi de processus dynamiques sur cellules vivantes. Des acquisitions sur du plus long terme sont possibles car le photoblanchiment (perte de fluorescence due à l’illumination) et la phototoxicité sont limités. En résumé… L’illumination structurée de type Lattice SIM permet de réaliser : i) L’acquisition d’images de fluorescence avec une résolution de 120nm XY (et 60nm après déconvolution SIM²) ; ii) L’imagerie super-résolue multi couleur (jusqu’à 4 couleurs) ; iii) L’utilisation de marqueurs fluorescents conventionnels (protéines fluorescentes ou sondes chimiques) sans aucune préparation particulière ; iv) Une imagerie super-résolue 3D (même à plusieurs microns de profondeur) et v) Le suivi de processus subcellulaires dynamiques à l’échelle nanométrique. Un exemple d’application de cette technique ? Citons une étude récente de l’équipe « Stress oxydant, protéines Fer-Soufre et cancer » du CNRS UPR2301 - Université Paris-Saclay - Institut de Chimie des Substances Naturelles (ICSN), Département de Chémobiologie. La protéine Cisd3 (ou MiNT) appartient à la famille des métalloprotéines NEET à centre [2Fe-2S]. Cisd3 est localisée dans la matrice mitochondriale. Elle serait impliquée dans le contrôle de la production d’espèces réactives de l’oxygène, le métabolisme du fer, la biogenèse des centres [Fe-S] et l’import de protéines dans la matrice mitochondriale. L’équipe de l’ICSN étudie l’expression et la localisation de Cisd3 dans des cellules soumises à différentes conditions de stress qui affectent la fonction mitochondriale et la disponibilité en fer (cf. figure – image de droite). Pour plus d'informations concernant ces prestations : romain.lebars@i2bc.paris-saclay.fr; sandrine.lecart@i2bc.paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, le 16 septembre 2019, la plateforme I2BC / Plateforme d’imagerie photonique (Imagerie-Gif) publiait son premier Scoop-it® / FOCUS PLATEFORME : Combiner la microscopie photonique spectrale automatisée et le machine learning au service d'un protocole d'imagerie quantitative 3D de grands volumes. Le lire à nouveau ? I2BC / Plateforme d’imagerie photonique (Imagerie-Gif). Les différents systèmes d'imagerie de la plateforme permettent des approches multi échelles allant du tissulaire (macroscopie), cellulaire (microscopie confocale) jusqu'au niveau subcellulaire (microscopie super résolue). La plateforme est hébergée au sein de l'Institut de Biologie Intégrative de la Cellule (I2BC), ce qui lui confère une forte expertise dans l'étude de différents modèles biologiques cellulaires : cellules animales, végétales, bactéries, levures, champignons. Afin de permettre aux utilisateurs d'exploiter au mieux les données obtenues sur les microscopes, ils sont également accompagnés en traitement et analyse des images. Des stations de calcul puissantes équipées de logiciels dédiés sont mises à leur disposition. La plateforme Imagerie-Gif peut accueillir les projets du secteur privé car elle est certifiée selon les normes ISO 9001 :2015/NFX 50-900 :2016. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 60 équipes de recherches et 15 plateformes technologiques, provenant de 8 unités de recherches (CGM, IBBMC, IGM, ISV, LEBS, VMS, SB2SM, SBiGeM). L’institut est réparti sur 3 sites de recherche (Campus d’Orsay Vallée de l’Université Paris-Saclay, Campus du CNRS de Gif sur Yvette et Campus du CEA / Centre de Saclay) au sein de 14 bâtiments jusqu’au rassemblement programmé en 2023 sur le campus du CNRS de Gif-sur-Yvette.

|
Scooped by
Life Sciences UPSaclay
February 9, 2023 10:40 AM
|
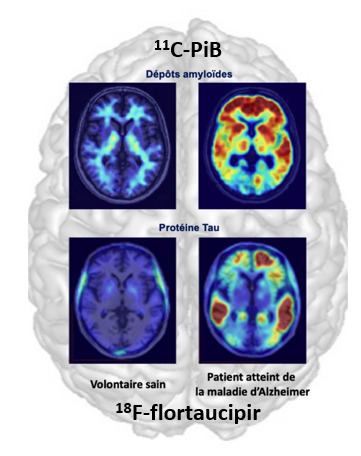
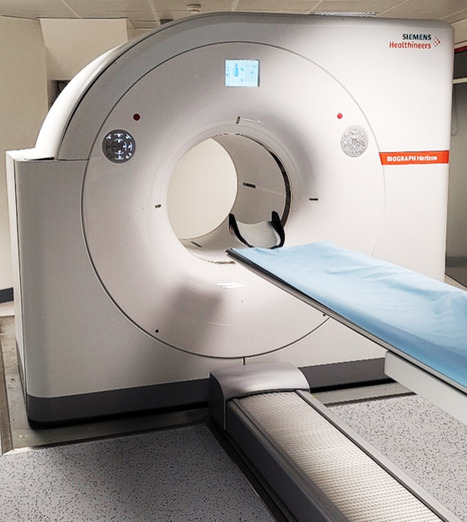
FOCUS PLATEFORME : L’Imagerie TEP « multi-traceur » pour une cartographie moléculaire du cerveau normal et pathologique ! La diversité des radiopharmaceutiques dédiés à l’imagerie moléculaire du cerveau permet aujourd’hui d’envisager le suivi de plusieurs biomarqueurs chez un même patient ! Novatrice, cette approche est aujourd’hui possible sur Paris-Saclay, où les chercheurs de BioMaps (UMR CEA/CNRS/Inserm/Université Paris-Saclay, Service Hospitalier Frédéric Joliot (SHFJ), Institut Joliot), en collaboration avec les cliniciens de l’hôpital Sainte-Anne (Unité de Neurologie de la Mémoire et du Langage, GHU Paris Psychiatrie & Neurosciences, Paris), utilisent cette approche « multi-traceur » pour apporter une lecture multiparamétrique voire mécanistique de l’évolution de la maladie d’Alzheimer. Le Service Hospitalier Frédéric Joliot (SHFJ) opère plusieurs plateaux techniques d’imagerie moléculaire par Tomographie par Emission de Positons (TEP), dont l’un d’entre eux, notamment équipée d’un imageur TEP à haute résolution (HRRT®, Siemens) (SHFJ / Imagerie préclinique et clinique, in vivo, TEP HR). Ces plateaux techniques sont adossés à une plateforme de radiochimie, unique sur Paris-Saclay et rare sur le territoire français, capable de produire une grande partie des radiopharmaceutiques pour l’imagerie TEP décrits dans la littérature. D’autre part, le SHFJ bénéficie de toutes autorisations pour la réalisation d’études cliniques et d’une infrastructure de radiopharmacie permettant l’administration à l’Homme de radiopharmaceutiques produits sur site, à la demande. Cette structure unique est largement sollicitée pour la réalisation d’études de neuroimagerie clinique, dans le cadre du diagnostic, du suivi ou de l’évaluation thérapeutique dans certaines maladies neurodégénératives ou psychiatriques. Cela inclut, en autres, la mesure de la charge amyloïde (11C-PiB ou 18F-florbetapir), la détection des fibrilles Tau (18F-flortaucipir) (en savoir plus ? Lagarde et al., J Neurol Neurosurg Psychiatry 2022), la quantification de l’activité neuro-immunitaire et neuro-inflammatoire (18F-DPA-714) (en savoir plus ? Hamelin et al., Brain 2018) ou, plus récemment, la mesure de la densité synaptique (11C-UCB-J). L’association de plusieurs marqueurs, parfois associée à celle de biomarqueurs périphériques plus accessible (via des prélèvements de sang ou de liquide céphalorachidien), permet de comparer directement leurs performances diagnostiques. Aussi, l’approche « multi-traceur » permet d’évaluer et de comparer la précocité des différents biomarqueurs en termes de détection précoce de la maladie, voire d’offrir un aperçu mécanistique de l’évolution de la maladie. Contacts : Nicolas Tournier (nicolas.tournier@cea.fr), Michel Bottlaender (michel.bottlaender@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI SHFJ / Imagerie préclinique et clinique, in vivo, TEP HR : La plateforme d'imagerie TEP Haute Résolution du SHFJ permet de réaliser des acquisitions par tomographie par émission de positons (TEP) cérébrales, précliniques ou cliniques, avec la meilleure résolution spatiale actuellement disponible pour ce type d'examen. L'imagerie TEP se fait au moyen de radiotraceurs extérieurs ou produits sur site. Des prestations d'analyse des images produites peuvent également être fournies. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.
|

|
Scooped by
Life Sciences UPSaclay
September 30, 2023 1:15 PM
|
La plateforme Penfield (ou plateforme d'imagerie du cerveau humain à très haute résolution temporelle par électroencéphalographie (EEG)) est l’une des 6 plateformes opérées par le CIAMS (laboratoire « Complexité, Innovation, Activités Motrices et Sportives »). Située à la Faculté des Sciences du Sport de l'Université Paris-Saclay (Campus d’Orsay), cette plateforme est régulièrement utilisée pour des projets de recherche s’inscrivant dans le domaine des neurosciences cognitives, comportementales et affectives. Elle vient particulièrement en support de deux champs d’investigations scientifiques actuellement développés au sein de l’équipe « Mouvement Humain, Adaptation et Performance Sportive » (MHAPS) du laboratoire. Le 1er axe de recherche a pour objectif d’identifier les déterminants d’une interaction naturelle avec un personnage virtuel (avatar) exprimant des émotions. Nos travaux s’intéressent au rôle de mouvements neutres (non communicatifs, telles que des oscillations posturales liées à l’équilibration humaine) pour améliorer le réalisme comportemental de personnages virtuels et rendre l’expression de leurs émotions plus crédible. De récents résultats de notre équipe révèlent que ces animations augmentent de façon significative la réaction affective face à un personnage exprimant de la douleur. Le 2ème axe de recherche vise à étudier les bases neuro-fonctionnelles du mouvement humain et de la plasticité sensorimotrice. Nos travaux s’intéressent notamment au rôle du sommeil dans le processus de consolidation de la mémoire, ainsi qu’aux effets d’une restriction sensorielle temporaire (i.e., immobilisation d’un membre) sur la plasticité cérébrale et la cohérence cortico-musculaire (couplage oscillatoire entre l’activité corticale et l’activité musculaire). Retour sur une publication récente (Treal et al., Heliyon 2023) mettant en valeur les équipements et expertises de la plateforme. Cette étude vise à mieux comprendre l’effet des mouvements neutres (i.e. oscillations posturales humaines) visant à rendre plus ‘vivant’ le personnage virtuel sur les corrélats cérébraux associés au traitement de son expression faciale de douleur. Nous avons présenté à 20 jeunes adultes (13 hommes et 7 femmes, âge moyen : 23 ans) des vidéos montrant un personnage virtuel avec une expression faciale de douleur. Dans une condition, le corps du personnage n’était pas animé (« condition figée ») et dans une autre condition, nous avons ajouté des oscillations posturales issues d’une capture de mouvement humain (« condition oscillations »). Dans cette étude les participants devaient juger l’intensité de la douleur exprimée par le personnage virtuel. Nous avons également enregistré la réponse cérébrale associée à la perception et au jugement de l’expression douloureuse au moyen de l’EEG. Nos résultats indiquent que les participants perçoivent l’expression faciale de douleur du personnage virtuel significativement plus intense en présence des oscillations biologiques du corps par rapport à la condition où il est « figé ». Surtout, l’amplitude de la réponse cérébrale associée à la réponse empathique suscitée par l’expression faciale de douleur du personnage est également significativement plus forte dans la condition « oscillations » par rapport à la condition « figée ». Cette étude démontre l’importance de prendre en compte ces éléments d’animation lors de l’utilisation de paradigmes expérimentaux s’appuyant sur la réalité virtuelle en neurosciences affectives et cognitives. Contacts : Aurore Meugnot (aurore.meugnot@universite-paris-saclay.fr); Arnaud Boutin (arnaud.boutin@universite-paris-saclay.fr); Jean Jeuvrey (jean.jeuvrey@universite-paris-saclay.fr). Plug In Labs Université Paris-Saclay : cliquer ICI CIAMS / Plateforme Penfield. Plateforme d'imagerie du cerveau humain à très haute résolution temporelle par électroencéphalographie (EEG). La plateforme EEG est utilisée à des fins de recherche fondamentale et appliquée en neurosciences cognitives et comportementales chez l'homme adulte sain. L'enregistrement du signal EEG peut également être couplé à des mesures électrophysiologiques (e.g., EMG, activité électrodermale) pour des enregistrements simultanés et complémentaires. A propos du CIAMS. Le CIAMS est un laboratoire multidisciplinaire (SDV et SHS) en sciences du mouvement et facteurs humains. Il contribue notamment au réseau Neurosciences, Mouvement, Handicap des unités de recherche en Sciences de la Vie de l'Université Paris-Saclay. Le CIAMS est un membre fondateur de la Fédération Demenÿ-Vaucanson (FéDeV), une Structure Fédérative de Recherche impliquant une quinzaine d’unités de recherche, dont la mission est de structurer la communauté scientifique de Paris-Saclay dans le domaine des sciences du mouvement.

|
Scooped by
Life Sciences UPSaclay
September 17, 2023 5:47 PM
|
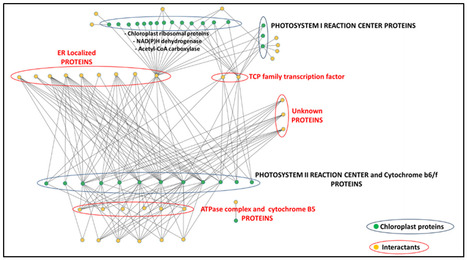
La plateforme SPOmics-Interactome (Institute of Plant Sciences Paris-Saclay (IPS2), Univ Evry, INRAE, CNRS, Université Paris-Saclay, Université Paris Cité) a été impliquée dans le projet international Arabidopsis Interactome Mapping - AIM afin d'établir la première carte du réseau d'interactions protéiques des plantes et aussi dans la construction de la plus grande base de données d'interacteurs d'effecteurs de pathogènes et dans plusieurs collaborations internationales en biologie végétale. Les protéines sont en effet les éléments de base de la transduction des signaux et des voies de biosynthèse, ainsi que les composants structurels qui soutiennent la vie. Au cours des dernières décennies, des preuves de plus en plus nombreuses soutiennent l'importance des complexes protéiques, plutôt que des protéines uniques, pour la régulation des processus biologiques. Ainsi, l'élucidation des interactions protéine-protéine qui se produisent dans les organismes vivants est un aspect central de la recherche biologique, et, avec les approches "omiques", elle représente une étape importante pour parvenir à une compréhension plus large des processus biologiques. Chez les eucaryotes, l'origine endosymbiotique des cellules et la séparation inhérente des compartiments intracellulaires nécessitent une communication efficace entre les organites et le noyau. Des preuves significatives suggèrent que le réticulum endoplasmique (RE) et les chloroplastes sont biochimiquement connectés, permettant, entre autres, l'échange d’eau oxygénée (H2O2). Ce composé peut également induire la formation de stromules plastidiques (protubérances tubulaires remplies de stroma qui s'étendent à partir du corps principal des plastides) pour transporter des signaux vers le noyau. Ces signaux entraînent l'activation de voies rétrogrades et, si la production de ROS est suffisamment importante, la mort cellulaire. La plateforme a récemment fait synthétiser l'ensemble de l'ORFeome du chloroplaste d'Arabidopsis afin de créer le premier réseau d'interaction protéine-protéine du chloroplaste par un système de double hybride sur levure (Y2H) à large échelle. Les résultats préliminaires sur 31 protéines codées par le génome du chloroplaste criblées contre la collection de 12 000 protéines d'Arabidopsis révèlent plusieurs interactions entre les protéines du chloroplaste et celles du RE impliquées dans la réponse aux stress biotiques et abiotiques et dans les voies de signalisation rétrograde. Ces résultats permettent de souligner l'existence de liens étroits entre les chloroplastes et le réticulum endoplasmique, et feront l’objet d’une présentation lors du congrès Plant Biology Europe (Marseille, 3-6 juillet 2023). Contact : Dario Monachello (dario.monachello@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI IPS2 / SPOmics-Interactome. SPOmics-Interactome est la plateforme d'étude des interactions protéine-protéine de l'IPS2. Les technologies qu'elle maitrise sont basée sur la seule méthode permettant la détection de ces interactions à haut débit et in vivo - le système double-hybride chez la levure (Y2H) - aujourd'hui optimisé et automatisé. A ce titre, grâce à l'utilisation de plaques 384 puits et d'un système robotisé d'une part, mais aussi d'un protocole entièrement en milieu liquide, la plateforme est capable de cribler un pool de 50 protéines hybrides (DB- X) contre la banque de protéines hybrides AD-AIM (environ 12000 protéines d'Arabidopsis) en deux mois. Le protocole généralement utilisé est le suivant : Les plasmides portant les ORFs codant pour les protéines d'intérêt fournis par nos collaborateurs dans le vecteur pDEST - DB sont utilisés pour transformer des levures, puis les protéines DB-X hybrides sont testées pour éliminer celles capables d'auto-activation. Les levures exprimant les protéines DB-X hybrides sont alors cultivées individuellement dans des milieux sélectifs, puis combinées en pools de 50 clones et criblées contre la banque AD-AIM. Après identification des protéines candidates, une analyse matricielle en Y2H est effectuée par « déconvolution » des 50 DB-proies et par tests individuels contre les candidats AD-appâts. Une étape finale de séquençage des protéines AD- et DB- permet la validation de l'identité des protéines en interaction. Chaque nouvel ORF criblé est intégré dans la banque AD- permettant une constante implémentation du réseau.

|
Scooped by
Life Sciences UPSaclay
August 24, 2023 4:47 AM
|
SAPS (Sciences Animales Paris-Saclay) est un collectif de recherche d’envergure internationale dédié à la biologie et à la santé animale, en lien étroit avec l’élevage. Créé en 2015, SAPS fédère sept unités de recherche, deux unités expérimentales et sept plateformes scientifiques, sous les tutelles conjointes de l’Institut national de recherche pour l’agriculture, l’alimentation et l’environnement (INRAE), d’AgroParisTech, du Centre National de la Recherche Scientifique (CNRS), de l’Ecole Nationale Vétérinaire d’Alfort (ENVA), de l’Agence nationale de sécurité sanitaire de l’alimentation, de l’environnement et du travail (ANSES) et du GIE LCH. Les membres de SAPS sont ancrés dans l’Université Paris-Saclay, notamment dans les Graduate Schools Biosphera, Life Science and Health, et également dans l’Université Paris-Est Créteil (UPEC) via ses partenariats avec l’ENVA et l’ANSES. En ce qui concerne plus particulièrement le périmètre Paris-Saclay, citons les UMRs BREED, GABI, MoSAR, VIM, ainsi que les UE IERP et SAAJ. SAPS (Sciences Animales Paris-Saclay) : quels sont les objectifs de ce réseau ? - Développer de nouvelles connaissances scientifiques en biologie et santé animale.
- Promouvoir les interactions entre les recherches sur les animaux d’élevage ou les animaux modèles et les recherches en santé animale et humaine.
- Renforcer les liens entre recherche en santé animale et santé humaine (One Health, zoonoses, effets de l’environnement).
- Développer les approches prédictives et « data science » dans le domaine de la biologie et de la santé animale.
- Promouvoir des systèmes d’élevage performants et respectueux de l’environnement.
- Contribuer au dialogue science-société et répondre aux attentes diversifiées des citoyens.
SAPS (Sciences Animales Paris-Saclay) : 4 grands domaines de connaissances fondamentales - i) la génétique, la génomique et l’épigénétique animale ;
- ii) la biologie de la reproduction, du développement et la différenciation cellulaire ;
iii) la santé animale et humaine : immunologie et infectiologie ; - iv) la biologie computationnelle et modélisation des systèmes biologiques complexes.
Contact : contact-saps@inrae.fr SAPS - Sciences Animales Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
July 8, 2023 11:50 AM
|
En IRM à très haut champ, les inhomogénéités spatiales du champ statique principal (B0) dans le cerveau s’accroissent à cause des disparités de susceptibilité magnétique entre les tissus biologiques, diamagnétiques, et l’air paramagnétique des cavités telles que les sinus ou les oreilles internes. Ces inhomogénéités sont la source d’artefacts dans les images acquises rapidement, notamment des distorsions et des pertes de signal (zones d’ombre très localisées). Si rien n’est fait, on peut donc perdre l’information ténue recherchée en IRM fonctionnelle notamment. Pour tenter d’y remédier, des techniques de compensation sont mises en place par les constructeurs, en équipant les IRM à haut champ de bobines dites de shim qui corrigent les harmoniques sphériques de la décomposition spatiale du champ jusqu’à l’ordre 2, voire 3 dans le cas d’Iseult. Cependant, à 11.7 Tesla, ces corrections restent insuffisantes pour réduire les artefacts des images rapides au niveau obtenu à plus bas champ. Un système dédié au cerveau humain a donc été développé en interne (NEUROSPIN, Institut Joliot, CEA, Centre de Saclay, Gif-sur-Yvette) et breveté. Il a donné naissance à une structure cylindrique dite SCOTCH, où vient s’insérer l’antenne radiofréquence dans laquelle la tête du sujet est placée. SCOTCH est un ensemble de multiples petites bobines contrôlées indépendamment, et complète le système de 8 grandes bobines fournies par le fabricant Siemens Healthineers pour corriger les inhomogénéités de B0 dans le cerveau humain. Pour concevoir ce fourreau de shim SCOTCH, les cartes de B0 de cerveaux de 100 sujets ont d’abord été acquises sur l'IRM à 3T de NeuroSpin. Puis, à partir de chacune d'elles ont été calculées les nappes de courants devant circuler idéalement sur une surface cylindrique pour contrer les disparités de champ. Une analyse en composantes principales a ensuite permis de retenir les lignes de courant les plus importantes, communes à tous les sujets. En séparant ces lignes, l’équipe d’instrumentation IRM de NeuroSpin a, pour la première fois, défini la taille, la forme et l'emplacement de multiples petites bobines optimisées pour « shimmer » le cerveau des sujets. Un premier prototype de fourreau SCOTCH a été construit, consistant en un cylindre de 3 cm d'épaisseur, d'environ 30 centimètres de diamètre et de longueur, et contenant 48 bobines disposées en 3 couches. Ce prototype a été testé sur une dizaine de sujets à 7T (NEUROSPIN / Imagerie in vivo chez l'homme, ex vivo et in vitro, IRM 7T) et a montré qu’il est équivalent à un système corrigeant les harmoniques sphériques jusqu’à l’ordre 7 avec une puissance illimitée, ce qui constitue un record mondial pour le cerveau. SCOTCH sera prochainement utilisé à 11.7T avec des courants accrus pour faire retomber le niveau d’artefacts au résiduel obtenu à 7T. Un moyen assurément de redonner le sourire aux chercheurs utilisant ces appareils. En savoir plus ? Pinho Meneses et al., NeuroImage 2022 Contact : Alexis Amadon (alexis.amadon@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI NEUROSPIN / Imagerie in vivo chez l'homme, ex vivo et in vitro, IRM 7T : Technologie : Imagerie par Résonance Magnétique (IRM) à ultra haut champ 7 Tesla. Recherche fondamentale et appliquée sur le cerveau chez l’homme Offre : Gestion de protocoles de recherche biomédicale d’imagerie, recrutement de volontaires sains, acquisition de données d'imagerie in vivo de cerveau chez l’homme ou in vitro de pièces anatomiques, développement et mises en place de séquences IRM (IRM anatomique, IRM de diffusion, spectroscopie RMN, IRM fonctionnelle) analyse et traitement de données d'imagerie. A propos de NeuroSpin. Neurospin est une infrastructure de recherche sur le cerveau exploitant des grands instruments d'imagerie. NeuroSpin offre à la communauté scientifique publique et privée la possibilité de faire progresser la connaissance du cerveau, et particulièrement du cerveau humain, en proposant un accès à des méthodologies de pointes en imagerie cérébrale et en neuro-informatique. NeuroSpin développe et met à la disposition de la communauté des instruments uniques, notamment en imagerie très haut champs et dans le domaine des big data. Cette offre s'inscrit dans le cadre des missions spécifiques de NeuroSpin qui sont : i) analyser les fonctions du cerveau humain, leur développement dans l'enfance, et l'impact de la culture et de l'éducation ; ii) identifier les marqueurs et les mécanismes de maladies neurologiques, psychiatriques et neurodéveloppementales ; iii) comparer le cerveau humain et celui d'autres espèces animales ; développer et tester des méthodes d'imagerie à toutes les échelles d'observation : par résonance magnétique (IRM), par électro- et magnéto-encéphalographie (EEG et MEG), et par électrophysiologie massivement parallèle ou l'imagerie photonique et v) développer des logiciels spécialisés dans le traitement et la modélisation des grands jeux de données en neuroimagerie. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
June 18, 2023 5:23 PM
|
Retour sur les ateliers PGD du GIS IBiSA. Organisés en 2022 et 2023, les derniers ateliers de formation du GIS ont porté sur le plan de gestion des données (PGD). Plus d’un tiers des plateformes IBiSA y ont participé, encourageant le GIS à envisager de nouvelles sessions sur ce thème et à les ouvrir aux CRB. En savoir plus. Les prochains ateliers de formation du GIS IBiSA. Les ateliers de formation du GIS s'invitent au menu sur le site ibisa.net. Dans l'onglet "Découvrir IBiSA", retrouvez désormais la page "Ateliers de formation" avec les prochaines sessions prévues en 2023 et toutes les informations utiles pour y participer. En savoir plus. Atelier 1 : Outils pour la gestion de projet et le pilotage de plateformes. Organisé le 13 octobre 2023 à Paris, cet atelier a pour objectif d’orienter le choix, la conception ou l'adaptation de logiciels et d'outils pour gérer facilement et efficacement les tâches et les projets sur une plateforme technologique. En savoir plus. Atelier 2 : Empreinte écologique des plateformes et bilan carbone. Prévu courant décembre 2023, cet atelier vise à aider les plateformes à mieux cerner les impacts de la recherche sur l'environnement, à calculer leur bilan carbone et à identifier des leviers pour réduire l'empreinte de leurs activités. En savoir plus. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! Vous souhaitez découvrir le potentiel complet de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes du domaine Sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle du CEA, du CNRS, d'INRAE, de l'Inria, de l'Inserm, de l’INCa, de la CPU et du Ministère de l’enseignement supérieur, de la recherche et de l’innovation (MESRI), il est l’unique instrument de financement commun à l’ensemble des établissements en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité en vue de la structuration et certification des plateformes. Plus d’infos sur ibisa.net. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire ! Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaire académique comme entreprise, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
June 4, 2023 5:28 PM
|
Le Service Hospitalier Frédéric Joliot (SHFJ, Orsay, Institut des Sciences du vivant Frédéric Joliot) opère plusieurs plateaux techniques d’imagerie moléculaire par Tomographie par Emission de Positons (TEP). L’un d’entre eux (SHFJ / Imagerie préclinique et clinique, in vivo, TEP/TDM) vient de remplacer son imageur clinique TEP couplé à la tomodensitométrie X (TEP-TDM) ! Le modèle en place aujourd’hui (Biograph Horizon®, Siemens Healthineers) a été installé en 2022 et est opérationnel depuis quelques mois ; il remplace une précédente version (Biograph TruePoint®, Siemens Healthineers), acquise en 2008. Ce nouveau TEP-TDM dispose de la technologie Temps-de-Vol qui permet d’améliorer la sensibilité et la spécificité des examens TEP. Cette sensibilité accrue permet notamment de réduire de 25% l’activité administrée aux patients ainsi que la durée de l’examen. En outre, le nouvel équipement permet une réduction de 30% de l’exposition du patient aux rayonnements X et une diminution des artefacts dans les images dus à la présence d’objets métalliques dans l’organisme, tels que des prothèses. Pour le docteur Duc-Loc Nguyen, médecin nucléaire au SHFJ, ce nouvel équipement permet au patient de bénéficier d’un examen moins irradiant et plus confortable, tout en gardant une qualité d’image optimale pour le diagnostic. Ce nouvel équipement est aujourd’hui utilisé majoritairement pour une activité de soins courants dans le cadre d’un bilan pour une maladie cancéreuse ou neurologique. Les médicaments radiopharmaceutiques (MRP) administrés aux patients sont marqués au fluor 18, un radionucléide émetteur de positons d’une demi-vie de 110 minutes. Parmi les MRPs les plus utilisés, citons le 2-[18F]fluoro-2-désoxy-D-glucose ([18F]FDG), un analogue du glucose fixé prioritairement par les cellules ayant un métabolisme des glucides élevé, notamment dans le cerveau et par les cellules cancéreuses. Viennent ensuite la 6[18F]fluoro-L-DOPA ([18F]FDOPA) pour l’évaluation de patients soit avec suspicion de syndrome parkinsonien, soit avec des tumeurs neuroendocrines, et la [18F]fluoro-Choline ([18F]Choline) pour les patients avec un carcinome hépatocellulaire (CHC), une hyperparathyroïdie ou un cancer de la prostate. Pour ces derniers patients, l’on peut également avoir recours au [18F]PSMA, un ligand de l’antigène membranaire spécifique de la prostate. Le nouveau TEP-TDM est également utilisé dans le cadre de la recherche impliquant la personne humaine, notamment dans la recherche sur la maladie d’Alzheimer. Dans ce cas, les MRPs administrés sont synthétisés dans les laboratoires du SHFJ, marqués au fluor-18 également mais aussi au carbone 11, un radionucléide émetteur de positions d’une demi-vie de 20 minutes. Contacts : Claude Comtat (claude.comtat@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI SHFJ / Imagerie préclinique et clinique, in vivo, TEP/TDM : La plateforme d'imagerie TEP/TDM permet de réaliser des acquisitions par tomographie par émission de positons (TEP) couplée à la tomodensitométrie (TDM), corps entier précliniques et cliniques. L'imagerie TEP se fait au moyen de radiotraceurs extérieurs ou produits sur site. Des prestations d'analyse des images produites peuvent également être fournies. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
May 20, 2023 6:43 AM
|
Les catalyseurs sont de véritables accélérateurs de réactions chimiques ! Non seulement, la catalyse est une astuce éprouvée qui n'utilise que de petites quantités du composé catalytiquement actif et qui, de plus, n'est pas consommé pendant la réaction, mais aujourd’hui la catalyse se révèle une vraie stratégie durable et un pilier incontournable de la chimie verte. La nature utilise des catalyseurs (enzymes) pour débloquer des réactions chimiques efficaces et sélectives. Les leçons tirées de la structure des sites actifs des enzymes sont aujourd’hui utilisées pour guider la conception des catalyseurs dans une approche bio-inspirée (en savoir plus ?). Cependant, la compréhension du fonctionnement de ces catalyseurs est nécessaire pour améliorer les performances dans le cadre d'une conception systématique guidée par le mécanisme réactionnel. L'exploration du mécanisme de réaction repose sur la capture d'intermédiaires importants au cours du cycle catalytique. C’est dans ce cadre que les travaux menés conjointement à l'I2BC (Institut de Biologie Intégrative de la Cellule, UMR 9198 (CEA, CNRS, UPSaclay), Gif-sur-Yvette; Institut Joliot, CEA, Saclay) et à l'ICMMO (Institut de Chimie Moléculaire et des Matériaux d'Orsay, UMR 8182 (CNRS, UPSaclay)) se développent et utilisent continuellement des plateformes spectroscopiques de pointe (voir ci-dessous) afin d'« attraper » ces espèces souvent considérées insaisissables. Dans deux articles publiés récemment, ce sont des intermédiaires critiques de catalyseurs à base de fer qui ont été « capturés ». Dans Pugliese et al., Angew.Chem. Int. Ed. 2022, les équipes ont mis en lumière un nouveau paradigme du cycle catalytique de la réduction du dioxyde de carbone (CO2) par des porphyrines de fer. En utilisant une porphyrine modifiée et décorée par des groupes fonctionnels d'urée agissants en tant qu'échafaudage de liaison hydrogène, ils ont pu améliorer de manière significative la production photocatalytique de monoxyde de carbone, un élément constitutif important des matériaux à base de carbone. En utilisant des techniques spectroscopiques complémentaires (UV-Vis, IR) telles que disponibles sur la plateforme I2BC / Plateforme de spectroscopies électroniques, ils ont pu montrer que pour ce catalyseur modifié, l'état d’oxydoréduction formel "Fe (I)" est déjà suffisant pour interagir avec le CO2 et effectuer sa conversion, alors que les variantes précédentes de ces catalyseurs devaient être activées à l'état formel "Fe (0)" pour lier le substrat CO2. Cette nouvelle découverte d'une fixation efficace du CO2 par les fonctions uréiques contribue à l'avancement de la conception de catalyseurs efficaces. Dans Pugliese et al., Chem. Sci. 2022, les équipes ont réussi à attraper l'espèce insaisissable Fe(III) hydroperoxo d'un catalyseur non hémique qui a été postulé comme l'espèce catalytiquement active pour les réactions de transfert d'atomes d'oxygène aux alcènes. Cette réaction produit des époxydes et des aldéhydes, qui sont des briques de base importants de la chimie. L'utilisation d'un photosensibilisateur, d'un médiateur d'électrons et d'un catalyseur à base de fer a permis de partager l'activation à deux électrons de l'O2 à la fois sur l'O2 et sur le catalyseur Fe(III) non hémique, évitant ainsi la tâche difficile de l'accumulation de charges sur le catalyseur tout en effectuant un processus d'activation à deux électrons. Il est important de noter que cette stratégie élégante élimine l'utilisation de produits chimiques sacrificiels pour réaliser la réaction de synthèse. En utilisant la spectroscopie RPE et le suivie par spectroscopique par flash laser (LFP), le groupe a pu piéger les espèces hydroperoxo de Fe(III) pendant la photocatalyse, excluant ainsi les voies potentielles d'auto-oxydation pour la réaction. Les connaissances acquises seront utiles pour optimiser l'efficacité globale du système en identifiant les étapes productives et délétères concurrentes dans le mécanisme. Cette fois, , en plus de la spectroscopie électronique, c’est aussi la plateforme I2BC / Plateforme de résonance paramagnétique électronique qui a été mise à contribution ! Contact : winfried.leibl@cea.fr ; philipp.gotico@cea.fr Plug In Labs Université Paris-Saclay : cliquer ICI I2BC / Plateforme de spectroscopies électroniques. La plateforme de Spectroscopies Électroniques (Institut de Biologie Intégrative de la Cellule, I2BC) offre ses services appliqués aux biomolécules à des équipes de recherche françaises et internationales. Nous sommes capables de suivre des changements spectroscopiques au niveau de la protéine dans des cellules intactes. La plateforme est équipée de plusieurs spectromètres d'absorption et de fluorescence (y compris un certain nombre de spectromètres PAM spécialisés) ainsi que des spectromètres à thermoluminescence. Pour certaines configurations, des cryostats sont disponibles pour les études à basse température, jusqu'à 77K ou 4K. La plateforme a développé (et continue à améliorer) un montage unique de spectroscopie optique résolue dans le temps (ca. 300 ps), surpassant les montages conventionnels (commerciaux) en sensibilité et en résolution temporelle. Ce type de méthodologie est particulièrement déterminant pour l'élucidation de processus irréversibles et/ou de processus qui se produisent dans des fenêtres temporelles allant de quelques centaines de picosecondes à des dizaines de nanosecondes, où les montages conventionnels performent mal ou ne peuvent pas être utilisées du tout. Cette plateforme fait partie du pôle des plateformes de Biophysiques de l'I2BC qui comprend les plateformes de RPE, FTIR, Résonance Raman, Spectroscopies Electroniques et Microscopie de fluorescence à super-résolution. I2BC / Plateforme de résonance paramagnétique électronique. La Résonance Paramagnétique Electronique (RPE) est la seule méthode de détection directe d'espèces paramagnétiques. Les applications de la spectroscopie RPE sont diverses comme le contrôle qualité ou la recherche moléculaire dans la biologie structurale, les matériaux et la physique quantique. La RPE permet la détection, l'identification et la quantification des métaux de transitions dans les états redox paramagnétiques (Mn, Fe, Cu, Co, Ni, etc), des espèces radicalaires et des états triplets. Elle est applicable à des mesures sur cellules entières et par des techniques de spin-trap elle permet la détection de ROS. A titre d'exemples, les expériences RPE ont produit des résultats très intéressants dans le domaine des structures des métalloprotéines, de leur fonctionnement par identification d'intermédiaires réactionnels comme par exemple pour les complexes photosynthétiques, les catalases/peroxydases, les protéines fer-soufre, les NO-synthases, les hémoprotéines, etc. Le parc des spectromètres RPE comprend 4 appareils en bande X dont un en mode pulsé. Les mesures se font en général à la température de l'hélium liquide mais un spectromètre est dédié aux mesures à températures ambiantes. Les mesures photo-cinétiques (t1/2 > à 1 ms) sont possibles grâce à la possibilité d'éclairer les échantillons par un laser Nd:YAG à 532 nm. La plateforme comprend également deux spectromètres fonctionnant dans la bande W permettant une très haute résolution spectrale et des mesures d'ENDOR pulsée, entre autres, permettant d'accéder à l'environnement des espèces paramagnétiques. Cette plateforme fait partie du pôle des plateformes de Biophysique de l'I2BC qui comprend les plateformes de RPE, FTIR, Résonance Raman, Spectroscopies Electroniques et Microscopie de fluorescence à super-résolution. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), constituée de 60 équipes de recherches et 15 plateformes technologiques, provenant de 8 unités de recherches (CGM, IBBMC, IGM, ISV, LEBS, VMS, SB2SM, SBiGeM). L’institut est réparti sur 3 sites de recherche (Campus d’Orsay Vallée de l’Université Paris-Saclay, Campus du CNRS de Gif sur Yvette et Campus du CEA / Centre de Saclay) au sein de 14 bâtiments jusqu’au rassemblement programmé en 2024 sur le campus du CNRS de Gif-sur-Yvette.
Vous êtes chercheurs, étudiants, ingénieurs ou techniciens ? Vous travaillez dans le public ou le privé ? Vous avez besoin d’un accompagnement scientifique, technique et pluridisciplinaire dans les processus de découverte d’un médicament ? Alors, venez découvrir les plateformes technologiques dédiées à l’innovation thérapeutique de la Faculté de Pharmacie de l’Université Paris-Saclay, l’Unité Mixte de Services IPSIT, mardi 21 janvier 2025de 13 H à 16 H. S’incrire Le format retenu est résolument court, 1 heure de visite, sur inscription, suivie d’échanges autour d’une dégustation, afin de donner une vision globale de l’unité mixte de services et de rencontrer son personnel. Résolument à l’interface de la chimie, de la biologie et de la clinique, l’IPSIT établit des ponts entre la cible pathologique et le médicament. Le processus de découverte d’un médicament requiert une approche multidisciplinaire associant des savoir-faire complémentaires, notamment dans les domaines de la chimie, de la galénique, de la biologie structurale, de la biologie moléculaire et cellulaire, sans oublier la génétique et la pharmacologie. L’IPSIT, par ses plateformes technologiques, apporte aux équipes une aide scientifique essentielle dans ce processus. L’IPSIT participe aussi à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Découvrez ainsi : - le pôle OMICS avec ses plateformes de transcriptomique et génomique (ACTAGen), protéomique (PROTEOMIC), spectrométrie de masse et lipidomique (SAMM)
- le pôle Imagerie Cellulaire avec ses plateformes de microscopie photonique super-résolue et d’analyse d’images (MIPSIT), de cytométrie en flux (CYM) et d’histopathologie (PHIC)
- le pôle Interaction avec la plateforme de criblage moléculaire (CIBLOT)
- Ainsi que des services transverses constituées par une animalerie ultra-moderne équipée de dispositifs d’exploration fonctionnelle (ANIMEX), une verrerie scientifique et technique (VERRE SCIEN-TECH) et un plateau de bio-informatique (BIOINFO).
->Contact : Valerie Domergue (valerie.domergue@universite-paris-saclay.fr), Directrice de l’IPSIT A propos de l’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes techniques, organisées en trois pôles technologiques (IMCELLF, OMICS et INTERACTIONS) et trois plateformes transverses. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
April 14, 2023 9:25 AM
|
La Région Ile-de-France contribue régulièrement, et au travers de différents dispositifs, à renforcer les compétences scientifiques et technologiques de son écosystème. A ce titre, elle réédite cette année son AAP SESAME - SESAME 2023 - AAP visant à donner aux laboratoires de recherche de son territoire les moyens nécessaires pour développer de nouveaux projets et pour mettre en œuvre des dispositifs expérimentaux originaux de recherche (comprenez « équipements »). Comme lors des précédentes éditions, ne sont éligibles à SESAME 2023, que des équipements dont le coût total est compris entre 0,2 M€ et 3 M€ HT ; D’autre part, pour rappel, l’aide prendra la forme d’une subvention dont le taux maximum d’intervention régionale pour l’acquisition de ces équipements s’élève à 66% du montant HT du coût total du projet. Ce taux d’intervention régionale pour les projets d’équipements en sciences humaines et sociales pourra néanmoins atteindre 100% du montant HT du coût total, à condition que 3 laboratoires franciliens distincts ou plus participent au projet. Les critères d'éligibilité et de sélection sont disponibles dans le texte de l'appel à projet SESAME 2023. D’autre part, le dossier de candidature est à compléter en ligne sur mesdemarches.iledefrance.fr, par le porteur du projet directement, après sélection par l’Université Paris-Saclay en ce qui nous concerne. Cette sélection se fera naturellement en prenant en compte, outre les critères de la Région mentionnés dans l'AAP, la plus-value collective pour l'Université Paris-Saclay. Attention également de bien prendre les critères d'exclusion mentionnées dans l’AAP, et si cela s'applique à votre projet, de tenir compte des remarques faites par le jury de la Région en cas de soumission les années précédentes. Ci-dessous, le rappel du calendrier, intégrant les échéances propres à l’Université Paris-Saclay : - le 31 mars 2023: Lancement de l’appel à projets SESAME 2023 ;
- le 5 mai 2023 au plus tard : les porteurs de projet transmettent aux écoles graduées/institut concernés leur projet dans l'état le plus avancé possible, avec copie à l'adresse vp.recherche@universite-paris-saclay.fr (et mention de l'institution qui porterait le projet) ;
- du 8 au 19 mai 2023: les GS/I évaluent les dossiers qui leur sont remontés et produisent un avis (classement en trois catégories A, B, C) et le cas échéant les priorisent au sein d'une catégorie s'il y a beaucoup de projets. Aussi, en fonction du nombre de projets rattachés au domaine Sciences de la Vie de l’Université Paris-Saclay et ses interfaces, une journée d’audition des porteurs pourrait être programmée au cours de cette période ;
- le 26 mai 2023: le CoDiReV sélectionne les huit projets que l'UPSaclay est autorisée à faire remonter ;
- entre le 27 mai et le 5 juin 2023 (14h00) s'effectuent les dépôts de projets auprès de la Région, par les institutions porteuses.
- le 5 juin 2023 (14h00): Date limite de dépôt des dossiers.
NB : La sélection des projets par la région Ile de France se fera courant septembre 2023, avec une décision finale communiquée après le vote des élus (novembre 2023).

|
Scooped by
Life Sciences UPSaclay
March 31, 2023 5:20 PM
|
Les conjugués protéines-médicaments sont considérés de nos jours comme des constituants essentiels de l’arsenal thérapeutique. En particulier, les conjugués anticorps-médicaments sont régulièrement proposés pour le traitement de certains cancers. On parle alors d’ « ADC » (ou Antibody Drug Conjugate), l’anticorps assurant un ciblage précis des cellules tumorales alors que le médicament lié permettant de les détruire. Comme tout médicament (ou candidat médicament), et afin d’améliorer son activité mais aussi obtenir son autorisation de mise sur le marché, la collecte d’informations précises sur sa biodistribution et ses concentrations plasmatiques et tissulaires après leur ingestion sont requises. L’étude du devenir in vivo de telles architectures moléculaires est cependant complexifiée par leur nature « duale » et suivre simultanément les deux constituants de ces conjugués (macromolécule d’une part, petite molécule d’autre part) peut s’avérer difficile, et faire appel à des approches analytiques multiples, très lourdes et couteuses à mettre en œuvre. Dans ce contexte, l’équipe du Dr. Laurent Devel (Institut Joliot, département médicaments et technologies pour la santé (DMTS), SIMOS, CEA Paris-Saclay) en collaboration avec les chercheurs de la plateforme de marquage isotopique du même département (Institut Joliot, DMTS, SCBM, CEA Paris-Saclay)), a mené une étude portant sur le développement d’une approche bio analytique originale. Cette étude, publiée récemment (Cahuzac et al., J. Med. Chem. 2022), met en jeu le marquage radioactif des deux constituants d’un conjugué anticorps-médicament (ADC) modèle, par deux isotopes radioactifs, émetteurs beta-moins tous les deux, mais se distinguant par des énergies d’émission différentes (carbone-14 et tritium). Ce double marquage permet à la fois la quantification absolue de la radioactivité présente (comptage par scintillation liquide) et son imagerie (radio-imagerie beta). L’incorporation de ces deux émetteurs beta a mis en jeu toute l’expertise de la plateforme : la partie « anticorps » a été marquée au carbone 14 via une réaction de chimie « click » et la partie « médicament » a été marquée au tritium par l’utilisation de nanocatalyseurs de Ruthénium. Cette stratégie de double marquage a permis de suivre les deux constituants de cet ADC, simultanément pendant la phase de circulation dans le plasma, mais aussi de quantifier la fraction accumulée dans différents organes et la tumeur chez le rongeur. En savoir plus ? Relire aussi un précédent FOCUS PLATEFORME rédigé sur le sujet (La radio-imagerie au service de l’évaluation pharmacologique et toxicologique de composés d’intérêt chez le petit animal, 23 mai 2022). A l’avenir, cette approche novatrice pourrait se généraliser à tout ADC ou conjugué protéine-médicament, et permettre une accélération de leur développement et mise à disposition du patient. Contacts: Gregory Pieters (gregory.pieters@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme de marquage isotopique du CEA / Paris-Saclay (Institut Joliot, Département Médicaments et Technologies pour la Santé, Service de Chimie Bioorganique et de Marquage, Gif-sur-Yvette) est unique sur le territoire Paris-Saclay. Forte de son expertise dans la préparation (synthèse, contrôle de qualité) et formulation de molécules marqués, elle assure régulièrement des prestations et collaborations, académiques comme industrielles, dans le domaine du (radio)marquage moléculaire. Elle offre également à la demande, son expertise et environnement unique de travail (laboratoires « chauds », équipements dédiés) pour l’analyse et la caractérisation d’échantillons radioactifs : mesure de puretés chimique et radiochimique par HPLC, détermination d'enrichissements isotopiques et d'activités spécifiques par SM, analyse et détermination structurale par RMN liquide comme solide, mesure d’activités radioactives par comptage à scintillation. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
March 19, 2023 11:52 AM
|
Résultats de l'appel d'offres Plateformes IBiSA 2022. Au bilan de cette dernière édition, 71 candidatures reçues, 8 labels, 8 labels à 2 ans et 3 174 000 € distribuées à 53 plateformes en sciences du vivant. IBiSA a mis l'accent sur l'exposome, thématique récemment introduite dans les infrastructures nationales. Découvrez les candidats retenus. Dynamiser la recherche sur l'exposome chimique humain. En 2022, IBiSA a soutenu plusieurs structures travaillant sur l'exposome. Parmi elles, AnimEx, une plateforme de France Exposome, la nouvelle infrastructure nationale en biologie et santé dédiée à la recherche dans ce domaine. Zoom sur l’exposome et les intérêts d'une telle coopération. En savoir plus. Les appels d'offres IBiSA 2023 sont ouverts. Vous souhaitez obtenir la labellisation IBiSA ou être soutenu par un financement du GIS ? Les appels d'offres Plateformes et CRB 2023 sont ouverts depuis le 23 janvier. Les candidatures se font en ligne sur le site du GIS IBiSA. En savoir plus. Les ateliers IBiSA se poursuivent en 2023. Face au succès grandissant des ateliers organisés sur le plan de gestion de données (PGD), le réseau de compétences du GIS IBiSA envisage de nouvelles formations pour aider les plateformes à calculer leur coût environnemental. En savoir plus. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! Vous souhaitez découvrir le potentiel complet de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes du domaine Sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. _____________________ A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle du CEA, du CNRS, d'INRAE, de l'Inria, de l'Inserm, de l’INCa, de la CPU et du Ministère de l’enseignement supérieur, de la recherche et de l’innovation (MESRI), il est l’unique instrument de financement commun à l’ensemble des établissements en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité en vue de la structuration et certification des plateformes. Plus d’infos sur ibisa.net. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire ! Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaire académique comme entreprise, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
March 2, 2023 4:45 PM
|
Fin 2022, l’unité expérimentale d’infectiologie expérimentale rongeurs et poissons (IERP, UE 0907) a acquis un nouvel équipement, cofinancé par le DIM1health et INRAE pour proposer dès cette année un nouveau service de criblage et d’analyse à haut débit (HTS), en complément des services déjà bien établis (développement de modèles Poisson Zèbre, infectiologie, phénotypage par imagerie in vivo (3D, 3D+T) et transparisation). Spécialisée dans le développement de modèles poisons zèbre pour l’étude des maladies humaines et animales, l’unité IERP est ouverte à la communauté scientifique notamment pour l’accès à des techniques de phénotypage innovantes (transparisation/imagerie 3D/3D+t de tissus épais) au moyen d’équipements de pointe distribués au sein du dispositif. Intéressés par le suivi in vivo de pathogènes traçables optiquement, de nanoparticules fluorescentes ou par la caractérisation des réponses de l’hôte (lignées transgéniques fluorescentes/ biosenseurs), l’unité IERP propose des approches d’imagerie in vivo non invasives par microscopie de fluorescence (stéréomicroscopes, confocal, 2-photons). Toutefois, le temps de préparation des larves de poisson zèbre pour l'imagerie (anesthésie/montage), la phototoxicité et la gestion d’importants jeux de données (traitement, analyses d’images, analyses statistiques, et stockage) limitent les possibilités d’approches à haut débit. Pour le développement de ces activités HTS, l’IERP a donc plutôt opté pour un COPAS (Union Biometrica), instrument dédié au tri et à l'analyse d’objets biologiques complexes par cytométrie en flux. Cet appareil déjà utilisé par les entreprises privées, était jusque-là non accessible à la communauté scientifique de l'Université Paris Saclay. Le COPAS est dédié à l'analyse et au tri de larves de poisson zèbre (Zf) de 20 à 700 µm, âgées de 3 à 5 jours après la fécondation. Les larves sont facilement collectées en tubes de 50 mL, puis introduites dans la gaine liquide d’entrainement en préservant leur intégrité et leur viabilité à tout moment pendant le chargement et l'analyse. Après collecte des données (densité optique, fluorescence 488/561), les larves peuvent être distribuées en plaques multi-puits pour le suivi individuel à différents temps d’analyse ou utilisées pour la conduite d’autres types d’analyses (transcriptomique/protéomique/métabolomique). Les données sont exploitées pour quantifier les nombres d’évènements, l’intensité des signaux de fluorescence mais aussi pour caractériser le profil d’expression de ces signaux dans le corps de la larve via l’outil d’analyse Profiler©. L'appareil est idéalement placé au sein du dispositif piscicole pour limiter le stress, la manipulation et le transport des larves en expérimentation. Contact : Christelle Langevin (christelle.langevin@inrae.fr) ou dimitri Rigaudeau (dimitri.rigaudeau@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI INRAE / Infectiologie expérimentale des rongeurs et poissons (IERP). L'infrastructure scientifique collective IERP a pour principales missions la production d'animaux à statut sanitaire et génétique défini, la réalisation in vivo d'expérimentation en infectiologie et le phénotypage des animaux infectés par imagerie. Dans le cadre de projets collaboratifs ou de prestations de service, l'infrastructure mène des projets en infectiologie, immunologie, vaccinologie, génétique, physiologie et microbiologie. Distribuée dans plusieurs bâtiments du campus, notre infrastructure réalise des projets multi-espèces au sein d'une animalerie rongeurs pour les modèles souris et rat et d'une pisciculture expérimentale hébergeant les espèces aquacoles (truite et carpe) et poisson-zèbre. Chaque installation dispose de laboratoires de confinements (BSL2/3) équipés de technologies avancées (IVIS, bi-photon) pour le suivi par imagerie des animaux infectés.
|



 Your new post is loading...
Your new post is loading...