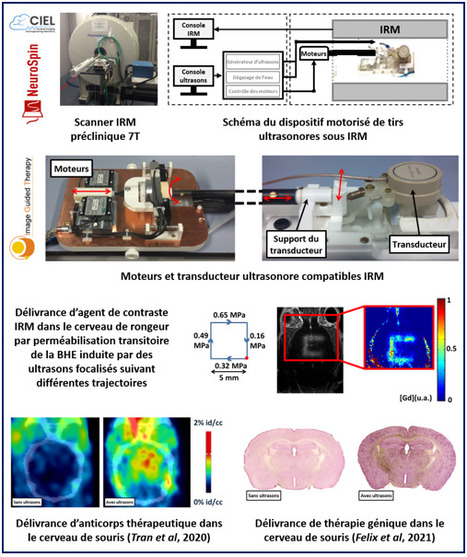
Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
June 21, 2025 5:48 AM
|
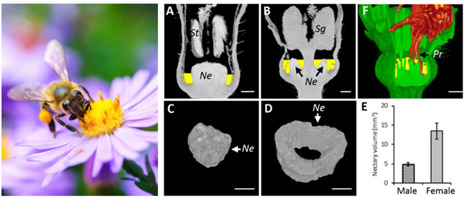
La plateforme « épigénomique et recherche translationnelle » de l’IPS2 (EPITRANS) est une infrastructure scientifique collective (ISC) de l’INRAE, labellisée IBISA et certifiée Iso9001. Elle a pour mission de développer et de mettre à disposition de la communauté scientifique des outils de génétique haut-débit, directe et inverse, chez les espèces cultivées. Elle propose d’identifier les gènes et les régulations épigénomiques majeurs contrôlant les caractères d’intérêts agronomiques puis à créer des allèles qui permettent d’améliorer la performance des plantes. Elle se positionne comme pierre angulaire entre la recherche fondamentale et la recherche appliquée, sur le territoire Paris-Saclay, mais aussi au niveau national et international. EPITRANS est intégrée à l’équipe de recherche FLOWER and CARPEL Developpement (FLOCAD, Dr. A. Bendahmane, IPS2) avec laquelle elle développe de fortes interactions scientifiques. Une illustration récente de cette association plateforme-équipe de recherche est le projet NectarGland, lauréat ERC, portant sur l’amélioration de l’attrait des fleurs pour leurs insectes pollinisateurs. Les insectes pollinisateurs sont essentiels à la vie sur Terre. Alors que la population humaine ne cesse de croître, les populations de pollinisateurs diminuent, menaçant ainsi la sécurité alimentaire. En agriculture, les principaux insectes pollinisateurs sont, de loin, les abeilles. Le manque de connaissances sur la manière dont les plantes cultivées attirent et récompensent les abeilles a freiné la sélection de variétés améliorant les relations mutuellement bénéfiques entre les cultures et leurs pollinisateurs. NectarGland est un projet pluridisciplinaire visant à améliorer la compréhension du développement des nectaires et de la sécrétion nectarifère en relation avec le butinage des insectes pollinisateurs, ces caractères ayant un impact direct sur le rendement en fruits, sa stabilité et les ressources alimentaires des abeilles. L’équipe de recherche FLOCAD va étudier les interactions plante-insecte par l’analyse génétique moléculaire et le phénotypage en s’appuyant sur le système modèle du melon, culture strictement entomophile, qui présente toutes les morphologies sexuelles florales (fleurs mâles, femelles, hermaphrodites) nécessaires. Le but est d’identifier les allèles contrôlant les caractères liés au nectar (développement des glandes et production) et à l’attraction des abeilles ainsi que leurs régulateurs clés potentiels. Une fois les gènes et leurs régulateurs identifiés ils seront validés génétiquement. Ce projet va utiliser différents outils proposés par la plateforme EPITRANS comme le clonage positionnel, le TILLING, le CRISPR, l’ATAC-seq et le DAP-seq. À travers ce travail, l’association de la plateforme EPITRANS & de l’équipe de recherche FLOCAD vise à développer une boîte à outils pour les sélectionneurs permettant d’adapter la morphologie et la composition chimique des fleurs afin d’améliorer l’activité de butinage des abeilles en les récompensant pour les services de pollinisations. En savoir plus sur NectarGland ? Une publication préliminaire ? Slavković et al., Trends Plant Sciences 2021 En savoir plus sur les activités d’EPITRANS ? Tous les ans, les 23 et 24 juin cette année, EPITRANS organise avec d’autres plateformes du plateau de Saclay les Journées Omiques Végétales. Lien pour le programme et l’inscription : https://ips2.u-psud.fr/fr/plateformes/spomics/colloque-spomics-2020.html Enfin, en janvier 2020, mars 2023 et juin 2024, EPITRANS publiait ses premiers FOCUS PLATEFORME … Redécouvrez- les ! -> Contact: fabien.marcel@inrae.fr / marion.dalmais@inrae.fr Plug In Labs Université Paris-Saclay : cliquer ICI IPS2 / EPITRANS. La plateforme EPIgénomique et recherche TRANSlationnelle (EPITRANS), Infrastructure Scientifique Collective (ISC) de l'INRAE, a pour missions de valoriser la recherche fondamentale en transformant les idées en produits et de faire le lien entre le chercheur et le secteur économique. Elle s'intéresse aux allèles et épi-allèles qui améliorent la performance des plantes dans un environnement de plus en plus contraint et plus particulièrement chez les espèces d'intérêt agronomique. Son activité consiste à développer et à mettre à disposition de la communauté scientifique des outils de génétique haut-débit, forward et reverse, chez les végétaux, notamment lorsque les études traditionnelles de génétiques ne peuvent s'appliquer (transformation génétique). Cinq outils sont ainsi proposés : le clonage positionnel par NGS, le TILLING (« targeted Induced Local Lesion IN Genome ») et l'ECO-TILLING, l'Epigénomique et le CRISPR. Le premier permet de cloner des gènes d'intérêts agronomiques pour ensuite étudier leur(s) fonction(s) par TILLING, par recherche de modifications épigénomiques, ou encore par édition du génome via le système CRISPR (selon les espèces). Le TILLING s'appuie sur la production de larges collections (<5000 lignées) de plantes mutées ou de collections de germoplasmes (ECOTILLING) combinée à une identification rapide et systématique des mutations dans les séquences cibles. Ces allèles, non génétiquement modifiés, sont proposés aux sélectionneurs pour améliorer leurs lignées élites en leur offrant une alternative aux collections limitées de germoplasmes. C'est pourquoi, la plateforme a des liens étroits et durables avec le secteur industriel dans différents domaines d'applications (Limagrain, Gautier Semence, Rijk Zwaan, Symrise). EPITRANS est leader en Europe dans le domaine de la recherche translationnelle de par la richesse de ses collections de mutants (260 000 lignées) et par la diversité des espèces cultivées disponibles (13 au total). La plateforme est labellisée par le GIS-IBISA (Infrastructures en Biologie Santé et Agronomie) et est certifiée Iso9001. A propos de l’IPS2. L’Institute of Plant Sciences Paris-Saclay ou IPS2 a pour mission de comprendre les mécanismes génétiques et moléculaires qui contrôlent la croissance de la plante et leurs régulations par les signaux endogènes et exogènes d’origine biotique (symbiotiques et pathogènes) et abiotique, notamment en relation avec le changement climatique. L’analyse de ces mécanismes est effectuée de manière intégrée à l’échelle de la cellule, de l’organe jusqu’à la plante entière. L’IPS2 applique une approche multidisciplinaire en combinant la génomique/epigénomique, la biologie cellulaire, la bio-informatique, la biochimie, la génétique, et la physiologie, développe des outils de modélisation indispensables pour une biologie prédictive, et facilite la recherche translationnelle des espèces modèles aux espèces cultivées.

|
Scooped by
Life Sciences UPSaclay
June 1, 2025 5:30 PM
|
Créé en 2023 autour de 8 plateformes et fort de son dynamisme, le réseau CyPSay (pour Cytométrie Paris-Saclay) s’enrichit en accueillant 5 plateformes supplémentaires ! Ainsi, CyPSay fédère à présent 13 plateformes : Il y a 2 ans, ce réseau a réuni CYM (ex PLAIMMO, IPSIT / Plateforme de Cytométrie H. Moissan), SpICy (ICP / SpectroImageries et Cytométrie), FlowCyTech (IDMIT, Institut JACOB) et I2BC / Plateforme de cytométrie (IMAGERIE-GIF) au réseau des plateformes du biocluster GENOPOLE, nommé OCCIGEN. Ce réseau génopolitain regroupe des équipements complémentaires présents sur ImCy (Imagerie-Cytométrie, Généthon), sur la plateforme de bioproduction (cellulaire, automatisée) de l’I-Stem, au LGRK (Laboratoire de Génomique et Radiobiologie de la Kératinopoïèse, Institut F. Jacob) et sur la plateforme de tri et clonage de levures opérée par Hybrigenics Services. S’y ajoutent aujourd’hui les plateformes CYMAGES (Plateforme de Cytométrie et Imagerie) de l’UVSQ, CurieCoreTech CYPIC de l’institut Curie (site Orsay), CyACT (Plateforme de Cytométrie Analyse Cellulaire et Tri /iRCM, Institut JACOB / CEA), @BRIDGe et le plateau de phénotypage de l’IERP à l’INRAE de Jouy-en-Josas qui étoffent l’offre de cytométrie du réseau. CyPSay recense maintenant toute une panoplie d’équipements, tous complémentaires, dont : Fortessa et Fortessa X-20 (BD Biosciences) ; cytoFLEX S et cytoFLEX LX (Beckman Coulter) ; Partec (Sysmex) ; NovoCyte 3000 (Agilent), MACS Quant 10 et MACS Quant X (Miltenyi) ; SP6800 (Sony) ; ZE5 (Biorad) , Aurora (Cytek), HELIOS (Standard Biotools), très souvent situés à proximité de trieurs : SH800 (Sony), ARIA IIu, ARIA III SORP et ARIA Fusion (BD Biosciences) ; MoFlo AstriosEQ, cytoFLEX SRT (Beckman Coulter), Influx (BD Biosciences), ainsi qu’un COPAS FP1000 (Union-Biometrica), cytomètre original permettant l’analyse et le tri d’objets biologiques complexes (larves de poisson zèbre, organoïdes) et un cytomètre imageur en flux ISX (Cytek) . A travers ce FOCUS PLATEFORME, nous souhaitons rappeler les trois principaux objectifs de ce réseau de proximité. A l’échelle des plateformes, il s’agit de partager les expertises respectives des partenaires lors de réunions régulières, de discuter des évolutions des technologies pour acquérir et analyser des données de cytométrie, d’identifier les besoins en investissements et de se fédérer pour des demandes de financement d’instruments et/ou de logiciels spécifiques. Pour chaque utilisateur, le réseau permet de proposer un accompagnement optimal dans tous types de projets en cytométrie. Enfin, à l’échelle de la communauté Paris-Saclay, le réseau vise à promouvoir la découverte de la cytométrie et de ses variantes à travers différentes journées de formation ou séminaires ouverts à tous. Sur ce dernier axe, la deuxième journée de formation à la cytométrie en flux s’est déroulée le mardi 19 novembre 2024 à Henri Moissan et a regroupé 35 personnes autour de présentations, de tables rondes et d’un déjeuner financé par la GS Heads (Action workshop) et la société Standard Bio Tools. Cette journée a été l’occasion de présenter les différentes cytométries et quelques applications spécifiques proposées sur les plateformes du réseau (le FRET, la mesure du pH intracellulaire, la prolifération et étude du cycle cellulaire, l’analyse de la phosphorylation des protéines). Ce fut une très belle réussite que nous souhaitons renouveler très rapidement ! Le réseau CyPSay travaille maintenant à la mise en place de webinaires techniques au cours de l’année et à l’organisation d’une nouvelle journée le 5 novembre 2025 à laquelle vous serez prochainement invités. A cette occasion, nous espérons tous vous y retrouver très nombreux ! Pour tout renseignement complémentaire, n'hésitez pas à contacter la plateforme du réseau CyPSay la plus proche. ->Contact : Marie-Laure Aknin (marie-laure.aknin@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI CYM, à l’origine du réseau CyPsay, a déjà publié plusieurs FOCUS PLATEFORME, n’hésitez-pas à les relire … IPSIT / Plateforme de Cytométrie H. Moissan (CYM). La plateforme de cytométrie en flux (CYM) de l'Unité Mixte de Service, Ingénierie et Plateformes au Service de l'Innovation Thérapeutique (UMS-IPSIT), située au rez-de-chaussée du bâtiment Recherche Henri Moissan (HM1) offre régulièrement ses services aux équipes de recherche académiques du territoire Paris-Saclay ainsi qu'aux industriels. Le personnel de la plateforme est à votre disposition pour vous aider à la réalisation de projets de recherche fondamentale, préclinique sur des modèles expérimentaux ainsi que pour des protocoles de recherche clinique. Son personnel est aussi à votre service pour la mise au point de nouvelles techniques utilisant la cytométrie en flux. Les équipements de cytométrie en flux de la plateforme permettent le phénotypage des cellules par la détection de molécules membranaires et intracellulaires (biomarqueurs) mais aussi des études fonctionnelles telles que la détection de phosphorylation des protéines, la prolifération cellulaire, la quantification de cytokines ou chimiokines excrétées ou la détection d'ARN. Enfin, des tris cellulaires à haut débit sont aussi proposés par la plateforme. Et en parallèle, elle étend maintenant son offre de service en mettant à votre disposition un nouvel appareil de mesure de haute technologie, le QuickPlex® SQ 120 (Meso Scale Discovery, MSD) permettant l’analyse en multiplex de biomarqueurs par electrochemiluminescence. Nos activités qui peuvent être en relations avec celle d'autres plateformes, permettent l'identification de nouveaux biomarqueurs pouvant être des cibles thérapeutiques. A propos d’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 11 plateformes techniques, organisées en trois pôles technologiques (IMCELLF, OMICS et INTERACTIONS) et trois plateformes transverses. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
May 18, 2025 4:21 PM
|
Aujourd’hui, focus sur un nouvel équipement, et une expertise unique sur le territoire Paris-Saclay : Le tri de micro-organismes pathogènes par cytométrie en flux ! Pour rappel, cette technique permet de mesurer, évènement par évènement, des caractéristiques morphologiques (taille, complexité/granularité) et de fluorescence sur une population de cellules ou d’organites en suspension. Le tri par cytométrie en flux rend également possible la séparation physique et donc la purification de ces évènements suivant un ou plusieurs critères définis (morphologie, fluorescence) et à partir de populations hétérogènes. Nouvel équipement et expertise unique sur Paris-Saclay ? L’écoute et l’anticipation des besoins utilisateurs (dialogue, mais aussi via enquêtes de satisfaction et de besoins) sur la plateforme de cytométrie de l’I2BC (I2BC / Plateforme de cytométrie (I2BC - plateformes IMAGERIE-GIF, Gif-sur-Yvette, CNRS/Institut Joliot CEA/UPSaclay) ont souligné une nécessité de réaliser des approches en cytométrie en flux dans des espaces L2. L'accès à l'expertise et aux équipements pour réaliser de telles expériences est restreint dans le périmètre de l'Université Paris-Saclay, et un réel besoin de prestations en environnement L2 a été identifié. C’est pour répondre à ce besoin d’un accès « facile » à un cytomètre de tri « user-friendly » permettant à l’utilisateur d’être autonome et la nécessité de travailler en L2 pour les projets de tri de micro-organismes pathogènes que la plateforme de cytométrie s’est équipée d’un CytoFLEX SRT (Beckman) en espace L2 dans l’I2BC. Ce nouveau trieur « de paillasse », financé en partie par l’ERM Paris-Saclay, est installé sous PSM de type II dans un environnement L2 de l’I2BC, et dédié aux micro-organismes pathogènes. Sa configuration a été discutée pour assurer sa complémentarité et sa compatibilité avec l’analyseur existant. L’appareil est équipé de 2 lasers (488 nm et 561 nm), de 7 détecteurs de fluorescence (525/40 nm, 585/42 nm, 610/20 nm, 675/30 nm, 690/50 nm, 710/50 nm et 780/60 nm) et 2 détecteurs de taille/granularité. Cette configuration permettra de lire un grand nombre de fluorochromes endogènes (GFP, mCherry, dTomato…) et exogènes (nombreux AlexaFluor, nombreux marqueurs d’acides nucléiques, …) pour mieux identifier les cellules d’intérêt, même si elles sont rares. Une fois les cellules d’intérêt identifiées, elles pourront être triées dans des tubes (le trieur bénéficie de 4 voies de tri en sortie) ou sur des plaques de 6 à 96 puits. Les bactéries triées pourront être remises en culture ou utilisées pour des expériences de biologie moléculaire (RNAseQ, ChIPseQ, Hi-C, par exemple). Le CytoFLEX SRT permet de préparer une configuration de tri facile et automatisée, avec une mesure de contrôle qualité assurée par des billes de fluorescence. Tous ces paramètres en font un trieur facile à utiliser en autonomie. L’équipement sera donc en accès autonome après formation par les ingénieurs de la plateforme (Karine Madiona et Mickael Bourge). N’hésitez pas à contacter la plateforme pour mettre au point vos expériences de tri qui pourront débuter au cours du 1er trimestre 2025. -> Contact : Mickael Bourge et Karine Madiona (plt-cyto@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme communique régulièrement via des FOCUS PLATEFORME. Envie de les relire ? I2BC / Plateforme de cytométrie (I2BC - plateformes IMAGERIE-GIF). La plateforme réalise environ 300 prestations par an pour divers groupes de recherche appartenant à différents organismes de tutelles ou de sociétés privées. Les publications du service traduisent les nombreuses collaborations développées avec les différents instituts de la communauté Paris-Saclay ainsi qu’avec d’autres partenaires tels que l’INRAe, l’INSERM, l’IRD, le CIRAD, des universités françaises et étrangères. Son expérience polyvalente et son expertise en sondes fluorescentes permettent d’adapter la cytométrie à des projets très divers issus de laboratoires publics et privés. Son partenariat avec SPS (Labex Saclay Plant Sciences) contribue à une activité importante dans le domaine de la biologie végétale. La plateforme de cytométrie en flux propose la mesure de fluorescence d'un ou plusieurs (> 10) fluorochromes simultanément, cellule par cellule. Cette technologie permet d'étudier: i) le dosage de la quantité d’ADN nucléaire en vue de l’étude de cycles cellulaires et d’endoréplication, ii) le dosage d’ADN à des fins de recherche en écologie et systématique, et d’amélioration des variétés (analyse de ploïdies), iii) le suivi de l’activité génique par l’expression d’un ou plusieurs gènes rapporteurs (tels que celui de la "Green Fluorescent Protein"-GFP et autres protéines fluorescentes), iv) des mesures d’activités métaboliques de la cellule (biosenseurs): dosage de calcium, pH, potentiel membranaire, poussées oxydatives, glutathion..., v) des analyses immunologiques, vi) le tri de cellules animales, de levures, de bactéries, de protoplastes et d’organites cellulaires. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.

|
Scooped by
Life Sciences UPSaclay
April 25, 2025 5:12 PM
|
L'Institut de Biologie Intégrative de la Cellule (I2BC, CNRS/CEA/UPSaclay, Gif-sur-Yvette) est une unité mixte de recherche soutenue par l'Université Paris-Saclay, le CNRS et le CEA, accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, répartis en 6 pôles. Redécouvrez toutes ces plateformes via un FOCUS PLATEFORME dédié ! Une des activités de cet institut généraliste est l’étude de la photocatalyse naturelle et artificielle. Ces études s’intéressent notamment à l’interaction lumière-matière et à la transformation de l’énergie lumineuse en énergie chimique, à la fois du point de vue des mécanismes fondamentaux mais aussi pour concevoir des photocatalyseurs capables d’oxyder l’eau ou de réduire le CO2. Pour se faire, l’I2BC peut s’appuyer sur son plateau de spectrométrie Raman, incluse dans la plateforme de Biophysique de Saclay, plateforme labellisée IBiSA (Infrastructures en Biologie Santé et Agronomie), et membre de l’infrastructure nationale en biologie santé FRISBI (French Infrastructure in Structural Biology). Cette plateforme comprend quatre spectromètres Raman à double réseaux dont un spécialisé dans l’imagerie, et un spectromètre à simple réseau, ainsi que deux spectromètres FT-Raman. La plateforme met aujourd’hui à la disposition de la communauté scientifique son nouvel équipement de Raman stimulé femtoseconde. Cet appareil permet d’avoir accès à la structure des états excités transitoires générés par l’absorption d’un photon. Dans sa configuration prévue, il permettra de sélectionner le signal d’un état transitoire déterminé, ce qui en fera un banc de mesure à ce jour unique au monde. Vous avez dit nouvel équipement ? Le système repose sur un laser femtoseconde de haute performance, délivrant trois faisceaux distincts, un éclair actinique, qui enclenche une réaction photoinduite dans l’échantillon, qui sera suivi à un temps donné de deux éclairs superposés permettant de mesurer un signal de Raman stimulé, c’est-à-dire la signature vibrationnelle de l’échantillon à cet instant. Avec cet appareil il est donc possible d’étudier la structure des états moléculaires excités et leur évolution dans le temps à l’échelle de la centaine de femtosecondes. La configuration choisie permet de mesurer en parallèle l’évolution de l’absorption et/ou de la fluorescence de l’échantillon. Ce qui fait de lui un modèle unique à ce jour, est la possibilité de choisir la longueur d’onde utilisée pour induire le Raman et se positionner en conditions de résonance, et donc d’observer sélectivement un état excité particulier, pour peu qu’il possède une transition d’absorption spécifique. Cet appareil, conçu par Light Conversion en Lituanie, fait partie de l’infrastructure « ultrafast » du PEPR LUMA (interactions LUmière MAtière) et vient d’être installé au sein de l’I2BC. Il aura pour vocation non seulement l’étude de la réactivité des états excités des photocatalyseurs moléculaires, mais sera largement utilisé pour décrire et comprendre les étapes primaires de la photosynthèse, ces quelques dizaines de picosecondes qui est au cœur de la transduction de l’énergie lumineuse en énergie de potentiel chimique par les plantes, les algues et les bactéries. -> Contact : Bruno Robert (bruno.robert@cea.fr) et Manuel Llansola-Portoles (manuel.llansola@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, en 2019, la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? FOCUS PLATEFORME : Comment les ponts hydrogène modulent les transferts d'énergies photosynthétiques ? Éléments de réponse via la spectroscopie vibrationnelle ! I2BC / Plateforme de spectroscopie RAMAN de résonance. Cette plateforme met à disposition des équipements de spectroscopies avancées Raman et FLN (7 spectromètres, avec plusieurs accessoires, large gamme de température possible (thermostats 273-320 K, cryostats 4-250 K)). Analyses faisables sur échantillons de toutes formes physiques, en particulier ceux qui contiennent des molécules pigmentées (voir ci-dessous). Le laboratoire se spécialise sur les propriétés physico-chimiques des cofacteurs pigmentés en biologie (caroténoïdes, chlorophylles, hèmes, flavines…), y compris des études in vivo des réactions biochimiques, photo-induites et régulatrices. L'état de l'échantillon (liquide, poudre, gel, solide…) limité seulement par la taille du signal (présence de molécules pigmentées nécessaires pour des mesures en milieux complexes). Exemples récents : processus régulateurs dans des membranes photosynthétiques in vivo (feuilles entières et micro-organismes), structure moléculaire des caroténoïdes et opsines dans la rétine humaine ex vivo. Cette plateforme fait partie du pôle des plateformes de Biophysiques de l'I2BC qui comprend les plateformes de RPE, FTIR, Résonance Raman, Spectroscopies Electroniques et Microscopie de fluorescence à super-résolution. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.

|
Scooped by
Life Sciences UPSaclay
April 13, 2025 5:22 PM
|
La création du « réseau des animaleries rongeurs » du périmètre de l'Université Paris Saclay est un pas stratégique et important vers l'avenir. Elle vise à fédérer la communauté des professionnels et des utilisateurs de ces dispositifs, à promouvoir le partage d'expériences et les échanges d'expertise lors de rencontres, d'animation d'événements et de formations. Ce réseau ne cherche pas seulement à bâtir une communauté, mais aussi à engager des discussions essentielles sur des évolutions métiers, la réglementation ou encore les développements technologiques, cruciales pour le fonctionnement global de nos animaleries et la dynamique de nos collectifs. Les avancées technologiques notamment liées à la révolution numérique, les difficultés de gestion des infrastructures (exigences réglementaires, ressources humaines, politiques tarifaires), les attentes sociétales (arrêt des expérimentations, bien-être animal) sont parmi les priorités qui imposent l'évolution rapide et l'adaptation continue du fonctionnement des animaleries. Le réseau se veut un carrefour d'échanges, permettant à tous les acteurs d'accéder à des pratiques innovantes et un partage d'outils et de connaissance. Une première journée du réseau très dynamique ! Le 23 janvier 2025, le réseau a franchi une étape importante avec l’organisation par l'unité IERP de sa première journée d’animation à l'INRAE de Jouy-en-Josas. Cet événement a rassemblé une quinzaine de participants des principaux organismes de recherche (INRAE, CNRS, Sorbonne université, CEA, …) autour de cette initiative dans une volonté d'échange et un besoin de rencontre. Les retours ont été très positifs : des échanges riches et dynamiques ont émergé, permettant aux acteurs présents de partager leurs préoccupations, et d’établir des connexions précieuses pour construire le réseau. Quels sont les premiers sujets abordés ? Après une traditionnelle série de mots de bienvenue (Christelle Langevin, Directrice de l'unité IERP ; Sabine Riffault, Déléguée Régionale INRAE Ile-de-France), deux premières présentations ont été faites, respectivement par i) Serban Morosan, Vice-Président du GIRCOR (La recherche animale en France et en Europe : enjeux et controverses) et Frédéric Dollé, DR CEA et chargé de mission pour le domaine Sciences de la Vie à Santé, VP-Recherche de l’Université Paris-Saclay (PlugInLabs Université Paris Saclay & FOCUS PLATEFORME … avec un regard particulier sur les plateformes en lien avec l’expérimentation animale). La deuxième partie de la journée a été consacrée à une présentation de plusieurs des animaleries « rongeurs » du territoire Paris-Saclay, dont : - ANIMEX (IPSIT, Faculté de pharmacie (Bat. Moissan), Saclay), représenté par Valérie Domergue
- ZOOTECHNIE 151 (ex-animalerie centrale, NeuroPSI, Saclay), représentée par Valérie Lavallée
- IERP (avec ses zootechnies aquatique et rongeur, Centre INRAE de Jouy-en-Josas), représenté par Dimitri Rigadeau, Natalia Maties et Christelle Langevin
- A-PINCUS (IBVB - UMS 44, Kremlin-Bicêtre), représenté par Evelyne Ferrary
- ANIM-C (Institut Joliot, CEA, Centre de Saclay), représenté par Karine Adel-Patient
- PNCA (Campus Agro Paris-Saclay, Saclay) représenté par Gaëlle Champeil-Potokar
Etaient également présents lors de cette première journée, Christelle Enond (pour les animaleries rongeurs de l’UMS 28, Sorbonne Université – INSERM, Paris) et Marie-Elise Schwartz (pour TPS-AQUA, la zootechnie aquatique de TEFOR, NeuroPSI, Saclay). Nos objectifs ? A travers ce réseau, nous aspirons à accroître la visibilité de chaque structure en mettant en avant les expertises et les spécificités de chacun pour révéler les complémentarités et identifier les redondances. En nous réunissant régulièrement, nous avons l'opportunité de promouvoir la découverte de nouvelles pratiques et innovations, tout en bâtissant un avenir plus collaboratif et raisonné dans une même dynamique. La Suite : Une deuxième journée de rencontres à venir ? Encouragés par le succès de cette première journée, nous travaillons déjà à l’organisation d’une seconde rencontre. Nous invitons tous les acteurs concernés à nous rejoindre pour approfondir nos échanges et renforcer cette dynamique collective. La création de ce réseau est une belle opportunité pour tous les acteurs des animaleries rongeurs de l'Université Paris Saclay. Contactez-nous si vous souhaitez nous rejoindre ! -> Contact : Natalia-Ana Maties (natalia-ana.maties@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en juin 2020, mars 2021, mars 2023, mars 2024 et mars 2025, l’IERP publiait ses premiers FOCUS PLATEFORME … Redécouvrez- les aujourd’hui ! INRAE / Infectiologie expérimentale des rongeurs et poissons (IERP). L'infrastructure scientifique collective IERP, labélisée IBISA, a pour principales missions la production et la fourniture d'animaux à statut sanitaire et génétique défini, la réalisation d'expérimentation in vivo en milieu confiné et le phénotypage des animaux en expérimentation. Ouverte à la communauté, elle a un rôle d'interface entre les laboratoires académiques, les industriels et les acteurs de la filière et offre des services standards ou à façon. Les projets hébergés couvrent les domaines de l'infectiologie, l'immunologie, la génétique, la physiologie et le lien entre hôte - microbe et environnement. Les spécificités de l'infrastructure sont donc son ouverture, son expertise en infectiologie associée à la fourniture d'accès à des locaux confinés (NSB2) pour la manipulation d'agents pathogènes et d'OGM; sa pisciculture expérimentale hors sol hébergeant des espèces aquacoles (truite et carpe) et des poissons-zèbres. Le plateau de phénotypage comprend des équipements de pointe distribués au sein même des dispositifs pour limiter le stress lié au transport des animaux et éviter les ruptures de confinement lors des expériences en NSB2. Les technologies proposées couvrent les besoins en imagerie in vivo (à fluorescence et bioluminescence), en transparisation et imagerie à feuille de lumière, en cytométrie d'organismes biologiques complexes (larves de poissons zèbres, organoïdes) et les études comportementales (systèmes home made, Noldus, Viewpoint). L'infrastructure est partie intégrante de réseaux thématiques (Emerg'in, Aquaexcel 3.0, Frontinov, PAHW, InnaSCo) et disciplinaires (RmuI, ImaBio, Rt-mfm, SBEA) nationaux et internationaux.

|
Scooped by
Life Sciences UPSaclay
March 30, 2025 10:02 AM
|
L'accélération et la digitalisation de la chimie sont devenues des priorités stratégiques face aux enjeux croissants en santé, en environnement et en compétitivité économique. Les crises sanitaires récentes, telles que la pandémie de COVID-19, ou encore les enjeux de santé mondiaux tels que les cancers ou les maladies neurodégénératives comme les maladies d’Alzheimer ou de Parkinson ont mis en lumière l'importance de développer rapidement des solutions thérapeutiques innovantes. Parallèlement, l'urgence climatique et les pressions réglementaires liées à la transition écologique imposent à l'industrie chimique de concevoir des processus plus durables et respectueux de l'environnement. L'intégration de technologies numériques, comme l'intelligence artificielle, la modélisation moléculaire ou encore l’expérimentation à haut débit et l'automatisation des laboratoires, permet d'accélérer le développement de nouvelles molécules, matériaux et procédés tout en réduisant les coûts et l'empreinte écologique. Cependant, le plein potentiel de ces intégrations nécessite une maitrise de l’état de l’art et de l’expertise de telles technologies, ce qui pourrait être accéléré par une mutualisation des efforts au sein de la communauté. C'est dans cette optique qu'a été créée en octobre dernier l'Association Francophone de la Chimie Accélérée et Digitalisée (AFCAD). Cette initiative vise à rassembler les acteurs plateformes académiques, industriels et institutionnels autour d'une vision commune : promouvoir l'innovation, partager les meilleures pratiques et relever ensemble les défis scientifiques et sociétaux liés aux technologies qui entourent la chimie de demain et plus particulièrement de la chimie digitalisée et accélérée grâce aux méthodes haut-débit (high-throughput) et potentiellement à l’automatisation. A travers l’organisation de journées thématiques, de workshops, et d’activités diverses, l'AFCAD ambitionne de devenir une plateforme favorisant une meilleure collaboration entre chercheurs, industriels et décideurs en facilitant le partage des données et des connaissances. L’objectif ultime de l’association est ainsi de renforcer l’impact et la visibilité de ce domaine stratégique au sein de la communauté. Le comité d’organisation de l’association est actuellement constitué de Bernard Roux (SYENSQO - France), Florian Medina (JnJ - Belgique), Claire Ceballos (ESCOM - France), Pascal Miéville (EPFL - Suisse), Patrick Pasau (UCB Biopharma – Belgique) et Eugénie Romero-Laboureur (CEA – France), représentant notamment le périmètre Paris-Saclay via la Plateforme d’expérimentation à haut débit (HTE) (Service de Chimie Bioorganique et de Marquage (SCBM, Département Médicaments et technologies pour la Santé, Institut Joliot, CEA, Centre de Paris-Saclay). Les chantiers en cours sont i) la mise en place dans un futur proche d’un site internet (avec l’aide de François Pacquet, BetterChem - France), et ii) un programme d’animations pour l’année 2025. Aussi, les adhésions 2025 sont ouvertes. Pour adhérer à l’association, ou pour toute question ou suggestion, n’hésitez pas à contacter le comité via le secrétariat à c.ceballos@escom.fr. -> Contact : Eugénie Romero-Laboureur (eugenie.romero@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, en avril 2022 et janvier 2023, la plateforme publiait ses premiers FOCUS PLATEFORME ! Les relire ? MTS / Plateforme d’expérimentation à haut débit (HTE). L’expérimentation à haut débit (High-Throughput Experimentation - HTE) consiste en la miniaturisation et la parallélisation de réactions chimiques à une échelle standard de 10 µM pour la découverte ou l’optimisation de réactions, permettant un gain de temps et d’argent considérables, tout en augmentant drastiquement le nombre de combinaisons possibles (format standard : plaque 96 puits). Inspiré de la plateforme HTE de l’Université de Pennsylvanie (Philadelphie), le CEA s’est récemment doté de l’expertise et de l’instrumentation nécessaires pour permettre le développement de nouvelles méthodologies de synthèse ainsi que la résolution de transformations chimiques difficiles. La plateforme d’expérimentation à haut débit, créée en 2021, est accessible aux utilisateurs du monde académique comme industriel. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
March 16, 2025 6:22 PM
|
Microcèbe : la petite sentinelle scientifique du CREMm. Le microcèbe n’a pas fini de dévoiler tous ses secrets ! Animal modèle hors pair au cœur de l’expertise du Centre de recherche sur Microcebus murinus (CREMm), il ouvre de vastes perspectives en biologie et médecine humaine. Le CREMm est soutenu par le GIS IBISA depuis 2015 pour son originalité et son savoir-faire, à la disposition des équipes de recherche du monde entier. Lire l'article. Marie-Pierre Dubrulle passe le flambeau de la qualité. Qualiticienne du GIS IBiSA depuis sa création, Marie-Pierre Dubrulle part bientôt à la retraite. Le 17 mars 2025, elle animera une dernière fois la réunion du comité de pilotage du réseau IQuaRe, dont elle est l'instigatrice. IBiSA soutient le travail exceptionnel qu'elle a accompli toutes ces années. Le réseau IQuaRe. Cécile Pouzet, bientôt aux commandes de la qualité. Responsable opérationnelle de la plateforme Toulouse réseau imagerie (TRI) et auditrice au sein du réseau IQuaRe depuis 2006, Cécile Pouzet a été désignée pour prendre en charge une partie des missions qualité du GIS IBiSA. Elle a déjà assisté aux réunions de février et mars du comité de direction. La mission qualité. De RIO à IBiSA, la qualité sur les plateformes technologiques. Retour sur une interview de Marie-Pierre Dubrulle, qui explique d'où vient la qualité sur les plateformes du GIS IBiSA. Une mission plus ancienne que le GIS lui-même, menée avec dévotion et expertise au bénéfice des utilisateurs des plateformes labellisées RIO avant de devenir IBiSA. Lire l’article. Les appels d’offres IBiSA 2025 sont toujours ouverts. L’appel d’offres Plateformes et l’appel à projets CRB sont encore ouverts, respectivement jusqu’au 7 et 14 avril 2025. Pour consulter les modalités et candidater, c’est par ici ! En savoir plus. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Plus d'infos sur le GIS IBiSA. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
March 2, 2025 11:39 AM
|
L’unité INRAE / Infectiologie Expérimentale des Rongeurs et Poissons (IERP) et l'unité ENS Paris Saclay/ Lumière, Matière et Interfaces (LuMIn) ont lancé l'initiative MesoSPIM Paris-Saclay en septembre dernier. Il est possible chimiquement de rendre transparents des échantillons biologiques fixés pour les imager en profondeur par des techniques de transparisation. Co-développées avec des dispositifs innovants de microscopie tridimensionnelle (3D), ces techniques ont révolutionné un grand nombre de domaines de la biologie en permettant la visualisation 3D d'objets biologiques complexes et de grande taille à des résolutions cellulaires. L’IERP a été leader dans le développement de ces techniques pour des applications en Santé Animale à INRAE. Face aux limites instrumentales actuelles, l'IERP et le LuMIn se sont associés pour s'engager dans l'aventure MesoSPIM : une communauté open-source (https://mesospim.org/) qui propose les plans de montage et les logiciels d’acquisition de Microscopes basés sur l’Illumination Sélective d'un Plan par une nappe de lumière (SPIM) capables d’imager de grands échantillons transparisés à une échelle mésoscopique (du mm au cm). La communauté MesoSPIM propose plusieurs configurations de microscopes, parmi lesquelles nous avons fait le choix du système Benchtop pour des critères de dimension maximale de l’échantillon observable, de résolution transversale et axiale, de modularité de configuration (passage d’échantillons de tailles variées, régimes de résolution…), d’optimisation de la vitesse d’acquisition, et enfin de volumétrie des données acquises, nécessitant la prise en compte du stockage des données. A ce jour, 33 systèmes ont été répertoriés à travers le monde et celui de l'Université Paris Saclay sera le 1er en France. L’implémentation du système a été initié en Septembre 2024 dans les locaux de l'ENS Paris Saclay. Après 4 mois et grâce à l'implication de Tristan Brechignac, étudiant de SupOptique co-encadré par les biologistes et les physiciens du projet, les premiers échantillons ont été imagés avec succès ! Le MesoSPIM a été déménagé dans les locaux de l'IERP début 2025 et accueillera les scientifiques de la communauté dès son inauguration le 6 juin prochain à INRAE, Jouy en Josas https://mesospim-upas.sciencesconf.org La configuration BenchTop (petite taille et transportable), sera idéale pour l'organisation de formations en imagerie, d'ateliers pratiques (MiFoBio) et de démonstrations lors de journées d’animation scientifiques. Les spécificités du système sont : i) Imagerie de grands volumes : Capable d'imager des échantillons de grande taille (plusieurs cm) avec une excellente qualité d'image (résolution spatiale 1.5 µm en latéral, 3.3 µm en axial sur l'ensemble du champ d'observation) ; ii) Axe d’illumination bilatéral, détection horizontale, rotation de l’échantillon à 360° ; iii) Adaptabilité : Compatible avec divers milieux de transparisation et indices de réfraction (cuves d'observation de formes et dimensions variables) ; iv) Vitesse d'acquisition : Acquisition rapide des données volumétriques ; v) Communauté open-source : Développement et partage des caractéristiques techniques au sein de la communauté MesoSPIM; vi) Flexibilité : Le microscope est modulaire, ce qui permet de l'adapter facilement à différents types d'échantillons et de configurations expérimentales. En savoir plus ? Vladimirov et al., Nat Commun 2024 -> Contacts : Christelle Langevin (christelle.langevin@inrae.fr) et François Marquier (francois.marquier@ens-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Enfin, en juin 2020, mars 2021, mars 2023 et mars 2024, l’IERP publiait ses premiers FOCUS PLATEFORME… Redécouvrez- les aujourd’hui ! INRAE / Infectiologie expérimentale des rongeurs et poissons (IERP). L'infrastructure scientifique collective IERP, labélisée IBISA, a pour principales missions la production et la fourniture d'animaux à statut sanitaire et génétique défini, la réalisation d'expérimentation in vivo en milieu confiné et le phénotypage des animaux en expérimentation. Ouverte à la communauté, elle a un rôle d'interface entre les laboratoires académiques, les industriels et les acteurs de la filière et offre des services standards ou à façon. Les projets hébergés couvrent les domaines de l'infectiologie, l'immunologie, la génétique, la physiologie et le lien entre hôte - microbe et environnement. Les spécificités de l'infrastructure sont donc son ouverture, son expertise en infectiologie associée à la fourniture d'accès à des locaux confinés (NSB2) pour la manipulation d'agents pathogènes et d'OGM; sa pisciculture expérimentale hors sol hébergeant des espèces aquacoles (truite et carpe) et des poissons-zèbres. Le plateau de phénotypage comprend des équipements de pointe distribués au sein même des dispositifs pour limiter le stress lié au transport des animaux et éviter les ruptures de confinement lors des expériences en NSB2. Les technologies proposées couvrent les besoins en imagerie in vivo (à fluorescence et bioluminescence), en transparisation et imagerie à feuille de lumière, en cytométrie d'organismes biologiques complexes (larves de poissons zèbres, organoïdes) et les études comportementales (systèmes home made, Noldus, Viewpoint). L'infrastructure est partie intégrante de réseaux thématiques (Emerg'in, Aquaexcel 3.0, Frontinov, PAHW, InnaSCo) et disciplinaires (RmuI, ImaBio, Rt-mfm, SBEA) nationaux et internationaux.

|
Scooped by
Life Sciences UPSaclay
February 8, 2025 9:51 AM
|
PAPPSO est une plateforme experte dans l'analyse des protéomes, qui se distingue, entre autres, par sa capacité à analyser de grandes cohortes d'échantillons très complexes. Pour repousser encore ses limites et pouvoir réaliser des analyses à haut-débit, PAPPSO s'équipera prochainement d'un nouveau spectromètre de masse ultra-rapide et sensible. Cet instrument sera installé courant 2025 à l'IDEEV, dans les locaux du site de PAPPSO adossé à l'UMR Génétique Quantitative et Evolution - Le Moulon. L'achat de ce nouvel équipement sera réalisé grâce à l'obtention de plusieurs cofinancements, dont 514 k€ issus de la région Île-de-France. Cette subvention vient récompenser le projet ProteHAD pour "PROTEomique à Haut-débit pour l'Agriculture de Demain", lauréat de l'appel d'offre SESAME 2024. Au travers de ce projet, PAPPSO vise à promouvoir la protéomique comme un outil d’analyse de premier plan dans les recherches menées actuellement dans les domaines de l’amélioration des plantes et de l’agroécologie. Utilisée dans des approches de biologie des systèmes, la protéomique représente en effet une source unique d’informations moléculaires pour expliquer, modéliser et prédire le phénotype des plantes en réponse à des interactions biotiques ou abiotiques ainsi que les relations plante-sol. La biologie des systèmes nécessite de prendre en compte la diversité génétique des espèces étudiées par la mise en œuvre de dispositifs expérimentaux comportant un très grand nombre d’échantillons. Grâce à l'acquisition d'un spectromètre de masse de dernière génération, PAPPSO pourra analyser en routine le protéome de grandes cohortes d’échantillons, sans faire de compromis sur les protéines de faible abondance telles que celles impliquées dans la régulation de l’expression des gènes. Au niveau de Paris-Saclay, ce nouvel équipement permettra de structurer une offre analytique unique en science du végétal, ouvrant ainsi la voie à des projets de recherche ambitieux et innovants. -> Contact : Mélisande Blein-Nicolas (melisande.blein-nicolas@inrae.fr), Céline Henry (celine.henry@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Envie de (re)lire les précédents FOCUS PLATEFORME de PAPPSO ? FOCUS PLATEFORME : Plateforme d'Analyses Protéomiques de Paris Sud-Ouest (PAPPSO) : de la Protéomique à la Métaprotéomique, à la recherche de biomarqueurs ! (25-nov.-19) FOCUS PLATEFORME : Un nouvel élan pour la Plateforme d’Analyse Protéomique de Paris Sud-Ouest (PAPPSO) (29-mai-23). Plateforme d’Analyse Protéomique de Paris Sud-Ouest (PAPPSO). L'objectif de la Plateforme d'Analyse Protéomique de Paris Sud-Ouest (PAPPSO) est de mettre à disposition des équipes qui s'adressent à elle un équipement, une compétence scientifique et un savoir-faire adaptés aux questions posées, depuis les plus simples (identification de protéines provenant d'un organisme entièrement séquencé) jusqu'aux plus complexes (quantification relative ou semi-absolue des variations, dynamique des modifications post-traductionnelles,...), dans le cadre de collaborations et de prestations. PAPPSO réunit deux plateaux techniques complémentaires. L'un, adossé à l'UMR Génétique Quantitative et Evolution ? Le Moulon (Gif-sur-Yvette), est spécialisé dans la biologie végétale (espèces modèles et cultivées), et l'autre, adossé à l'UMR Microbiologie de l'Alimentation au service de la Santé (MICALIS, Jouy-en-Josas), est spécialisé en microbiologie, biologie animale (modèles bovin, murin) et, plus ponctuellement, en biologie humaine. Ses équipements de toute dernière génération permettent à PAPPSO de proposer la réalisation d'analyses par spectrométrie de masse, avec ou sans pré-fractionnement des protéines ou des peptides, et avec ou sans marquage isotopique. PAPPSO s'est spécialisée dans le haut débit (analyse quantitative de cohortes de grande taille), dans l'analyse d'échantillons très complexes (métaprotéomique), et dans la peptidomique. Elle développe les outils de bioinformatique et d'analyse de données qui permettent de traiter ce type d'expérience, et accompagne les utilisateurs dans l'interprétation de leurs données.

|
Scooped by
Life Sciences UPSaclay
January 26, 2025 9:53 AM
|
La plateforme Observatoire du Végétal - Chimie Métabolisme dite OV-Chimie (Institut Jean Pierre Bourgin - Sciences du végétal, IJPB, centre INRAE de Versailles-Saclay) est dédiée à l’analyse des petites molécules du végétal, grâce notamment au développement d’analyses métabolomiques ciblée et non ciblée par spectrométrie de masse. Le projet CONNEXION, porté conjointement par l’OV-Chimie et l’Institut Lavoisier de Versailles (ILV), appuyé par le consortium SMaCS et soutenu par l’Université Paris-Saclay, a été proposé courant 2024 à la région Ile-de-France dans le cadre de l’appel d’offre SESAME. CONNEXION s’inscrit dans le développement de la bioéconomie, avec pour objectifs de contribuer à produire de la biomasse en quantité et qualité et de la transformer en molécules à haute valeur ajoutée, dont certaines pour usage agricole ou leur rôle dans la nutrition, grâce à des méthodes d’analyse chimique de pointe comme la spectrométrie de masse (MS). CONNEXION vient d’obtenir une subvention de 500 k€ de SESAME, qui s’ajoutant aux financements acquis (fonds propres des unités porteuses, GIS IBISA, EUR SPS, ANR, CNRS, AAP Emergence…) devrait permettre l’acquisition en 2025 de deux spectromètres de masse, dont un de très haute résolution couplé à une chromatographie liquide (LC) bidimensionnelle (LC/extraction sur phase solide en ligne (LC/SPE en ligne) + LC Ultra Haute Performance UHPLC), pour l’OV-Chimie. L’achat de cet équipement permettra une caractérisation plus exhaustive des composés présents tels que les métabolites spécialisés, les lipides ou les oligosaccharides afin i) d’acquérir une meilleure connaissance des mécanismes qui sous-tendent la capacité des plantes à s’adapter à des conditions extrêmes et se défendre contre les attaques pathogènes, ii) de découvrir de nouvelles molécules bioactives utilisables en agriculture, nutrition et/ou en pharmacologie, et iii) de mieux caractériser les produits issus des cultures pour développer les filières industrielles du biosourcé. Selon la configuration utilisée, le nouvel équipement permettra par la SPE en ligne de réduire les étapes chronophages de préparation des échantillons, ou par la deuxième séparation chromatographique de multiplier le pouvoir de séparation et donc l’exhaustivité de l’analyse. Cet appareil pourra aussi être utilisé pour toute approche de métabolomique ou lipidomique appliquée aux sciences de la vie. -> Contacts : François Perreau (francois.perreau@inrae.fr) et Aline Voxeur (aline.voxeur@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, le 13 janvier 2020, la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? FOCUS PLATEFORME : L'analyse des métabolites spécialisés des plantes : un outil pour l'étude des interactions plantes/microorganismes, une spécificité de la plateforme Chimie-Métabolisme-Métabolome de l'Observatoire du Végétal IJPB / Observatoire du végétal - Plateforme Chimie-Métabolisme. La plate-forme "Observatoire du végétal, Chimie-Métabolisme" propose la quantification, la caractérisation et l'identification de composés chimiques du végétal : les hormones végétales et autres molécules signalisantes, les métabolites centraux (sucres, acides aminés, acides organiques) ou spécialisés (flavonoîdes, caroténoïdes, sphingolipides, glucosinolates, ...), ainsi que les composés issus de la biomasse végétale (oligosaccharides, paroi végétale, polyphénols,...). Elle se fonde sur l'expertise de ses agents en analyse de chimie du végétal, ciblée et non ciblée et en traitement des données (statistiques, réseaux moléculaires) pour la métabolomique, la lipidomique et l’oligoglycomique. Elle s'appuie aussi sur des appareils de chromatographie, notamment couplés à de la spectrométrie de masse performante (GC-MS, UPLC-MS/MS, UPLC-Q-TOF). La plate-forme a noué plusieurs collaborations industrielles ces dernières années (LVMH, Syngenta, Nestlé, Sederma).

|
Scooped by
Life Sciences UPSaclay
January 12, 2025 5:19 PM
|
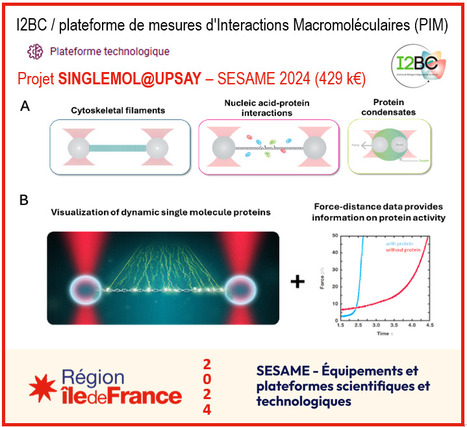
La plateforme de mesures d'Interactions Macromoléculaires (PIM) a été créée en 2010 et résulte de l’extension d’un service de microcalorimétrie mis en place dès 2000 sur le périmètre Paris-Saclay. Cette plateforme fait aujourd’hui partie des 17 plateformes (https://www.i2bc.paris-saclay.fr/facilities/) de l’Institut de Biologie Intégrative de la Cellule (I2BC, Gif-sur-Yvette, Institut Joliot, CEA, Saclay). Redécouvrez toutes ces plateformes via un FOCUS PLATEFORME dédié ! L'objectif de la plateforme de mesures d'Interactions Macromoléculaires (PIM) est de proposer aux utilisateurs un ensemble unique de techniques permettant de détecter et de quantifier les interactions entre molécules biologiques (interactions protéine-ligand, protéine-protéine, protéine-acides nucléiques…). Cette plateforme fait partie de l’infrastructure nationale FRISBI et est labellisée IBISA (pôle BioStruct@UPsay, I2BC). Le financement obtenu dans le cadre de l’AAP SESAME 2024 (projet SINGLEMOL@UPSACLAY) porte sur l'installation du premier instrument de mesures des interactions macromoléculaires à l'échelle de la molécule unique sur une plateforme académique ouverte. Des entreprises privées ont récemment mis au point des technologies disruptives qui permettent d'effectuer ces analyses à l'échelle de la molécule unique au sein d'un seul instrument, rendant la technologie accessible à un plus grand nombre d'utilisateurs. Ces appareils de mesures par pinces optiques, tels que le système C-Trap® (Lumicks), constituent une révolution dans le domaine car il intègre au sein d'un seul instrument la microfluidique, la microscopie en temps réel, et les mesures de force à l'échelle du picoNewton. En complément du financement SESAME de la région Ile-de-France, ce projet s'appuie sur un soutien financier important de FRISBI, de l'I2BC, de l'institut Jacob du CEA (Fontenay aux Roses) et de l'Institut Pasteur (Paris). Cet investissement est, de plus, très complémentaire des investissements majeurs réalisés ces dernières années à l'I2BC et à SOLEIL en cryomicroscopie électronique, en imagerie super-résolution et FIB-SEM et en mesure d’interactions des protéines, car il permettra d'apporter des informations fonctionnelles cruciales sur les assemblages moléculaires au cœur des mécanismes du vivant. Le C-Trap® … comment cela fonctionne-t-il ? Dans le C-Trap®, deux faisceaux de lumière focalisée sont générés et sont suffisamment puissants pour piéger des billes microscopiques entre lesquelles sont fixés des filaments nucléiques ou protéiques (actines, tubulines). Les faisceaux permettent également de piéger des séparations de phase liquide-liquide ou condensats (Figure 1A). Le C-Trap ou les technologies équivalentes de dernière génération incorporent donc dans un même ensemble un système micro-fluidique intégré qui sépare les réactifs, via des canaux à flux laminaire voisins, sans barrières physiques, avec un système de microscopie qui permet la visualisation simultanée et en temps réel de molécules isolées et marquées par fluorescence (Figure 1B). Ils offrent enfin la possibilité d'étirer et de relâcher les molécules capturées entre deux billes. Selon la nature des objets piégés par les pinces optiques (billes greffées, gouttelettes), différentes applications sont possibles. La technologie proposée permettra par exemple, i) de visualiser par microscopie de fluorescence le déplacement de protéines ou d’assemblages moléculaires complexes sur des filaments nucléiques ou protéiques, greffés sur des billes piégées entre des pinces optiques, ii) de caractériser les forces mises en œuvre lors de changements de conformations de ces filaments nucléiques ou protéiques, induits par des moteurs moléculaires (Figure 1B), iii) de mesurer les propriétés physico-chimiques de viscosité de condensats capturés entre ces pinces optiques, et iv) d'exercer des forces sur des récepteurs de cellules déposés sur une surface et d'analyser les cascades de signalisation produites. Légende Figure : Décrypter les mécanismes moléculaires à l’échelle de la molécule unique. A Représentation des différents modes expérimentaux exploitant l'utilisation de pinces optiques combinées à la microscopie confocale pour étudier les interactions dynamiques au niveau de la molécule unique. B (Gauche) Représentation des protéines interagissant dynamiquement avec l'ADN. Les lignes vertes représentent le déplacement en temps de plusieurs protéines fluorescentes interagissant avec un ADN double brin fixé entre deux billes piégées par des lasers. (Droite) Exemple de courbe force-vs-distance montrant l'activité d'une protéine dissociant de l'ADN double-brin et formant de l'ADN simple brin moins résistant. Cet investissement fédère 27 équipes de 12 instituts en Île-de-France, qui ont, pour certaines d'entre elles, une expertise reconnue dans l'étude des molécules uniques. Cet instrument sera disponible pour les acteurs de la recherche publique ou privée en Île-de-France et sur le territoire national. Il s'agit d'une technologie de rupture qui accroîtra de façon importante le rayonnement de la région autour de l'étude des mécanismes moléculaires et cellulaires, dans le domaine des Sciences du Vivant et de la Santé. Cette technologie de pointe viendra renforcer le parc d'instruments uniques de l'Université Paris-Saclay, accessibles via des plateformes ouvertes appartenant aux infrastructures nationales FRISBI et FranceBioImaging. -> Contacts : Stéphanie Marsin (stephanie.marsin@i2bc.paris-saclay.fr) ou simplement plateforme-pim@i2bc.paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ICI Envie de (re)lire les précédents FOCUS PLATEFORME de PIM ? I2BC / Plateforme de mesures d'Interactions Macromoléculaires (PIM). La Plateforme de mesures d'Interactions Macromoléculaires (PIM) de l'I2BC offre un large éventail d'équipements et d'expertises en biochimie et biophysique pour réaliser des mesures de contrôles qualités ainsi que des analyses fonctionnelles d'échantillons de protéines : microcalorimétrie, ultracentrifugation analytique (UCA), MST, SEC-RALS-LALS, BLI, SPR, switchSENSE. Les prestations offertes par la plateforme concernent : 1) La caractérisation biophysique de protéines ou de complexes biologiques (SEC-RALS-LALS, UCA), 2) L'analyse d'interactions biologiques (ITC, SPR, BLI, switchSENSE, MST), 3) L'analyse de la stabilité des macromolécules et des complexes biologiques pour notamment aider à la formulation (DSC, DSF). Les appareils et prestations sont mis à disposition de l'ensemble des laboratoires de l'Université Paris-Saclay, des autres laboratoires académiques ainsi qu'aux industriels. Pour plus d'informations concernant ces prestations : plateforme-pim@i2bc.paris-saclay.fr. Cette plateforme fait partie du pôle des plateformes de Biologie Structurale de l'I2BC qui comprend : i) les plateformes de Cristallisation, RMN, CryoEM, Mesures d'Interactions Macromoléculaires, la génération et production de protéines artificielles (αRep) et ii) les plateaux techniques d'Expression de protéines solubles ou membranaires en levures, Expression des protéines en cellules d'insectes. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles.

|
Scooped by
Life Sciences UPSaclay
December 15, 2024 5:10 PM
|
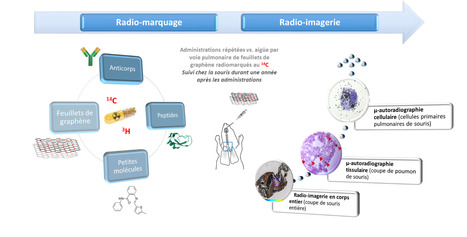
Les matériaux à base de graphène suscitent un intérêt considérable en raison de leurs propriétés uniques et polyvalentes dans des secteurs aussi variés que l'énergie, l'électronique, la photonique, ou encore les applications biomédicales. Cependant, la production et l'utilisation croissantes de ces matériaux nécessitent une évaluation approfondie de leur impact potentiel sur la santé humaine. L’étude collaborative entre la plateforme de marquage isotopique et la plateforme imagerie ex-vivo (beta-imagerie) du Département Médicaments et Technologies pour la Santé – DMTS (Institut Joliot, CEA Paris-Saclay, Gif-sur-Yvette) a démontré que le radio-marquage combiné à la radio-imagerie sont des techniques puissantes pour évaluer la biodistribution de composés au sein de l'animal entier jusqu'au niveau cellulaire (Sallustrau et al., Nanoscale 2023). En effet, ce travail donne un aperçu de l'impact chez la souris, sur une période d'une année, d'une exposition répétée à de faibles doses versus d'une exposition aiguë à une forte dose de feuillets de graphène administrés par voie respiratoire. Les résultats ont montré que la majeure partie de la radioactivité se retrouvait dans les poumons dans les deux cas, avec des temps d'élimination plus longs dans le cas de l'administration aiguë à forte dose. Pour la première fois, la μ-autoradiographie a été utilisée pour détecter la présence de feuillets de graphène dans les cellules pulmonaires isolées des poumons de souris exposées. Dans l'ensemble, ces résultats montrent l'accumulation à long terme du matériau dans les poumons sur une année, quel que soit le protocole d'administration (répétées vs. aiguë), et la biopersistance plus élevée du matériau en cas d'exposition aiguë à forte dose. De façon intéressante, nous avons observé que, contrairement à d’autres nanomatériaux carbonés étudiés (nanotubes de carbones, graphène oxydé), les feuillets de graphène traversent très peu, voir pas, la barrière air-sang pulmonaire vers d’autres organes (foie, rate, reins…), probablement du fait de leur architecture en feuillet. Le marquage isotopique est souvent associé au développement de médicaments car il offre aux chercheurs un moyen quantitatif et hautement sensible d’évaluer le devenir in vivo de molécules d’intérêt biologique. Les isotopes de l’hydrogène (2H, 3H) et du carbone (11C, 13C, 14C) sont des références en termes de traceurs mais leur incorporation au sein des molécules par radiosynthèse peut se révéler complexe et coûteuse. La plateforme de marquage isotopique du Service de Chimie Bioorganique et de Marquage -SCBM dispose d’une expertise unique dans le radiomarquage de petites molécules organiques et de nanoparticules carbonées. Cette dernière a récemment mis au point plusieurs méthodes de marquage innovantes et efficaces permettant de marquer des molécules bioactives ainsi que des médicaments par les isotopes du carbone et de l’hydrogène (2H, 13C, 3H, 14C), ceci en dernière étape de synthèse, limitant ainsi les coûts et les déchets. Dans le cadre particulier du marquage de nanoparticules carbonées (nanotubes de carbones, graphène) le carbone-13 (13C, isotope stable) et le carbone-14 (14C, isotope radioactif émetteur de β-, T1/2 = 5730 ans), sont des isotopes particulièrement intéressants car ils peuvent être incorporés au sein de la structure de la nanoparticule sans en modifier les propriétés physico-chimiques, permettant ainsi d’étudier leur comportement et leur devenir in vitro et in vivo. La plateforme imagerie ex-vivo (beta-imagerie) du Service d’Ingénierie Moléculaire pour la Santé - SIMoS possède deux équipements uniques de radio-imagerie (µ- et β-imager 2000 de chez Biospace Lab) capables de détecter des isotopes radioactifs (3H, 14C et 99mTc) avec une très grande sensibilité, de l’ordre de la femtomole, et une résolution adaptée aux isotopes de faibles énergies (3H et 99mTc, 10-100 µm). Les échantillons analysés sur ces appareils sont divers, comprenant des gels SDS-PAGE transférés sur membranes PVDF, des coupes d’organes, ou encore des coupes de corps entier obtenues après l’administration in vivo des composés radiomarqués chez le petit animal. Ainsi, pour un composé radiomarqué donné, il est possible i) d’évaluer l’impact de la voie d’administration (intraveineuse, intrapéritonéale, sous-cutanée, intranasale, intra-trachéale, intracérébroventriculaire) sur sa pharmacocinétique et sa biodistribution, ii) d’observer son éventuel passage des barrières biologiques (air-sang, hémato-encéphalique, materno-fœtale), et même iii) de repérer les zones tissulaires précises, telle que les aires cérébrales, dans lesquelles il rencontre ses cibles d’interaction privilégiées. Besoin d’une expertise sur ces sujets, ou envie d’une collaboration ? N’hésitez pas à nous contacter ! -> Contact : Antoine Sallustrau (antoine.sallustrau@cea.fr), Mathilde Keck (mathilde.keck@cea.fr), ou encore « Plateformes de marquage isotopique et d’imagerie ex-vivo » (Plateforme-Isotopes-PK-Biodis@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Plateforme de marquage isotopique. Cette plateforme possède une expertise (unique sur Paris-Saclay) en synthèse de composés marqués avec des isotopes stables (2H / deutérium et 13C / carbone-13) et par des isotopes radioactifs de type bêta (3H / tritium, 14C / carbone-14 et 125I / iode-125. Forte de son expertise dans la préparation (synthèse, contrôle de qualité) et formulation de molécules marqués, elle assure régulièrement des prestations et collaborations, académiques comme industrielles, dans le domaine du (radio)marquage moléculaire. Elle offre également à la demande, son expertise et environnement unique de travail (laboratoires « chauds », équipements dédiés) pour l’analyse et la caractérisation d’échantillons radioactifs : mesure de puretés chimique et radiochimique par HPLC, détermination d'enrichissements isotopiques et d'activités spécifiques par SM, analyse et détermination structurale par RMN liquide comme solide, mesure d’activités radioactives par comptage à scintillation. Enfin, la plateforme propose des solutions pour le traitement de déchets liquides radioactifs, en particulier ceux contenant du carbone-14 et du tritium. Plateforme imagerie ex-vivo (beta-imagerie). Le marquage de biomolécules avec des isotopes tels que le tritium ou le carbone-14 permet d'établir les propriétés pharmacocinétiques ainsi que la biodistribution tissulaire de différents types de molécules que ce soit dans un contexte d'études de toxicité (nanotoxicologie, neurotoxicité) ou de développement pré-clinique de molécules thérapeutiques. Ce dernier objectif peut être atteint en utilisant des radio imageurs numériques, tels que ceux développés par la société Biospacelab (BetaIMAGER® TRACER). Le bêta-imageur permet d'acquérir des images en temps réel, avec une sensibilité de comptage extrême (0,007 cpm/mm2 pour le tritium et 0,01 cpm/mm2 pour le carbone-14) et une quantification absolue du signal. Ces études de biodistribution sont typiquement effectuées sur des modèles rongeurs et permettent d'adresser des questions concernant par exemple la capacité des molécules étudiées à passer les barrières physiologiques (intestinales, hémato-encéphaliques ou materno-fétale) ou à transloquer d'un compartiment tissulaire vers un autre. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric-Joliot étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Ces derniers sont conduits sur des plateformes techniques de premier plan, pour la plupart labélisées IBiSA ou intégrées dans des Infrastructures Nationales en Biologie et Santé (INBS) : France Life Imaging (imagerie biomédicale), FRISBI (biologie structurale intégrée), MétaboHUB (métabolomique) et NeurATRIS (recherche translationnelle en neurosciences). Joliot est implanté principalement sur le centre CEA-Paris-Saclay, mais aussi Orsay et Gif-sur-Yvette en ce qui concerne le territoire Paris-Saclay.

|
Scooped by
Life Sciences UPSaclay
December 1, 2024 4:19 PM
|
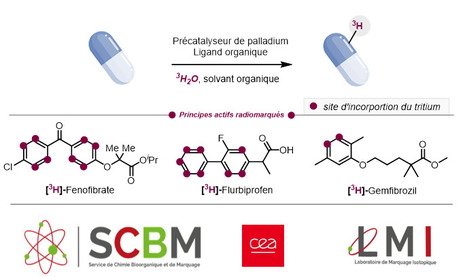
Vous avez dit marquage isotopique par le tritium ? Découvrez ci-dessous une nouvelle méthode mise au point au sein du Service de Chimie Bioorganique et de Marquage (SCBM, Département Médicaments et technologies pour la Santé, Institut Joliot, CEA, Centre de Paris-Saclay) et tout particulièrement sa plateforme de marquage isotopique, le tout dans le contexte d’une collaboration internationale, avec des chercheurs de l’Indian Institute of Technology Bombay et de l’Université de Fribourg. Les molécules deutérées et tritiées connaissent une utilisation croissante dans des domaines variés tels que la santé, l'environnement et l'affichage. L'incorporation de deutérium dans les principes actifs permet notamment de ralentir leur vitesse de métabolisation, réduisant ainsi les doses à administrer aux patients et les effets secondaires associés à leur ingestion. Dans le domaine de l'affichage, l'utilisation de molécules deutérées permet d'augmenter significativement la durée de vie et, dans certains cas, les performances des écrans OLED (Organic Light Emitting Diodes). En recherche pharmaceutique, l'incorporation d'atomes de tritium dans un candidat médicament fournit des informations essentielles pour sa mise sur le marché, notamment concernant sa distribution, son absorption et son excrétion in vivo. L'un des défis majeurs pour les chimistes aujourd'hui est de développer des méthodes efficaces et, si possible, à large spectre, permettant d'incorporer des atomes de deutérium et de tritium dans des molécules complexes (médicaments, fluorophores...) en une seule étape de synthèse à partir de la molécule d'intérêt. Dans ce contexte, un travail collaboratif entre des équipes de recherche indiennes et allemandes et des chercheurs de la plateforme de marquage isotopique a permis de mettre au point une nouvelle méthode de marquage efficace et polyvalente. Cette approche permet l'incorporation d'un grand nombre d'atomes de deutérium et de tritium sur des molécules complexes en utilisant l'eau lourde ou l'eau radiomarquée au tritium comme source isotopique. La méthode développée utilise un précatalyseur de palladium et un ligand disponibles commercialement et stables à l'air ce qui facilite sa mise en œuvre. Cette combinaison permet d'échanger plusieurs atomes d'hydrogène par des atomes de deutérium ou de tritium sur des molécules complexes telles que les principes actifs. En savoir plus ? Chitrala et al., Angew. Chem. Int. Ed. 2024. Ces avancées pourraient faciliter l'accès à des molécules à haute valeur ajoutée, notamment des standards internes pour la quantification ou des analogues radiomarqués de candidats médicaments. -> Contact : Grégory Pieters (gregory. pieters@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Plateforme de marquage isotopique. Cette plateforme possède une expertise (unique sur Paris-Saclay) en synthèse de composés marqués avec des isotopes stables (2H / deutérium et 13C / carbone-13) et par des isotopes radioactifs de type bêta (3H / tritium, 14C / carbone-14 et 125I / iode-125. Forte de son expertise dans la préparation (synthèse, contrôle de qualité) et formulation de molécules marqués, elle assure régulièrement des prestations et collaborations, académiques comme industrielles, dans le domaine du (radio)marquage moléculaire. Elle offre également à la demande, son expertise et environnement unique de travail (laboratoires « chauds », équipements dédiés) pour l’analyse et la caractérisation d’échantillons radioactifs : mesure de puretés chimique et radiochimique par HPLC, détermination d'enrichissements isotopiques et d'activités spécifiques par SM, analyse et détermination structurale par RMN liquide comme solide, mesure d’activités radioactives par comptage à scintillation. Enfin, la plateforme propose des solutions pour le traitement de déchets liquides radioactifs, en particulier ceux contenant du carbone-14 et du tritium. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.
|

|
Scooped by
Life Sciences UPSaclay
June 15, 2025 11:12 AM
|
Le service hospitalier Frédéric Joliot (SHFJ, Institut Joliot, Orsay), avec l’UMR BioMaps (UMR CEA/CNRS/Inserm/Université Paris-Saclay) s’est doté d’un nouvel imageur par Tomographie par émission de positrons (TEP) associé à la tomodensitométrie (TDM) préclinique, un IRIS® (Inviscan). Cet investissement stratégique vise à renforcer les capacités d’analyse de la plateforme d’imagerie in vivo longitudinal (BIOMAPS / PASREL / Imagerie préclinique et clinique, in vivo, microTEP (SHFJ)) en ouvrant de nouvelles perspectives pour la recherche biomédicale. Le nouvel imageur se distingue par sa capacité à réaliser des acquisitions simultanées sur plusieurs rongeurs, jusqu’à quatre, permettant une augmentation significative du rendement et de la précision des analyses. Cette configuration est idéale pour le suivi longitudinal et la validation de nouveaux radioligands, facilitant ainsi une évaluation détaillée de biomarqueurs clés, notamment ceux liés à la réponse immunitaire. Deux études récentes illustrent les possibilités offertes par cette technologie : (1) L’imagerie de l’infiltration des lymphocytes T CD8+ dans le cadre du cancer du poumon non à petites cellules, dans un modèle orthotopique. Dans le cadre du projet collaboratif IMMUABLE, avec Eric Tartour et Corinne Tanchot de l’HEGP et Hervé Nozach et Bernard Maillère du SIMOS (Institut Joliot/UPSaclay), l’équipe ImmunoMaps de Charles Truillet cherche à prédire l’efficacité des immunothérapies et des problématiques de résistance aux traitements grâce à l’imagerie de certaines populations immunitaires. L’utilisation de l’IRIS® (Inviscan) a permis de démontrer le rôle crucial des lymphocytes T CD8+ dans la réponse antitumorale, ouvrant la voie à de nouvelles stratégies thérapeutiques pour surmonter les résistances aux traitements. (2) Optimisation du passage du témozolomide à travers la barrière hémato-encéphalique. Toujours au sein de BioMaps, la même équipe (ImmunoMaps) avec Anthony Novell (équipe IMATHER) et Fabien Caillé (BIOMAPS / PASREL / Plateforme de développement de radiotraceurs (SHFJ)), a montré par imagerie TEP/TDM du témozolomide une amélioration de la délivrance du médicament par ultrasons thérapeutiques tout en préservant un maximum les régions saines du cerveau. Dans ce contexte, le témozolomide a été radiomarqué (isotopiquement) au carbone-11 (sans affecter donc ses propriétés thérapeutiques). Cette technologie ouverte en plateforme au secteur académique et privé permet d’évaluer le développement de nouvelles thérapies, de dérisquer leur utilisation et d’explorer des biomarqueurs prédictifs pour une meilleure sélection des patients pouvant bénéficier de ces traitements. Cet équipement a été co-financé par l’ITMO Cancer et par IBiSA. -> Contact : Charles Truillet (charles.truillet@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme a déjà publié plusieurs FOCUS PLATEFORME ces dernières années. Les relire ? BIOMAPS / PASREL / Imagerie préclinique et clinique, in vivo, microTEP (SHFJ) : Une expertise et des équipements de pointe pour révolutionner vos recherches en imagerie biomédicale. La plateforme d’imagerie biomédicale préclinique en imagerie TEP du SHFJ met à votre disposition un environnement technologique de pointe pour l’étude des mécanismes biologiques et le suivi des médicaments. En permettant de visualiser la distribution des médicaments dans l’organisme, l’imagerie par Tomographie par Emission de Positons (ou TEP) contribue à réduire les risques liés à leur transfert en clinique. Grâce au ciblage de plusieurs biomarqueurs, elle offre également un outil puissant pour prédire l’efficacité thérapeutique, notamment en évaluant l’impact du système immunitaire sur les traitements. Des équipements de dernière génération pour une imagerie préclinique avancée. La plateforme dispose de trois imageurs précliniques TEP et TEP/TDM (TEP couplé à la tomodensitométrie, aussi appelée TEP/CT) pour une visualisation en profondeur et une quantification dynamique des radioligands après injection chez le petit animal : i) Deux Inveons (Siemens) – Référence en imagerie moléculaire haute résolution, ii) Un IRIS (Inviscan) – Flexibilité et performance pour une exploration avancée. Une approche complémentaire. Des analyses ex vivo de haute précision. En complément de l’imagerie in vivo, la plateforme propose un panel de techniques ex vivo pour une analyse multi-échelle, qu’il s’agisse d’échantillons radioactifs ou non : Autoradiographie, immunohistologie & immunofluorescence – Pour une caractérisation tissulaire précise, Cytométrie en flux (single cell) – Une analyse fine au niveau cellulaire, Hébergement d’animaux et modèles expérimentaux adaptés – Disponibilité de modèles orthotopiques de glioblastomes ou de cancers du poumon, ainsi que de modèles classiques de tumeurs sous-cutanées. Des développements sur mesure peuvent être envisagés selon les besoins scientifiques et thérapeutiques. A propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
May 25, 2025 5:22 PM
|
La plateforme de simulation et réalité virtuelle du laboratoire CIAMS (CIAMS / Plateforme Bernstein) est un environnement de simulation et d'analyse interactive du comportement, conjuguant réalité virtuelle immersive, système de capture du mouvement avancé et serveurs de simulation de pointe. Hébergés au sein du Virtualdata Center, ces serveurs intègrent des modèles computationnels innovants pour étudier les déterminants du comportement de manière immersive et interactive. La plateforme intègre le système de capture du mouvement Qualisys de la salle Bernstein, utilisant des ensembles de marqueurs passifs et actifs, ainsi qu'un équipement de réalité virtuelle complet, incluant oculométrie et capture des expressions faciales. Cette infrastructure permet d'intégrer des modèles computationnels de la cognition, tels que le modèle de Conscience Projective (PCM), et des outils d'Intelligence Artificielle avancés, notamment les Large Language Models (LLM). L’ensemble permet de contrôler des agents artificiels incarnés dans des humains virtuels, avec lesquels les participants peuvent interagir en temps réel dans des environnements immersifs. Cet ensemble technologique de pointe est dédié à l'étude des mécanismes cognitifs et affectifs influençant les comportements, avec un accent particulier sur les interactions sociales et les interactions humain-machine. Il offre des opportunités uniques pour explorer le rôle causal de ces processus dans des contextes variés et immersifs. En savoir plus ? Rudrauf et al., Brain Sciences 2023 ; Sergeant-Perthuis et al., Biological Cybernetics 2025 -> Contact : David Rudrauf (david.rudrauf@universite-paris-saclay.fr) et Jean Jeuvrey (jean.jeuvrey@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme a déjà publié plusieurs FOCUS PLATEFORME ces dernières années. Les relire ? CIAMS / Plateforme Bernstein. Cette plateforme accueille 2 principaux environnements dédiées à des thématiques spécifiques. (i) Le premier environnement est un mur interactif NeoOne de la société NeoXperience. Ce mur immersif et interactif représente une nouvelle génération d'exergames et permet de jouer aux jeux vidéo tout en bougeant, en situation d'opposition et/ou de coopération (jusqu'à 10 joueurs simultanés). Plusieurs environnements (sport, écologie, voyage, aventure...) exigent des joueurs de toucher des cibles statiques ou dynamiques, à la main ou avec un lancer de balles. Il existe également 5 casques de RV permettant de la TERV (thérapie par Exposition à la Réalité Virtuelle) de la société C2Care. Les casques permettent d'exposer à des situations de phobies, d'addiction. Elles permettent aussi de la stimulation cognitive et/ou physique en RV ou en RA (réalité augmentée). (ii) Le second euxième environnement est une plateforme de simulation et analyse interactive du comportement, combinant réalité virtuelle immersive, système de capture du mouvement, et serveur de simulation implémentant des modèles computationnels. Le serveur de simulation est hébergé dans le Virtualdata Center de l’Université Paris-Saclay. La plateforme est aussi associée au système de capture du mouvement Qualisys de la salle Bernstein, des ensembles de marqueurs passifs et actifs, et un kit réalité virtuelle. La plateforme permet d’implémenter des modèles computationnels de la cognition, comme le modèle de Conscience Projective (PCM), et divers outils d’Intelligence Artificielle, comme des Large Language Models (LLM), pour contrôler des agents artificiels, qui sont incarnés dans des humains virtuels, avec lesquels des participant.e.s en réalité virtuelle peuvent interagir en temps réel. L’ensemble est utilisé pour étudier le rôle causal de mécanismes cognitifs et affectifs dans la génération des comportements, avec un intérêt particulier pour les interactions sociales, et plus généralement les interactions humains-machines. A propos du CIAMS. Le CIAMS est un laboratoire multidisciplinaire (SDV et SHS) en sciences du mouvement et facteurs humains. Il contribue notamment au réseau Neurosciences, Mouvement, Handicap des unités de recherche en Sciences de la Vie de l'Université Paris-Saclay. Le CIAMS est un membre fondateur de la Fédération Demenÿ-Vaucanson (FéDeV), une Structure Fédérative de Recherche impliquant une quinzaine d’unités de recherche, dont la mission est de structurer la communauté scientifique de Paris-Saclay dans le domaine des sciences du mouvement.

|
Scooped by
Life Sciences UPSaclay
May 11, 2025 4:50 PM
|
EvryRNA est une plateforme logicielle génopolitaine qui met à disposition de la communauté scientifique des outils pour l’analyse des acides ribonucléiques (ARN) non-codants. Ces molécules ont révélé ces dernières décennies leur rôle majeur dans les mécanismes biologiques et dans diverses maladies dont les cancers. La plateforme EvryRNA, dirigée par le Pr Fariza Tahi, est hébergée au sein du laboratoire IBISC (Informatique, Bio-informatique et Systèmes Complexes - Université d’Évry Paris-Saclay). Elle fait partie des 23 infrastructures et plateformes mutualisées sur le biocluster Genopole. L’équipe Bioinformatique des ARN dirigée par Fariza Tahi a développé 5 nouvelles contributions computationnelles dédiées à l’analyse des ARN qui viennent s’ajouter au panel d’outils interactifs librement accessibles sur la plateforme EvryRNA. Les outils développés permettent ainsi d’identifier les ARN dans les séquences génomiques, de déterminer leur caractère codant ou non-codant, ou encore de prédire leurs conformations dans l’espace, déterminantes pour leur fonction. Les ARN ont en effet la propriété de se replier grâce aux liaisons des acides ribonucléiques qui les constituent. Focus sur les 5 nouvelles contributions. La première, nommée MMnc, est une méthode basée sur de l’apprentissage profond permettant la prédiction et la classification des ARN non-codants. Elle exploite plusieurs sources de données (telles que la séquence, la structure secondaire et l'expression) à l'aide d'une intégration de données multimodale basée sur l'attention. Elle permet par ailleurs une représentation interprétable des résultats de classification grâce notamment aux cartes auto-organisatrices (SOM). MMnc peut être ré-entrainé sur de nouveaux sets de données. MMnc offre la possibilité d’intégrer de nouvelles sources de données pour une meilleure caractérisation des classes d’ARN. La deuxième, appelée « State-of-the-RNArt », est une revue complète des méthodes actuelles de prédiction des structures 3D d’ARN. A travers un benchmark d’une dizaine d’outils existants, les prédictions des différentes méthodes ont été comparées sur des jeux de données actuels. Un outil de visualisation est aussi disponible afin d’avoir un aperçu visuel des différentes prédictions. State-of-the-RNArt offre une visualisation des structures 3D prédites avec leurs comparaisons (alignement) avec les structures de référence ou natives. State-of-the-RNArt propose des comparaisons avec un grand nombre de métriques calculées grâce à RNAdvisor, un autre outil développé par les membres de la plateforme EvryRNA. La troisième, RNAAdvisor2, est une version web serveur de l’outil RNAdvisor pour le calcul intégré des différentes métriques d’évaluation de prédiction de structures 3D d’ARN. Dans cette version, une nouvelle fonction de notation a été ajoutée permettant de comparer entre elles les structures prédites et d’évaluer la qualité structurelle dans l'espace des angles de torsion. RNAAdvisor2 permet aux utilisateurs d'effectuer des évaluations de la qualité des structures d’ARN sans avoir à installer toutes les différentes exigences pour chaque métrique d’évaluation ou fonction de notation. RNAAdvisor2 fournit une visualisation des structures ainsi que de leurs alignements. La quatrième est AlphaFold 3 for RNAs, un travail d’évaluation et de comparaison de la dernière version d’AlphaFold, AlphaFold 3 (https://doi.org/10.1038/s41586-024-07487-w) pour la prédiction des structures 3D des ARN. Une visualisation des structures 3D prédites sur différents jeux de données et avec différentes métriques d’évaluation est fournie, ainsi que leurs alignements (superpositions) avec les structures natives ou de références. Enfin, la cinquième contribution récemment mise en ligne est RNA-TorsionBERT, un outil qui permet la prédiction des angles de torsion d’ARN à partir d’un modèle de langue pré-entraîné. La connaissance des angles de torsion des ARN pour chaque résidu peut aider à reconstruire son repliement global. RNA-TorsionBERT prédit des angles de torsion de la structure 3D à partir de la séquence seule, un atout pour la prédiction de la structure de nouveaux ARN orphelins. Les ARN non-codants ouvrent un large champ scientifique. Les deux dernières décennies ont été le théâtre de la découverte d’une multitude d’ARN non-codants, issus des régions de notre génome qui ne codent pas de protéines et que l’on a longtemps pensé inutiles. Les biologistes connaissaient bien la fonction des ARN ribosomiques et des ARN de transfert pour traduire le message des gènes en protéines. Mais récemment, ils ont révélé le rôle biologique essentiel de nombreux autres ARN (micro-ARN, petits ARN interférents, longs ARN non-codants...). Ces ARN non-codants agissent comme des régulateurs de l’expression des gènes, donc comme des acteurs du développement des organismes, de l’adaptation aux changements environnementaux, etc. Ils interviennent dans les processus biologiques, mais aussi dans les maladies, notamment les cancers et les maladies neuro-dégénératives. Mieux connaître les ARN non-codants pourra notamment contribuer à mieux comprendre ces pathologies, mais aussi à envisager de nouvelles approches thérapeutiques. Une plateforme labellisée et soutenue par Genopole. Genopole soutient l’activité de la plateforme EvryRNA, profitable notamment à son axe stratégique des thérapies innovantes. Plus d’information sur EvryRNA ? cliquer ici -> Contact : Fariza.Tahi@univ-evry.fr / Julien.Picot@genopole.fr Plug In Labs Université Paris-Saclay : cliquer ICI La plateforme a déjà publié plusieurs FOCUS PLATEFORME ces dernières années. Les relire ? GENOPOLE / Plate-forme logicielle de bioinformatique EvryRNA. La plateforme EvryRNA est une plateforme logicielle mettant à disposition de la communauté scientifique différents logiciels dédiés aux ARN non-codants pour la prédiction de leurs structures 2D et 3D, la prédiction des structures de complexes d'ARN, la prédiction et la classification d'ARN non-codants, l'identification et la prédiction à grande échelle dans des séquences génomiques d'ARN non-codants, notamment les petits ARN : microARN, piARN, etc. Tous les logiciels disponibles sur la plateforme EvryRNA ont fait l'objet de publications scientifiques à comité de lecture. A propos de Genopole. Biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à la bioéconomie, Genopole réunit 78 entreprises, 17 laboratoires de recherche académiques, 23 infrastructures, plateformes et plateaux techniques mutualisés, ainsi que des formations universitaires (Université d’Évry Paris-Saclay). Son objectif : favoriser l’émergence et la croissance de sociétés de biotechnologie, le transfert d’innovations vers le secteur industriel, le développement de la recherche et l’enseignement supérieur en sciences de la vie. Genopole est un Groupement d’intérêt public principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon.

|
Scooped by
Life Sciences UPSaclay
April 20, 2025 5:36 PM
|
Au sein du laboratoire CIEL (Cerebral Imaging and Engineering Laboratory) de NeuroSpin (Institut des Sciences du Vivant Frédéric Joliot, Centre CEA Paris-Saclay, Gif-sur-Yvette), le groupe d’Imagerie Moléculaire et Délivrance de Molécules Actives dirigé par Benoît Larrat conduit des programmes de recherche pour démontrer que l'utilisation de scanners IRM (Imagerie par Résonance Magnétique) à haut champ magnétique (7 Tesla et au-delà), combinée au développement de nouveaux agents de contraste fonctionnalisés à très forte relaxivité, permet de réaliser l'imagerie moléculaire de pathologies cérébrales sur des modèles rongeurs (tumeurs, accident vasculaires). Pour étudier des biomarqueurs cérébraux intratissulaires comme les plaques amyloïdes dans la maladie d’Alzheimer, un dispositif de perméabilisation temporaire de la barrière hémato-encéphalique (BHE) par ultrasons guidés sous IRM a été mis au point pour garantir l’accessibilité de l’agent de contraste fonctionnalisé à sa cible. Ce dispositif non invasif de perméabilisation temporaire de la BHE a été développé en partenariat avec la société Image Guided Therapy pour fonctionner sous guidage IRM à l’aide du scanner préclinique 7 T de la plateforme (NEUROSPIN / Imagerie in vivo preclinique, ex vivo et in vitro, IRM 7T), puis est optimisé pour des applications in vivo sur des modèles rongeurs. Ce dispositif permet, grâce à un système de motorisation et un logiciel de pilotage dédiés, de contrôler in situ en temps réel les trajectoires mécaniques et électroniques des ultrasons, et ainsi de définir précisément les lieux de délivrance de molécules thérapeutiques aux tissus cérébraux. Une rétroaction fondée sur un transfert en temps réel des images IRM permet de contrôler les effets biologiques potentiels des ultrasons, et le cas échéant de modifier certains paramètres acoustiques pendant la session d’imagerie. Au cours des dernières années, grâce à différentes collaborations nationales, mais également locales avec d’autres laboratoires de l’Université Paris-Saclay, le groupe de Benoît Larrat a montré que ce dispositif non invasif de perméabilisation temporaire de la BHE permet notamment de délivrer dans le cerveau de souris des anticorps thérapeutiques monoclonaux comme le Cetuximab (Tran et al, J Control Release 2020, 328:304-312, collaboration avec le laboratoire Biomaps d’Orsay), ainsi que des thérapies géniques comme le vecteur AAV9-CMV-GFP (Felix et al, Pharmaceutics 2021, 13(8):1245, collaboration avec le Centre de génétique de Marseille). -> Contact : Alfredo Lopez-Kolkovsky (alfredo.lopezkolkovsky@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI NEUROSPIN / Imagerie in vivo preclinique, ex vivo et in vitro, IRM 7T. NeuroSpin dispose d’un scanner préclinique 7 Tesla de la société Bruker BioSpin (modèle Biospec 70/16), équipé d’une électronique Avance III. L’espace disponible à l’intérieur des gradients (90 mm) permet l’imagerie de petits animaux (souris, rats). En dehors d’un haut champ magnétique, une autre particularité de ce système est de disposer de bobines de gradient de champ magnétique permettant de produire un champ variable de 740 mT/m (approximativement 10 fois plus puissant que ce qu’il est possible de faire avec des scanners cliniques), et ainsi d’accéder à des résolutions spatiales élevées et adaptées pour l’imagerie chez le petit animal. De plus, ce système dispose d’une variété d’antennes radiofréquences dédiées à des applications spécifiques : des antennes surfaciques et volumiques pour le cerveau de rat et pour celui de souris (imagerie et spectroscopie 1H), une antenne volumique double noyau 1H/19F pour le cerveau de rat ou le corps de souris, ainsi que des antennes surfaciques pour la spectroscopie de noyaux X (13C, 31P) dans le cerveau de rongeur. Enfin, le scanner préclinique 7 T est équipé d’un système unique d’ultrasons focalisés, conçu spécialement pour réaliser des tirs ultrasonores dans le cerveau de rongeur de façon non invasive. Ce système original, qui a été co-développé avec la société Image Guided Therapy, est doté de moteurs pour un positionnement du point focal ultrasonore arbitrairement choisi dans le cerveau. Il permet de réaliser des expériences de perméabilisation transitoire de la barrière hémato-encéphalique, d’ablation tissulaire par nécrose thermique ou encore de neuromodulation induite par ultrasons. Les tirs ultrasonores, ainsi que les déplacements du transducteur, sont pilotés depuis un logiciel recevant aussi en temps réel les images IRM en cours d’acquisition. À propos de NeuroSpin. NeuroSpin est une infrastructure de recherche sur le cerveau exploitant des grands instruments d'imagerie. NeuroSpin offre à la communauté scientifique publique et privée la possibilité de faire progresser la connaissance du cerveau, et particulièrement du cerveau humain, en proposant un accès à des méthodologies de pointes en imagerie cérébrale et en neuro-informatique. NeuroSpin développe et met à la disposition de la communauté des instruments uniques, notamment en imagerie très hauts champs et dans le domaine des big data. Cette offre s'inscrit dans le cadre des missions spécifiques de NeuroSpin qui sont : i) analyser les fonctions du cerveau humain, leur développement dans l'enfance, et l'impact de la culture et de l'éducation ; ii) identifier les marqueurs et les mécanismes de maladies neurologiques, psychiatriques et neurodéveloppementales ; iii) comparer le cerveau humain et celui d'autres espèces animales ; développer et tester des méthodes d'imagerie à toutes les échelles d'observation : par résonance magnétique (IRM), par électro- et magnétoencéphalographie (EEG et MEG), et par électrophysiologie massivement parallèle ou l'imagerie photonique et v) développer des logiciels spécialisés dans le traitement et la modélisation des grands jeux de données en neuroimagerie. À propos de l’Institut des sciences du vivant Frédéric Joliot : L’Institut des sciences du vivant Frédéric Joliot (CEA-Joliot) étudie les mécanismes du vivant pour, à la fois, produire des connaissances et répondre à des enjeux sociétaux au cœur de la stratégie du CEA : la santé et la médecine du futur, le numérique et la transition énergétique. Les travaux, fondamentaux ou appliqués, reposent sur des développements méthodologiques et technologiques. Les collaborateurs du CEA-Joliot sont pour moitié impliqués dans des unités mixtes de recherche (UMR), en partenariat avec le CNRS, l'INRAE, l’INRIA, l'Inserm, l’Université Paris-Saclay et l’Université de Paris. Le CEA-Joliot est implanté principalement sur le centre CEA-Paris-Saclay. Des équipes travaillent également à Orsay, Marcoule, Caen, Nice et Bordeaux.

|
Scooped by
Life Sciences UPSaclay
April 6, 2025 5:27 PM
|
La plateforme de Barbeau vient tout récemment d’être récompensée du « Best ICOS Ecosystem Station Award 2025 », prix décerné pour la qualité des métadonnées produites et sa « réactivité ». Cette récompense est décernée tous les ans depuis 2024 dans le domaine « Ecosystème » de l’infrastructure de recherche européenne ICOS (ESFRI et reconnue IR sur la feuille de route nationale des infrastructures de recherche française), lors du MSA (Monitoring System Assembly) qui se tient tous les ans dans un pays européen différent, cette année en Espagne à Madrid. En 2024, ICOS compte un peu plus de 100 stations « Ecosystèmes » dans toute l’Europe, installées sur forêts, prairies, cultures, zones humides, tourbières… Dans le cadre de son engagement dans l’IR, l’équipe de la station est pleinement mobilisée dans le suivi des nombreux instruments et capteurs déployés sur la plateforme qui produit des données 24h/24 et 7j/7. Le monitoring et le renseignement des métadonnées en quasi temps-réel permet de maintenir un haut niveau de qualité des données produites. Ces métadonnées sont très importantes car elles permettent à la fois de renseigner l’état des systèmes de mesures automatiques (en panne, en maintenance, en calibration…) mais également celui des mesures réalisées lors des campagnes sur le terrain (période de mesure, qualité des échantillons, état de la météo…). Les données sont ainsi « flaggées » selon un système de notation afin d’évaluer leur qualité in fine. Atteindre un niveau élevé dans la qualité des mesures et des données produites est essentiel pour une évaluation plus fine de l’évolution de l’écosystème forestier dans le cadre des changements climatiques, une diminution de l’incertitude des modèles de fonctionnement écosystémiques et continentaux et enfin une meilleure calibration/validation des produits d’acquisition satellites. -> Contact : Daniel Berveiller (daniel.berveiller@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Aussi, en janvier 2023, la plateforme publiait son premier FOCUS PLATEFORME ! Le relire ? FOCUS PLATEFORME : La station de recherche forestière de Barbeau-Fontainebleau : un site exceptionnel pour étudier le fonctionnement de la forêt Station de recherche forestière de Barbeau / ICOS (Integrated Carbon Observation System). Les activités de la plateforme de recherche forestière de Barbeau sont centrées sur la compréhension des processus qui gouvernent le fonctionnement de l'arbre (chênes et charmes) et de l'écosystème forestier dans son ensemble. Depuis 2005, un fort investissement est entrepris sur le site, notamment au travers d'équipements de mesure situés en haut d'un pylône de 35 mètres de hauteur et dédiés en partie à la mesure des échanges de dioxyde de carbone (CO2) et de vapeur d'eau entre la forêt et l'atmosphère. La plateforme effectue des observations long-terme de l’écosystème forestier sur plus de 200 variables, dans l’air, sur les arbres et dans le sol, au travers notamment de l’Infrastructure de Recherche européenne ICOS (www.icos-ri.eu), dans laquelle la station est labelisée Classe 1 depuis 2019. La plateforme accueille également de nombreux projets de recherche nationaux et internationaux et effectue de la prestation de service.

|
Scooped by
Life Sciences UPSaclay
March 23, 2025 6:03 PM
|
L'Institut de Biologie Intégrative de la Cellule (I2BC, Gif-sur-Yvette, Institut Joliot, CEA, Saclay) est une unité mixte de recherche associée ) l'Université Paris-Saclay, au CNRS et au CEA, accueillant une soixantaine d’équipes de recherche et hébergeant 15 plateformes technologiques et 2 plateaux techniques, répartis en 6 pôles. Redécouvrez toutes ces plateformes via un FOCUS PLATEFORME dédié ! Aujourd’hui, FOCUS PLATEFORME s’intéresse à un nouvel équipement – le FIDA, pour Flow-Induced-Dispersion-Analysis, récemment acquis chez Fida Biosystems par la plateforme de mesures d'Interactions Macromoléculaires (PIM) (I2BC- plateformes Biologie Structurale). La technologie FIDA utilise la détection de fluorescence des systèmes dispersés en diffusion de Taylor (TDA) dans des capillaires. La mesure des coefficients de diffusion des espèces en solution à partir des taylorgrammes permet de calculer la taille des biomolécules, en déterminant avec précision leur rayon hydrodynamique (Rh). Il est alors possible de caractériser les interactions biomoléculaires en fonction de la taille des complexes formés. Les mesures effectuées à l'aide du FIDA peuvent être réalisées dans des milieux complexes tels que le plasma, le sérum, les lysats cellulaires et les milieux de fermentation. L’appareil dispose de deux détecteurs indépendants pour suivre les molécules en UV (280 nm) lorsqu’il s’agit de détecter la fluorescence intrinsèque des protéines ou en fluorescence (488 nm) pour les protéines marquées. Cette technologie a l’avantage de couvrir une grande gamme de taille de molécules, permettant ainsi de travailler avec des petites molécules ainsi qu’avec des systèmes multivalents sub-nanomètre tels que des liposomes ou capsides de virus. En effet, le FIDA peut mesurer des molécules dont le Rh varie entre 0,5 et 1000 nm. Au sein d’un échantillon, il est possible d’identifier les différentes populations de taille. Les mesures de caractérisation des complexes biomoléculaires montrent l’évolution du Rh en fonction de la concentration croissante d’analyte (partenaire non marqué). A partir de cette courbe, l’affinité KD peut être déduite. Des variations de 5 % de taille du Rh peuvent être détectées lors d’une interaction avec un partenaire non fluorescent, et les très faibles affinités (de l’ordre du pM) peuvent être mise en évidence. Dans l’exemple ci-dessus (Rodolphe Auger, Equipe de Thierry Touzé, I2BC, dpt Microbio), nous avons caractérisé l’interaction d’un peptide avec une protéine membranaire solubilisée en détergent, ce qui était très compliqué avec les techniques complémentaires de la plateforme, et qui est rendu possible avec le FIDA. Additionnellement à ces mesures, les données brutes du FIDA fournissent également des informations supplémentaires essentielles au contrôle-qualité des échantillons, notamment des détails sur le fluorophore libre qui n'a pas réagi, l'agrégation des protéines, l'inadéquation du tampon, les changements de viscosité et l'adhésivité des protéines. Plus récemment, le FIDA a été utilisé avec succès pour caractériser en nombre et en taille les condensats : séparations de phase liquide (LLPS, Stender et al., Nat. Commun. 2021) en fonction de la température. L'instrument est entièrement automatisé pour les flacons ou les plaques de 2 x 96 puits avec un contrôle de la température de bout en bout de 4 à 55°C, contrôlable par incréments de 0,1°C. Chaque test ne nécessite que quelques microlitres d'échantillon. La preuve de concept de l’intérêt de la technique sur les systèmes multi-partenaires ou des capsides de virus a été réalisée avec succès. Plusieurs articles scientifiques sont en cours de révision. Contacts : Magali Noiray (Magali.noiray@i2bc.paris-saclay.fr) ; Magali Aumont (magali.aumont-nicaise@i2bc.paris-saclay.fr); Stéphanie Marsin (stephanie.marsin@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Envie de (re)lire les précédents FOCUS PLATEFORME de PIM ? I2BC / Plateforme de mesures d'Interactions Macromoléculaires (PIM). La Plateforme de mesures d'Interactions Macromoléculaires (PIM) de l'I2BC offre un large éventail d'équipements et d'expertises en biochimie et biophysique pour réaliser des mesures de contrôles qualités ainsi que des analyses fonctionnelles d'échantillons de protéines : microcalorimétrie, ultracentrifugation analytique (UCA), MST, SEC-RALS-LALS, BLI, SPR, switchSENSE. Les prestations offertes par la plateforme concernent : 1) La caractérisation biophysique de protéines ou de complexes biologiques (SEC-RALS-LALS, UCA), 2) L'analyse d'interactions biologiques (ITC, SPR, BLI, switchSENSE, MST), 3) L'analyse de la stabilité des macromolécules et des complexes biologiques pour notamment aider à la formulation (DSC, DSF). Les appareils et prestations sont mis à disposition de l'ensemble des laboratoires de l'Université Paris-Saclay, des autres laboratoires académiques ainsi qu'aux industriels. Pour plus d'informations concernant ces prestations : plateforme-pim@i2bc.paris-saclay.fr. Cette plateforme fait partie du pôle des plateformes de Biologie Structurale de l'I2BC qui comprend : i) les plateformes de Cristallisation, RMN, CryoEM, Mesures d'Interactions Macromoléculaires, la génération et production de protéines artificielles (αRep) et ii) les plateaux techniques d'Expression de protéines solubles ou membranaires en levures, Expression des protéines en cellules d'insectes. A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.

|
Scooped by
Life Sciences UPSaclay
March 9, 2025 5:20 PM
|
La Région Ile-de-France contribue régulièrement, et au travers de différents dispositifs, à renforcer les compétences scientifiques et technologiques de son écosystème. A ce titre, elle réédite cette année son AAP SESAME - SESAME 2025 - AAP visant à donner aux laboratoires de recherche de son territoire les moyens nécessaires pour développer de nouveaux projets et pour mettre en œuvre des dispositifs expérimentaux originaux de recherche (comprenez « équipements »). Comme lors des précédentes éditions, ne sont éligibles à SESAME 2025, que des équipements dont le coût total est compris entre 0,2 M€ et 3 M€ HT ; D’autre part, pour rappel, l’aide prendra la forme d’une subvention dont le taux maximum d’intervention régionale pour l’acquisition de ces équipements s’élève à 66% du montant HT du coût total du projet. Ce taux d’intervention régionale pour les projets d’équipements en sciences humaines et sociales pourra néanmoins atteindre 100% du montant HT du coût total, à condition que 3 laboratoires franciliens distincts ou plus participent au projet. Les critères d'éligibilité et de sélection sont disponibles dans le texte de l'appel à projet SESAME 2025. D’autre part, le dossier de candidature est à compléter en ligne sur mesdemarches.iledefrance.fr, par le porteur du projet directement, après sélection par l’Université Paris-Saclay en ce qui nous concerne. Cette sélection se fera naturellement en prenant en compte, outre les critères de la Région mentionnés dans l'AAP, la plus-value collective pour l'Université Paris-Saclay. Attention également de bien prendre les critères d'exclusion mentionnées dans l’AAP, et si cela s'applique à votre projet, de tenir compte des remarques faites par le jury de la Région en cas de soumission les années précédentes. Ci-dessous, le rappel du calendrier, intégrant les échéances propres à l’Université Paris-Saclay : - le lundi 3 mars 2025: Lancement de l’appel à projets SESAME 2025.
Les porteurs de projets sont invités à prévenir le plus tôt possible leur école graduée/institut (GS/I) de leur intention de préparer un dossier. - le vendredi 18 avril 2025 au plus tard: les porteurs de projet transmettent aux GS/I concernés leur projet dans l'état le plus avancé possible, sans oublier la fiche résumé complétée (fiche UPSaclay) ; Egalement, copie de l’ensemble est envoyé par mail à l'adresse vp.recheche@universite-paris-saclay.fr (copie à frederic.dolle@cea.fr) avec mention de l'institution qui porterait le projet.
- du samedi 19 avril au jeudi 15 mai 2025 : les GS/I évaluent les dossiers qui leur sont remontés et produisent un avis (classement en trois catégories A, B, C) et le cas échéant les priorisent au sein d'une catégorie s'il y a beaucoup de projets. Le retour des évaluations des GS/I au CoDiReV est attendu pour le mardi 20 mai 2025 au plus tard.
Aussi, comme les années précédentes, une journée d’audition des porteurs des projets rattachés au domaine Sciences de la Vie de l’Université Paris-Saclay et à ses interfaces est programmée ; la date pressentie est le mercredi 14 mai 2025 (à confirmer). - le mercredi 28 mai 2025, le CoDiRev se réunira, sélectionnera les huit projets que l'UPSaclay est autorisée à faire remonter dans le cadre de cet AAP, et fera parvenir aux porteurs de projets la lettre de soutien de l’Université Paris-Saclay.
- les porteurs de projets auront ensuite jusqu’au mardi 3 juin 2025 (17h00) – date/heure limite de dépôt – pour finaliser leur dossier et le déposer auprès de la Région IdF.

|
Scooped by
Life Sciences UPSaclay
February 14, 2025 12:45 PM
|
Les appels d’offres IBiSA 2025 sont ouverts. IBiSA vient tout juste de lancer les éditions 2025 de l’appel d’offres Plateformes et de l’appel à projets CRB. Ils sont respectivement ouverts jusqu’au 7 et 14 avril 2025, avec une date limite fixée au 3 et 17 mars 2025 pour les présélections. A vos dossiers ! En savoir plus. Résultats de l’appel d’offres Plateformes 2024. Pour l'édition 2024 de son appel lancé aux Plateformes, le GIS IBiSA a reçu 70 candidatures dans tous les domaines des sciences du vivant. Après 67 visites, il a décerné 22 labels et distribué 2 287 000 € de financements à 48 plateformes technologiques en biologie, santé et agronomie. Lire l’article. Du cerveau au podium, découvrez la plateforme PAM. Les jeux de Paris 2024 ont été le théâtre de nombreux records olympiques et personnels. Pourquoi s'arrêter là ? Pour la préparation mentale de ses athlètes, le sport français peut s'appuyer sur les atouts de la recherche et des structures de pointe comme la Plateforme d'analyse du mouvement (PAM), spécialiste de la motricité humaine. Lire l’article. Vous souhaitez recevoir la newsletter publiée par IBiSA chaque trimestre ? Inscrivez-vous ! A propos d’IBISA. Le GIS IBiSA coordonne la politique nationale de labellisation et de soutien aux infrastructures de biologie, santé et agronomie. Placé sous la tutelle de 8 partenaires, il est l’unique instrument de financement commun à l’ensemble des établissements de recherche en sciences du vivant. Grâce à deux appels d’offres dédiés, les plateformes et centres de ressources biologiques (CRB) peuvent candidater à la labellisation IBiSA et accéder à des financements conséquents pour des investissements jugés nécessaires à leurs missions. Le GIS conditionne son soutien à une ouverture large à la communauté scientifique. Il encourage également la création de structures de pilotage, concertation et coopération, l'animation de réseaux thématiques et les démarches qualité. Plus d'infos sur le GIS IBiSA. Vous souhaitez découvrir le potentiel de Paris-Saclay en termes de plateformes ? L’interface Plug In Labs Université Paris-Saclay recense et rend visible plus de 200 plateformes dans le domaine des sciences de la vie - des plateaux techniques, des plateformes technologiques, des infrastructures d’expérimentation, mais aussi des collections - en d’autres termes, des espaces de laboratoires dotés d’équipements, souvent uniques, ou de banques de ressources, associés à un fort potentiel humain, les opérant et les maintenant au meilleur niveau technologique. A propos de Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
February 2, 2025 12:06 PM
|
La plateforme de microscopie MIPSIT fait partie des 10 plateformes technologiques de l’IPSIT (Ingénierie et Plateformes au Service de l'Innovation Thérapeutique), située dans le bâtiment Recherche Henri Moissan au pôle Biologie-Pharmacie-Chimie (plateau de Moulon, Orsay). Au service de la communauté scientifique, MIPSIT accueillera fin 2025, le premier microscope de fluorescence commercial de l’Université Paris-Saclay, associant imagerie super-résolue et optique adaptative, qui confère à l’instrument la capacité de maintenir une résolution spatiale de quelques dizaines de nanomètres même à une centaine de micromètres de profondeur dans des tissus. C’est le besoin d’un dispositif d’imagerie optique à super-résolution adapté aux échantillons épais, exprimé par un grand nombre d’équipes (plus de 35) et de plateformes des UFR de Pharmacie, Sciences et Médecine de l’Université Paris-Saclay et le constat qu’aucun instrument de ce type n’était disponible sur le périmètre de l’Université, qui ont motivé le projet OASIS « Optique Adaptative et Super-résolution pour l’Innovation en Santé ». Ce projet a pour ambition de lever les limites technologiques actuelles d’imagerie de l’organisation de complexes moléculaires dans leur contexte non seulement cellulaire mais aussi tissulaire, sur échantillons fixés ou vivant. Porté conjointement par la plateforme MIPSIT, l’équipe Biophotonique, Neurosciences et Optométrie du LuMIn (Laboratoire Lumière, Matière et Interfaces) à l’ENS Paris-Saclay et l’UAR-MIC (Multimodal Imaging Center) à l’Institut Curie d’Orsay, OASIS est lauréat de l’appel à projet régional SESAME 2024. L’acquisition de cet instrument, d’un budget total de 810 k€, sera donc réalisée grâce à la subvention de la région Île-de-France de 457,5 k€ (56,5% du montant de l’équipement souhaité) et à plusieurs co-financements dont ceux obtenus auprès de l’ERM 2025 de l’UPSaclay, l’INSERM, l’ENS-Paris-Saclay, l’Institut Curie et des ressources propres des unités porteuses ou partenaires. L’équipement sera un microscope confocal à balayage de pointe, réalisant l’imagerie de super-résolution de type STimulated Emission Depletion, et doté d’un module d’optique adaptative (OA). L’intégration de cette technologie, développée à l’origine pour l’imagerie en astronomie, permet de corriger des aberrations dues aux inhomogénéités d’indice de réfraction afin de maintenir une résolution spatiale élevée à ≈100 µm de profondeur dans des tissus. La meilleure résolution atteignable est de 25 nm en STED 2D et de 65 nm isotrope en STED 3D. Ces performances résultent de l’intégration de plusieurs avancées technologiques en plus de l’OA, telles que l’illumination adaptative, qui dose de façon dynamique la puissance du laser appliquée en fonction des signaux fluorescents dans les structures d’intérêt, permettant de limiter très largement la photo-dégradation et rendant ainsi possible l’imagerie d’échantillons vivants en STED. L’instrument possèdera aussi des détecteurs matriciels qui améliorent le rapport signal sur fond et la résolution même en mode confocal conventionnel. Grâce à 6 longueurs d’onde de lasers d’excitation différentes en mode impulsionnel, il pourra détecter 5 fluorophores dans un même échantillon en STED (6 en confocal), et permettra également la séparation spectrale et l'imagerie de durée de vie de fluorescence, offrant ainsi une polyvalence inégalée sur le périmètre. -> Contact : Séverine Domenichini (severine.domenichini@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI En savoir plus sur l’équipe Biophotonique, Neurosciences et Optométrie (LuMIn) ? En savoir plus sur le Multimodal Imaging Center (CURIE / MIC) ? Aussi, en 2020, la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? FOCUS PLATEFORME : La microscopie photonique Super-Résolue : Une nouvelle dimension au service de l'innovation thérapeutique IPSIT / Plateforme d'imagerie cellulaire (MIPSIT). La plateforme d’imagerie cellulaire MIPSIT fait partie de l’unité mixte de service UMS-IPSIT de la Faculté de Pharmacie de l’Université Paris-Saclay. Depuis l’été 2022, notre unité est située sur le plateau du Moulon à Orsay, dans le bâtiment Henri Moissan qui regroupe des unités recherche de Biologie, Pharmacie et Chimie de l’Université Paris-Saclay. Cette plateforme, ouverte aux laboratoires de recherche académiques et privés, a pour principale mission de proposer une expertise et un accès à des outils technologiques performants dans le domaine de la microscopie photonique et l’analyse d’images. La visualisation de la localisation de molécules d’intérêt ou le suivi de processus dynamiques dans les trois dimensions de l’espace au niveau tissulaire, cellulaire ou subcellulaire est une approche méthodologique supplémentaire dans la compréhension des pathologies humaines et animales. A propos d’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 11 plateformes techniques, organisées en trois pôles technologiques (IMCELLF, OMICS et INTERACTIONS) et trois plateformes transverses. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
January 19, 2025 10:13 AM
|
Les résultats sont tombés en fin d’année 2024 : ce sont quatre projets d’équipements du domaine Sciences de la Vie & Santé de l’Université Paris-Saclay qui sont lauréats de l’AAP SESAME 2024, et qui à ce titre seront subventionnés à hauteur de 66% de leur demande. L’Université Paris-Saclay est aussi lauréate de deux autres projets cofinancés dans le cadre de ce dispositif, relevant du domaine Sciences & Ingénierie. Vous avez dit SESAME ? Le dispositif SESAME (pour « Soutien aux Équipes Scientifiques pour l’Acquisition de Moyens Expérimentaux ») vise à cofinancer des équipements scientifiques nécessaires au fonctionnement des laboratoires de recherche publics franciliens pour mener des projets d’envergure dans des champs thématiques autres que les Domaines de recherche et d’Innovation Majeurs (DIM) et les Questions d’Intérêt Majeur (QIM), et hors du périmètre d’intervention de Genopole et des projets inscrits au Contrat de Plan État-Région 2021-2027. SESAME 2024 : Demande de subventions versus montants alloués par la région ? Sur les 34 projets de recherche éligibles et évalués par la Région Ile-de-France (représentant un total de 28,38 millions d’euros HT de demandes de subvention), seuls 12 ont été sélectionnés via SESAME 2024, et ceci pour un montant de financement HT de 6 013 812 €. Ces 12 projets recouvrent différentes thématiques scientifiques, dont 6 en biologie et santé, 2 en chimie et biochimie, 1 en physique, matériaux et procédés, 2 en énergie, environnement et agro-sciences, mais également 1 en sciences humaines et sociales. SESAME 2024 : Comment se positionne l’Université Paris-Saclay ? Sur ces 12 projets, 6 sont portés par l’Université Paris-Saclay, soit la moitié ! Deux projets relèvent du domaine Sciences & Ingénierie : HREELM (microscopie à perte d’énergie des électrons à haute résolution, ISMO (1 090 000 €) et CLIO (mise à niveau de la plateforme de spectroscopie IRMPD, ICP, 895 000 €), et quatre relèvent du domaine Sciences de la Vie & Santé : CONNECTION, OASIS, PROTEHAD et SINGLEMOL@UPSACLAY. Ci-dessous les premières informations concernant les quatre projets Sciences de la Vie & Santé, projets que vous découvrirez par ailleurs plus en détails dans les prochaines semaines via des FOCUS PLATEFORME dédiés. - CONNEXION | 500 000 € (IJPB / OV-CM) - Nouveaux outils d’analyse de mélanges complexes par spectrométrie de masse pour la bio-économie.
- OASIS| 457 500 € (IPSIT / MIPSIT) - Optique adaptative et super-résolution pour l'innovation en santé.
- PROTEHAD| 514 000 € (GQE-Le Moulon / PAPPSO) - Protéomique à haut-débit pour l'agriculture de demain.
- SINGLEMOL@UPSACLAY| 429 000 € (I2BC / PIM) - Technologie de rupture de mesures à l'échelle de la molécule unique pour la caractérisation des mécanismes moléculaires et cellulaires fondamentaux du vivant liés à des enjeux sociétaux de santé.
A suivre donc ! -> Contact : Frédéric Dollé (frederic.dolle@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ICI Plug In Labs Université Paris-Saclay. Plug In Labs Université Paris-Saclay ou PILUPS pour les intimes, est le portail numérique unique retenu par l’Université Paris-Saclay pour la mise en valeur et promotions des compétences, expertises et technologies des laboratoires et plateformes technologiques de son territoire. Piloté par l’Université Paris-Saclay et la SATT Paris-Saclay, financé par l’IDEX et le Fonds national de valorisation, PILUPS est accessible à tous depuis 2017, partenaires académiques comme entreprises, en particulier les PME. Un seul site web : https://www.pluginlabs-universiteparissaclay.fr. Et une seule adresse mail : pluginlabs@universite-paris-saclay.fr.

|
Scooped by
Life Sciences UPSaclay
January 4, 2025 12:40 PM
|
Vous êtes chercheurs, étudiants, ingénieurs ou techniciens ? Vous travaillez dans le public ou le privé ? Vous avez besoin d’un accompagnement scientifique, technique et pluridisciplinaire dans les processus de découverte d’un médicament ? Alors, venez découvrir les plateformes technologiques dédiées à l’innovation thérapeutique de la Faculté de Pharmacie de l’Université Paris-Saclay, l’Unité Mixte de Services IPSIT, mardi 21 janvier de 13 H à 16 H. S’incrire : https://www.eventbrite.fr/e/billets-a-la-decouverte-des-plateformes-technologiques-de-lipsit-1040593552327 Le format retenu est résolument court, 1 heure de visite, sur inscription, suivie d’échanges autour d’une dégustation, afin de donner une vision globale de l’unité mixte de services et de rencontrer son personnel. Résolument à l’interface de la chimie, de la biologie et de la clinique, l’IPSIT établit des ponts entre la cible pathologique et le médicament. Le processus de découverte d’un médicament requiert une approche multidisciplinaire associant des savoir-faire complémentaires, notamment dans les domaines de la chimie, de la galénique, de la biologie structurale, de la biologie moléculaire et cellulaire, sans oublier la génétique et la pharmacologie. L’IPSIT, par ses plateformes technologiques, apporte aux équipes une aide scientifique essentielle dans ce processus. L’IPSIT participe aussi à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Découvrez ainsi : - le pôle OMICS avec ses plateformes de transcriptomique et génomique (ACTAGen), protéomique (PROTEOMIC), spectrométrie de masse et lipidomique (SAMM)
- le pôle Imagerie Cellulaire avec ses plateformes de microscopie photonique super-résolue et d’analyse d’images (MIPSIT), de cytométrie en flux (CYM) et d’histopathologie (PHIC)
- le pôle Interaction avec la plateforme de criblage moléculaire (CIBLOT)
- Ainsi que des services transverses constituées par une animalerie ultra-moderne équipée de dispositifs d’exploration fonctionnelle (ANIMEX), une verrerie scientifique et technique (VERRE SCIEN-TECH) et un plateau de bio-informatique (BIOINFO).
-> Contact : Valerie Domergue (valerie.domergue@universite-paris-saclay.fr), Directrice de l’IPSIT A propos de l’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes techniques, organisées en trois pôles technologiques (IMCELLF, OMICS et INTERACTIONS) et trois plateformes transverses. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
December 8, 2024 4:55 PM
|
Retour sur deux publications récentes issues du laboratoire Structure et Activité des Biomolécules Normales et Pathologiques (SABNP, Inserm U 1204 – Université d’Évry-Val-d’Essonne - Université Paris-Saclay) qui héberge la plateforme de biologie structurale de Genopole et qui, grâce à cette dernière, élucide des mécanismes clés associés à des cancers et des maladies neurodégénératives. Pour rappeler brièvement le contexte, FUS et TDP-43 sont deux protéines de liaison à l'ARNm hébergeant des domaines enclins à s’auto-assembler et dont les mutations pathologiques sont liées à la progression de maladies neurodégénératives. Alors que FUS et TDP-43 forment des compartiments réversibles riches en ARNm dans le noyau, les mutations pathologiques favorisent leur agrégation cytoplasmique respective dans les neurones indépendamment l’une de l’autre. En combinant des analyses dans un contexte in cellulo et in vitro à haute résolution par microscopie à force atomique, les chercheurs du laboratoire SABNP ont découvert que la protéine TDP-43 est spécifiquement recrutée dans les assemblages de FUS pour former des sous-compartiments riches en TDP-43, mais sans réciprocité (Demongin C et al., J Biol Chem 2024). La présence d'ARNm permet d’augmenter la proportion de TDP-43 miscible dans les compartiments de FUS. En conséquence, les auteurs ont également constaté que la forme pathologique tronquée de TDP-43, TDP-25, dont la capacité de liaison à l’ARN est altérée, ne se mélange plus avec FUS. Dans leur ensemble, ces résultats montrent que la liaison de FUS le long des ARNm naissants permet à TDP-43, qui est très sujet à l'agrégation, de s’incorporer dans la phase riche en FUS pour former des sous-compartiments riches en ARNm. Ce lien fonctionnel entre FUS et TDP-43 pourrait expliquer leur implication commune dans la sclérose latérale amyotrophique. Par ailleurs, toujours au sein de la plateforme de biologie structurale de Genopole, des analyses réalisées par spectroscopie RMN ont permis de révéler le rôle unique de FUS dans l’activation de PARP-1 (Mamontova et al., Cell Reports 2023). L'activation de PARP-1 suite à des dommages à l'ADN conduit à la synthèse de longues chaînes de poly(ADP-ribose) (PAR), qui sert de signal pour la réparation de l'ADN. Par RMN, les auteurs montrent que FUS, une protéine de liaison à l'ARN, est spécifiquement dirigée vers PAR via son motif de reconnaissance à l’ARN pour augmenter la synthèse de PAR dans les cellules HeLa après un stress génotoxique. Sur la base des résultats de ce travail, les auteurs proposent un modèle dans lequel, suite à un arrêt transcriptionnel qui libère FUS de l'ARNm naissant, FUS peut être recrutée par PARP-1 pour stimuler la synthèse de PAR. Ce modèle offrira sans nul doute de nouvelles perspectives pour comprendre le rôle des protéines FET dans les cancers et dans certaines maladies neurodégénératives dans lesquelles FUS est impliquée. Pour connaitre les modalités d’accès à la plateforme de biologie structurale, n’hésitez pas, contactez-nous ! Vous souhaitez relire leurs précédents FOCUS PLATEFORME ? FOCUS PLATEFORME : La haute résolution arrive sur la plateforme de biologie structurale de Genopole ! (13-sept.-21) ; FOCUS PLATEFORME : Plateforme de biologie structurale (Genopole) ou la combinaison d'une expertise propriétaire avec l'utilisation d'un équipement de pointe ! (14-mars-22) -> Contact : Julien Picot (Julien.Picot@genopole.fr) Plug In Labs Université Paris-Saclay : cliquer ICI GENOPOLE / Plateforme de biologie structurale. La plateforme de biologie structurale offre des équipements de pointe et l’expertise associée en spectroscopie RMN et en microscopie à force atomique. Concernant la spectroscopie RMN, les domaines d’activités sont d’une part l’analyse de la structure de protéines en solution, leur repliement, leur stabilité et leur dynamique en solution, d’autre part l’analyse des interactions ligand-protéine, protéine-protéine et protéine-acides nucléiques. Concernant la microscopie à force atomique, les domaines d’activités sont la caractérisation d’objets biologiques à l’échelle nanométrique, l’observation à l’air ou en milieu liquide de molécules uniques (ADN ou protéines), et l’étude de la formation de complexes ADN-ligands, protéine-protéine et microtubules-partenaires. Enfin, une partie de l’activité de la plateforme concerne la modélisation de la dynamique moléculaire. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 66 entreprises de biotechnologies, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : soutenir les politiques nationales de réindustrialisation et de souveraineté sanitaire, créer et soutenir les entreprises de biotechnologies et le transfert de technologies vers le secteur industriel (notamment pour décarboner la production), favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).
|



 Your new post is loading...
Your new post is loading...