Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
March 7, 2020 6:25 PM
|
Le processus de la découverte d’un médicament requiert une approche multidisciplinaire associant des savoir-faire complémentaires, notamment dans les domaines de la chimie, pharmacotechnie, immunologie, biologie structurale, biologie moléculaire et cellulaire, sans oublier la génétique et la pharmacologie. Parmi les technologies dont disposent les unités de recherche de l’IPSIT (Ingénierie et Plateformes au Service de l'Innovation Thérapeutique), il y a l’imagerie photonique. Celle-ci occupe une place stratégique depuis l’acquisition sur la plate-forme d’imagerie cellulaire MIPSIT, d’un microscope STED (STimulated Emission Depletion) dit « super-résolu ». Cette technique purement optique aux deux prix Nobels (A. Einstein 1921 et Stephan Hell 2014), permet d’accéder à des dimensions de l’ordre de quelques dizaines de nanomètre (> 50 nm), soit quatre fois plus résolue que la microscopie confocale à balayage laser. Elle ouvre de nouvelles perspectives dans le cadre du développement des nanomédicaments, notamment la caractérisation de leur structure qu’elle soit nanoparticulaire et/ou microparticulaire. Exemple choisi : Dans le domaine de la galénique, l’acheminement d’un principe actif au-delà des barrières biologiques tout en garantissant son intégrité puis son relargage contrôlé sur sa cible biologique est un défi majeur. Il s’agit donc de concevoir de très petites entités à base de lipides ou de polymères biocompatibles dans lesquelles les médicaments sont encapsulés. Notre toute première étude STED visait à tester les limites aérodynamiques de poudres sèches microparticulaires (NEM, Nano-Embedded Microparticles) en vue de leur administration par inhalation. Notre travail a consisté à visualiser ces édifices et leurs sous-unités associées entre elles (voir illustration) grâce à un sucre, le tréhalose, puis à évaluer in vitro leur comportement à la désintégration après une aérosolisation sur une couche de mucus. (Christian A. Ruge et al., Colloids and Surfaces B: Biointerfaces, 2016). D’autres domaines ont recours à la super-résolution (Ramet L. et al., J. Neuroscience, 2017 ; Arthur T. Molines et al. Biology Open, 2018; Frauke Beilstein et al. PLoS Pathog 15: e1008209). En neurobiologie, par exemple, nous avons pu localiser et/ou colocaliser les différents transporteurs vésiculaires du glutamate dans les varicosités du cerveau de souris au niveau du striatum. En physiologie végétale, la microscopie super-résolue STED a permis de mettre en évidence la désorganisation du réseau de microtubules corticaux chez les mutants d’EB1 (End Binding one) au niveau de la racine d’Arabidopsis Thaliana et enfin, en virologie, d’explorer l’organisation dynamique des différentes glycoprotéines sur l'enveloppe virale de HSV-1 (virus de l’herpès simplex 1) participant à l'optimisation du processus de liaison cellulaire et de fusion membranaire lors de l’infection. Le STED vient ainsi compléter les modalités d’acquisition multi-échelles, multi-couleurs proposées par la plate-forme MIPSIT de la faculté de pharmacie à Châtenay-Malabry. La plate-forme technique et de recherche en biologie cellulaire et imagerie de l’UMS-IPSIT (MIPSIT) est installée depuis novembre 2000 sur le site de la Faculté de Pharmacie de l’Université Paris-Sud. Cette plate-forme, ouverte à toute équipe de recherche, a pour principale mission de proposer une expertise et un accès à des outils technologiques performants dans le domaine de la microscopie photonique et l’analyse d’images. La visualisation de la localisation de molécules d’intérêt ou le suivi de processus dynamiques dans les trois dimensions de l’espace au niveau tissulaire, cellulaire ou subcellulaire est une approche méthodologique supplémentaire dans la compréhension des pathologies humaines. Elle est membre du Réseau d’Imagerie Cellulaire de Paris-Saclay (RIC). Contacts : Valérie Nicolas (valerie.nicolas@universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ICI

|
Scooped by
Life Sciences UPSaclay
February 23, 2020 11:01 AM
|
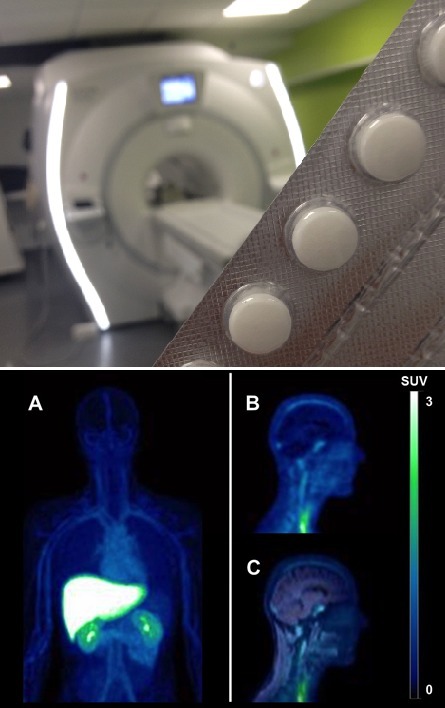
Les chercheurs du Service Hospitalier Frédéric Joliot (CEA/DRF/Institut des Sciences du Vivant Frédéric Joliot, CEA, Orsay) développent des approches d’imagerie permettant d’étudier les effets et la cinétique des médicaments dans l’ensemble des organes du corps humain. Dans ce cadre, la tomographie par émission de positons (TEP) trouve une place prépondérante dans le développement de candidats médicaments. Ces approches d’imagerie pharmacologique reposent principalement sur le suivi de l’expression de biomarqueurs liés à la pathologie dans les tissus cibles. Ainsi, l’utilisation de radioligands spécifiques de cibles pharmacologiques permet de déterminer, de façon indirecte, le taux d’occupation des cibles associé à une dose de médicament donnée. Aujourd’hui, grâce aux progrès de la radiochimie des émetteurs de positons, la production d’analogues chimiques stricts de nombreux médicaments (marquage isotopique) permet d’envisager de nouvelles approches d’imagerie pharmacologique par TEP et d’étudier la cinétique des médicaments au niveau des tissus cibles. Dans cette optique, l’isotope de choix est le carbone-11 (demi-vie : 20,4 minutes) mais aussi parfois le fluor-18 (demi-vie : 109,8 minutes), lorsque le principe actif présente un atome de fluor dans sa structure (voir aussi SHFJ/Radiochimie). La sensibilité de la TEP permet aussi de faire le choix d’administrer des microdoses - quelques microgrammes seulement - ou des doses pharmacologiques du principe actif. De plus, depuis 2015 le SHFJ dispose d’un appareil hybride (illustration haute), couplant imagerie TEP et imagerie par résonnance magnétique (IRM) particulièrement adapté à l’imagerie pharmacologique (SHFJ/Imagerie préclinique et clinique, in vivo, TEP/IRM). Par rapport au TEP-CT (tomodensitométrie ou scanner X) conventionnel, également disponible au SHFJ (SHFJ/Imagerie clinique, in vivo, TEP/CT), la TEP-IRM permet de limiter l’exposition des sujets aux rayonnements ionisants (pas de contributions de doses de rayons X). Il est également possible d’acquérir simultanément des données structurelles et, dans certains cas, des informations fonctionnelles relatives aux effets du médicament dans les tissus (IRM pharmacologique). Le travail des physiciens médicaux sur l’optimisation des conditions d’acquisition et de reconstruction permet de générer des images dynamiques corps-entier (4D) qui décrivent la cinétique des molécules marquées dans les différents organes. Depuis quelques années, cette approche d’imagerie multimodale et pharmacologique a été mise à profit pour déterminer l’exposition tissulaire de molécules radiomarquées dans le cadre de protocoles cliniques, notamment celle du glyburide marqué au carbone-11 (11C-glyburide, protocole IsotoPK (Promoteur CEA, ANR-16-CE17-0011-01)). Ce protocole, comme d’autres menés actuellement au SHFJ avec la 11C-buprénorphine ou le 11C-erlotinib par exemple, visent à déterminer l’importance de paramètres pharmacocinétiques sur l’exposition tissulaire et les effets locaux des médicaments chez l’Homme. Exemple choisi avec le 11C-glyburide (illustration basse) qui dévoile sa biodistribution dite corps-entier chez l’Homme (A, imagerie TEP) mais aussi, avec précision, sa distribution dans les différentes régions du cerveau (B, imagerie TEP seule et C, imagerie duale TEP et IRM) (Marie et al., Neurology 2019). La plateforme SHFJ/Imagerie préclinique et clinique, in vivo, TEP/IRM permet de réaliser des acquisitions simultanées TEP et IRM (3 T) cliniques et précliniques, corps entier, synchronisées ou non à la respiration, statiques ou dynamiques. L’imagerie TEP se fait au moyen de radiotraceurs extérieurs ou produits sur site (SHFJ/Radiochimie). Les compétences sont disponibles pour l’analyse et l'interprétation des données pharmacocinétiques en regard de données d’imagerie multimodale acquises au SHFJ ou dans d'autres centres. Contact : nicolas.tournier@cea.fr Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
February 9, 2020 12:33 PM
|
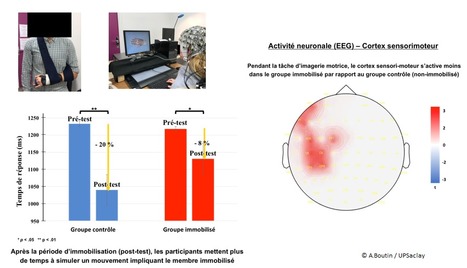
La plateforme d'imagerie du cerveau humain à très haute résolution temporelle par électroencéphalographie (CIAMS/EEG) située à l’UFR STAPS, est utilisée pour des projets de recherche s’inscrivant dans le domaine des neurosciences cognitives et comportementales. Rattachée à l’équipe ‘’Mouvement Humain, Adaptation et Performance Sportive’’ (MHAPS), elle est aussi l’une des cinq plateformes que compte à ce jour le CIAMS (Laboratoire Complexité, Innovation, Activités Motrices et Sportives). Deux champs d’investigations scientifiques sont particulièrement portés par MHAPS et notre plateforme : Un premier, qui a pour objectif d’identifier les déterminants d’une interaction naturelle avec un personnage virtuel (avatar) exprimant des émotions, et un deuxième, visant à étudier les bases neurofonctionnelles du mouvement humain et de la plasticité sensorimotrice. Retour sur une utilisation récente de la plateforme CIAMS/EEG. Si 12 heures d’immobilisation du membre supérieur suffisent pour induire une réorganisation du cortex sensorimoteur et perturber la coordination et la précision d’un mouvement de pointage (Huber et al., 2006, Nat Neurosci), d’autres travaux ont spécifiquement démontré l’impact d’une immobilisation de courte durée (24h-48h) sur la capacité à se représenter mentalement un mouvement impliquant le membre immobilisé (e.g., Toussaint & Meugnot, 2013, J Exp Psychol Learn Mem Cogn). Ces résultats sont par ailleurs en accord avec le postulat d’une équivalence fonctionnelle entre la pratique physique et l’imagerie motrice (IM), tant sur le plan des processus cognitifs engagés que de l’activité cérébrale (Jeannerod, 2001, NeuroImage ; Hardwick et al., 2018, Neurosci Biobehav Rev). Plus récemment, notre plateforme a contribué à une nouvelle étude originale dans ce contexte (Meugnot, Cagnard & Boutin, en préparation). L’originalité de notre étude a été d’utiliser une tâche d’IM, consistant à s’imaginer faire un mouvement sans l’exécuter physiquement, afin d’évaluer l’effet d’une restriction sensorimotrice (i.e., immobilisation du membre) sur les réorganisations corticales au niveau du cortex sensorimoteur controlatéral au membre immobilisé. Nos résultats indiquent qu’une période d’inactivité de 48 h du membre supérieur suffit pour détériorer la performance dans une tâche d’IM qui requiert la simulation du membre immobilisé. Cette baisse de performance est associée à une réduction de l’activité cérébrale (enregistrée au moyen de l’EEG) au niveau du cortex sensorimoteur, et spécifiquement sur l’hémisphère cérébral contrôlant le membre immobilisé. La plateforme CIAMS / EEG est utilisée à des fins de recherche fondamentale et appliquée en neurosciences cognitives et comportementales chez l'homme adulte sain. L'enregistrement du signal EEG peut également être couplé à des mesures électrophysiologiques (e.g. EMG, activité électrodermale) pour des enregistrements simultanés et complémentaires. Contacts : Aurore Meugnot (aurore.meugnot@u-psud.fr) ; Arnaud Boutin (arnaud.boutin@u-psud.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
January 26, 2020 1:12 PM
|
C’est au Service Hospitalier Frédéric Joliot (SHFJ) du CEA à Orsay (DRF/Institut des Sciences du Vivant Frédéric Joliot), dans l’UMR d’Imagerie Biomédicale Multimodale Paris-Saclay BIOMAPS, que la magie opère. Là-bas, à l’abri des regards, la plateforme de radiomarquage (SHFJ/Radiochimie) met en œuvre une étrange alchimie : elle transforme un simple atome radioactif en un outil diagnostique puissant pour l’imagerie tomographique à émission de positons (TEP). Que ce soit avec du carbone-11 (demi-vie 20,4 min) ou du fluor-18 (demi-vie 109,8 min), produits sur place avec un cyclotron médical pour radiomarquer de façon covalente de petites molécules, ou avec du zirconium-89 (demi-vie 78,4 heures) complexé avec des « biologics » (protéines, anticorps), une myriade de radiotraceurs est mise à la disposition de la science. Oncologues, neurologues, pharmaciens, biologistes se précipitent pour étancher leur soif de recherche, qu’elle soit biomédicale (chez l’Homme) ou préclinique (sur petits et gros animaux). Marquage isotopique de médicaments, traceurs de la neuroinflammation, des agrégats protéiques ou des récepteurs neuronaux, métabolisme et hypoxie tumorale, imagerie de l’infection… Voilà une liste non exhaustive des prouesses réalisées par la plateforme de radiochimie du SHFJ, à votre service. Equipées de 4 automates de synthèses, d’un laboratoire de contrôle qualité et d’une radiopharmacie toujours de garde, les petites fées de la plateforme sont parées pour exhausser vos souhaits. D’autant que la famille s’agrandit. En 2020, un nouveau laboratoire de production de radiopharmaceutiques en salle blanche verra le jour, doublant la capacité de production actuelle. De quoi ravir toute demande en radiotraceurs et radiopharmaceutiques originaux pour faire de l’imagerie TEP, l’outil diagnostique et pronostique par excellence. Vous êtes intéressés mais vos activités semblent trop éloignées d’Orsay ? Pas de panique ! Grâce à notre service de livraison Express, nous livrons à domicile ces produits de qualité pharmaceutique en un temps record. La plateforme de Radiochimie du SHFJ (SHFJ / Radiochimie) a pour vocation de synthétiser des radiotraceurs pour la recherche biomédicale, en particulier pour l’imagerie par tomographie d’émission de positons, à partir d’émetteurs de positons produits sur site (Carbone 11, Oxygène 15, Fluor 18) ou importés (par exemple Zirconium 89). Contact : fabien.caille@cea.fr

|
Scooped by
Life Sciences UPSaclay
January 11, 2020 4:49 PM
|
La plateforme Chimie-Métabolisme-Métabolome de l’Observatoire du Végétal de l’Institut Jean-Pierre Bourgin (IJPB), à l’INRAE de Versailles, propose l’analyse de petites molécules aux différentes équipes de recherche en biologie des plantes de Paris-Saclay et au-delà. Dotée d’une solide expérience en métabolomique des plantes par GC-MS, elle met aussi en place des développements méthodologiques pour l’analyse métabolomique non ciblée par LC-MS/MS haute résolution. Exemple choisi : Entamée dans le cadre d’un projet collaboratif d’étude de l’interaction blé/rhizosphère, cette méthodologie implique une standardisation de plusieurs méthodes combinées d’extraction, d’analyse et de traitement des données. L’utilisation de traitement automatique de ces données (Mzmine) et de réseaux moléculaires (MetGem) a permis dans un premier temps de discriminer et d’identifier divers composés différenciant les interactions entre différents génotypes et bactéries de la rizhosphère. Forte de cette expérience, la plate-forme s’est engagée dans un nouveau projet, intitulé « Holoviti », et porté par l’UMR Agroécologie (INRA de Dijon et Université de Bourgogne). Celui-ci vise à étudier la vigne en tant qu’holobionte, c’est-à-dire en considérant l'association entre le végétal (hôte) et les microorganismes associés, dont le fonctionnement est influencé par la dynamique de leur interaction. L’association des résultats GC-MS et LC-MS haute résolution permettra d’avoir une vue très large des différences métabolomiques des divers holobiontes (pour des plantes saines ou dépérissantes) étudiés. Cette approche métabolomique bénéficiera à court terme aux projets étudiant les interactions entre organismes (plantes/plantes mais aussi plantes/microorganimes) en cours au sein de Paris-Saclay et sera notamment bénéfique au projet « Métabolites Spécialisés » de l’EUR SPS. La plate-forme Chimie-Métabolisme-Métabolome propose la quantification, la caractérisation et l'identification de composés chimiques du végétal : les hormones végétales et autres molécules signalisantes, les métabolites primaires (sucres, acides aminés, acides organiques) ou secondaires (flavonoïdes, caroténoïdes, sphingolipides, glucosinolates...), ainsi que les composés issus de la biomasse végétale (paroi végétale, polyphénols...). Elle s'appuie sur l'expertise de ses agents en analyse de chimie du végétal, et sur des appareils de chromatographie, notamment couplés à la spectrométrie de masse (GC-MS, UPLC-MS/MS, UPLC-Q-TOF). La plateforme a noué plusieurs collaborations industrielles ces dernières années (LVMH, Syngenta, Nestlé, Sederma). Contact : François Perreau (francois.perreau@inra.fr); Gregory Mouille (gregory.mouille@inra.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
December 22, 2019 11:48 AM
|
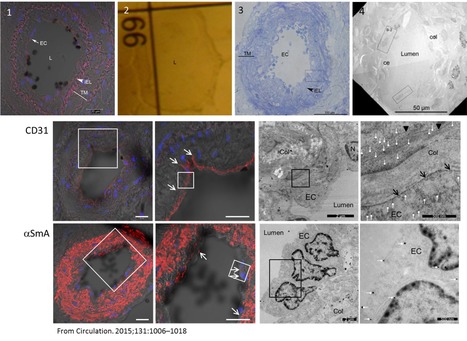
La plateforme d’imagerie cellulaire de l’INSERM UMR-S 999 se trouve sur le site de l’Hôpital Marie Lannelongue au Plessis-Robinson. L’axe principal de recherche de l’unité qui l’héberge est l’étude de l’hypertension artérielle pulmonaire (HTAP), de la physiopathologie à l’innovation thérapeutique. L’HTAP est une maladie pulmonaire rare et sévère qui est définie par une élévation des pressions sanguines au niveau de l’artère pulmonaire (>20mmHg, au repos). D’un point de vue physiopathologique, l’HTAP se caractérise par l’obstruction des artères pulmonaires de calibre inférieur à 500 µm. Ce remodelage vasculaire pulmonaire entraine une augmentation des résistances vasculaires pulmonaires conduisant à une hypertrophie cardiaque droite puis à terme à une insuffisance cardiaque. Il n'existe actuellement aucun traitement curatif. Exemple choisi de développement. Afin de mieux comprendre les mécanismes cellulaires et moléculaires à l’origine de ce remodelage vasculaire pulmonaire, la plateforme d’imagerie cellulaire a mis au point, en collaboration avec la plateforme MIMA2 (INRA, Jouy-en Josas), une méthode originale et simplifiée de la microscopie corrélative permettant i) de sélectionner une artère pulmonaire d’intérêt en microscopie photonique et ii) de suivre à l’échelon ultra-structural en microscopie électronique par transmission les modifications de localisation de protéines d’intérêt. Les travaux menés sur notre plateforme ont notamment permis de démontrer, grâce à cette méthodologie, que la transition endothéliale-mésenchymateuse (changement phénotypique des cellules endothéliales) participait au remodelage vasculaire pulmonaire observé dans l’HTAP (Ranchoux et al., Circulation 2015) et que le canal potassique KCNK3 était moins exprimé par les cellules musculaires lisses des artères pulmonaires remodelées et absent de la membrane plasmique (Antigny et al., Circulation 2016). Cette technique a fait l’objet d’une brève description lors de l’European Microscopy Congress, à Lyon en 2016 (C. Login-Péchoux et al, Simple CLEM method to asses rare pulmonary vascular remodeling) La plateforme d’imagerie cellulaire de l’INSERM UMR-S 999 est équipée d’outils basés sur l’imagerie photonique (imagerie calcique, microdissection laser, électrophysiologie, imagerie confocale) permettant l’identification, la localisation de molécules d’intérêt à l’échelle génomique, transcriptionnelle et protéique et le suivi de processus dynamiques intra- ou extra-cellulaires. En 2019, l'obtention d'un financement par le département Sciences de la Vie de l’Université Paris-Saclay (AAP SDV "Petit Equipement de Laboratoire") a permis le renouvellement de notre source de fluorescence destinée principalement à l’imagerie calcique (Fura-2AM ou Fluo-4/3AM). Ce système est associé à un changement de longueur d’onde d’excitation plus rapide que notre ancien monochromateur (de l’ordre de la micro seconde) permettant un suivi plus fin des signaux de fluorescence émis par les cellules. De plus, le recours à des LEDs comme source de lumière permet de s’affranchir de tout mouvement mécanique susceptible de se dérègler ou de s’user entrainant une meilleure reproductibilité expérimentale. Contacts : Fabrice Antigny (fabrice.antigny@u-psud.fr); Catherine Rucker-Martin (catherine.rucker-martin@u-psud.fr); Raphaël Thuillet (raphael.thuillet@u-psud.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
December 8, 2019 12:38 PM
|
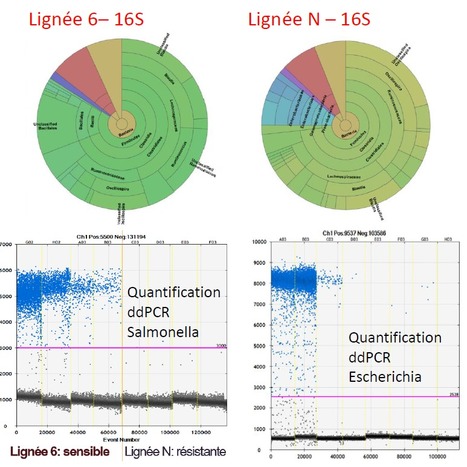
Légende figure : Composition bactérienne du microbiote de poulet par séquençage 16S et quantification de bactéries d’intérêt par ddPCR : Salmonella enteritidis chez le poulet et Escherichia coli dans un lysat protéique complexe. Exemple choisi : Le poulet est porteur sain de Salmonella enterica (SE), bactérie responsable d’intoxications alimentaires majeures. Dans le cadre de ses recherches, l’équipe GeMS de GABI (UMR 1313 - Génétique Animale et Biologie Intégrative) souhaite diminuer le portage de salmonelles, et ceci via 2 stratégies : la première, vise à améliorer la résistance au portage des salmonelles, et pour cela une quantification précise de la présence de cette bactérie dans l’intestin du poulet est requise ; la deuxième, vise à moduler la composition du microbiote de ce poulet. Un modèle animal original est disponible à l’INRA avec deux lignées consanguines divergentes, lignée N plus résistante que lignée 6 au portage (voir illustration ci-dessus). Après infection de 220 animaux par la bactérie, le contenu du caecum a été prélevé et l’ADN extrait. Le séquençage 16S a alors été réalisé par @BRIDGe sur son MiSeqMC (Illumina) et a permis de montrer une différence très significative de composition du microbiote entre les 2 lignées, qui se différencient sur plus de 390 unités taxonomiques. Une différence a aussi été trouvée entre porteurs faibles ou forts de Salmonella enterica au sein de la lignée 6, ouvrant ainsi la voie à une meilleure compréhension du rôle du microbiote sur le portage. De plus, grâce à l’utilisation de PCR digitale (Droplet DigitalTM PCR ou ddPCR, BioRad), @BRIDGe a pu démontrer avec certitude que les animaux résistants au portage (lignée N) ne présentaient aucune copie d’ADN de Salmonella enterica dans leur caecum (voir aussi illustration, les points bleus représentent les gouttelettes où le gène ciblé a été amplifié). A la différence des techniques classiques de qPCR, la ddPCR permet une quantification absolue de quelques copies d’ADN avec une fiabilité surprenante ! Cette haute précision de la PCR digitale et sa robustesse ont aussi été utilisées pour permettre à un industriel français de quantifier l’ADN bactérien Gram négatif (probablement Escherichia coli) présent dans un lysat industriel de nature inconnue de la plateforme. Avec l’appui des experts en microbiologie de l’unité MICALIS (INRA, Jouy-en-Josas), nous avons défini un protocole ddPCR de quantification des bactéries Gram négatif qui a été validé sur des échantillons témoins de bactéries E. Coli et appliqué ensuite avec succès à la quantification de ces bactéries dans le lysat industriel sur la base d’une simple gamme de dilution (illustration). La détection et quantification de bactéries Gram négatif en milieu complexe est aujourd’hui proposée en prestation de service. Ce travail a fait l’objet d’une communication orale lors du 11th European Symposium of Poultry Genetics, symposium qui s’est tenu à Prague (République Tchèque) les 23, 24 et 25 octobre 2019 (Cazals A et al., A combined analysis of genetic and microbial factors determining resistance to Salmonella carriage in chicken). L’offre de services et d’équipements de la plateforme @BRIDGe (animal Biological Resources for Integrated and Digital Genomics, Domaine de Vilvert, Centre INRA de Jouy-en-Josas), intègre la conservation de l’échantillon biologique et son analyse au niveau moléculaire, cellulaire ou tissulaire. Les domaines d’activité sont la génomique structurale et fonctionnelle, la microgénomique (analyse génomique à l’échelle cellulaire), l’analyse morphologique. Le Centre de Ressources Biologiques permet une gestion sécurisée des échantillons, leur caractérisation et leur distribution, ainsi que la fourniture de procédures expérimentales et d’outils pour l’analyse des données. La biobanque et certains équipements de génomique font partie de l’infrastructure nationale CRB-Anim soutenue par le programme ‘Investissements d’Avenir’ de 2012 à 2019. @BRIDGe a un partenariat avec la société Excilone qui lui permet d’accéder aux technologies de pointe dans le domaine de la microgénomique (instruments, analyse genomique tissus FFPE, exosomes, etc.). La plateforme @BRIDGe est certifiée ISO 9001:2015 et NFS 96 900 pour sa partie biobanque. La biobanque a été labellisée ISC (Infrastructure Scientifique Collective) par l’INRA en 2018. Contact : Michèle Tixier-Boichard (michele.boichard@inra.fr), Claudia Bevilacqua (claudia.bevilacqua@inra.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
November 24, 2019 5:11 PM
|
La plateforme PAPPSO (ou Plateforme d’Analyses Protéomiques de Paris Sud-Ouest) est une structure entièrement dédiée à la protéomique et qui a su relever les défis de la Métaprotéomique ! La métaprotéomique, ou protéomique des consortia microbiens, est une expertise analytique devenue indispensable dans un contexte d’émergence forte des études d’impact des communautés microbiennes sur l’environnement et la santé. A ce titre, PAPPSO collabore étroitement avec les unités Micalis, MaIAGE et MetaGenoPoliS de l’INRA/Jouy-en-Josas, mais aussi avec diverses équipes parisiennes de l’AP-HP (Hôpitaux Saint-Antoine (Harry Sokol) et Pitié-Salpêtrière (Karine Clément)) ou en province (IPHC, Strasbourg), et ceci sur des projets innovants orientés diagnostic et santé humaine, et dans des domaines aussi variés que les maladies inflammatoires chroniques de l’intestin (MICI), l’obésité et les interventions nutritionnelles ou chirurgicales associées, ou encore les facteurs de risque cardiovasculaire. Exemple choisi : Dans le cadre du projet ProteoCardis (2015-2019, ANR, Catherine Juste), PAPPSO vient d’achever l’analyse du métaprotéome intestinal de la plus grosse cohorte jamais constituée dans son domaine : 138 patients souffrant de maladies coronariennes, 50 témoins (volontaires sains) et 30 sujets avant et après chirurgie bariatrique ! D’un point de vue technique, la préparation des échantillons a été poussée à l’extrême afin de révéler aussi bien les protéines du cytoplasme que les protéines d’enveloppes en interaction avec les protéines humaines du tractus digestif. L’acquisition des données de spectrométrie de masse a pris à lui seul plus de 4000 heure-machines (!) et l’interprétation de ces dernières a nécessité le développement de nouveaux outils bio-informatiques adaptés au traitement de données massives : X!TandemPipeline et MassChroQ (logiciels libres d’accès et disponibles sur le site de la plateforme PAPPSO sous licence GPLv3). Résultats préliminaires : 560 000 peptides et 100 000 protéines représentant 4600 fonctions KEGG (Kyoto Encyclopedia of Genes and Genomes) mais aussi de 3000 espèces bactériennes identifiées. Inutile de préciser que la valorisation complète des données acquises est titanesque et se poursuivra donc encore sur plusieurs mois. Seront dans un premier temps identifiées les fonctions et consortia bactériens associés au risque cardiovasculaire avec pour objectif la découverte de marqueurs précoces utilisables pour le diagnostic et le suivi des patients. Dans un deuxième temps, les données générées viendront aussi alimenter d’autres projets génériques ou appliqués, et pris en charge par la plateforme. PAPPSO, largement ouverte à la communauté scientifique Paris-Saclay, possède une expertise forte dans l’analyse des micro-organismes et animaux à Jouy-en-Josas mais aussi des plantes à la ferme du Moulon de Gif-sur-Yvette. PAPPSO est spécialisée dans le développement de méthodes analytiques, d’outils bio-informatiques (http://pappso.inra.fr/bioinfo/) et d’approches statistiques permettant de répondre au mieux aux attentes de ses partenaires. Sa carte de visite est aujourd’hui sa capacité unique à analyser de grandes cohortes et des échantillons très complexes. Sur les cinq dernières années, la plateforme a été et continue d’être partenaire de projets de prématuration de la SATT Paris-Saclay, et porteurs ou partenaires de projets financés par l’ANR mais aussi l’Europe. PAPPSO est aussi co-inventeur de plusieurs brevets dont un portant sur des peptides marqueurs de la maladie de Crohn (EP3234190A2, US20170349930A, priority date 2014-12-19). Contact : Céline Henry (celine.henry@inra.fr), Michel Zivy (michel.zivy@inra.fr) et Véronique Monnet (veronique.monnet@inra.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
November 10, 2019 11:13 AM
|
La Plateforme de biochimie environnementale (Biochem-Env) a été créée en 2012 dans le cadre de l’Infrastructure de Recherche « Analyses et Expérimentations pour les Ecosystèmes » (AnaEE France). Rattachée à l’UMR ECOSYS, elle offre ses services aux équipes de recherche locales, nationales et internationales dans les champs de l’agroécologie et de l’écotoxicologie. Elle réalise le développement et la mesure d’indicateurs biochimiques dans l’environnement et les organismes des écosystèmes continentaux. Ses missions concernent 1) la conduite de projets de recherche et la réalisation de prestations portant sur l’expérimentation et l’observation sur les écosystèmes, dans le contexte de l’Open Science, 2) le développement, la validation et le transfert des méthodes d’analyse et d’interprétation des résultats, incluant la normalisation, 3) la mise à disposition de compétences, de matériels et de locaux pour l’analyse, et 4) la réalisation d’actions d’expertise, de veille scientifique et technique, ainsi que de formation. A la création de la plateforme, les méthodes de mesure d’indicateurs biochimiques tels que les activités enzymatiques du sol étaient peu standardisées, et mobilisaient des protocoles relativement lourds et chronophages. Cette diversité de méthode, associée à des modes différents d’expression des résultats, constituait des handicaps à la mise en place de programmes d’expérimentation ou d’observation de grande envergure, comme la comparaison et l’interprétation de résultats issus de différentes équipes. Biochem-Env a développé, en partenariat avec la société Hamilton, une chaine robotisée de mesure actuellement dédiée aux activités enzymatiques de sols et sédiments. Cette chaine permet à la plateforme de traiter quotidiennement la mesure simultanée de plusieurs activités enzymatiques sur une soixantaine d’échantillons, avec fidélité et justesse. La méthode a été validée par un essai inter-laboratoire européen et fait l’objet d’une norme internationale ISO 20130:2018 (Soil quality - Measurement of enzyme activity patterns in soil samples using colorimetric substrates in micro-well plates). Forte de divers labels (IBISA, ISC INRA) et certificat (ISO9001), Biochem-Env développe maintenant son offre de service à destination de son réseau de partenaires académiques et privé (Cheviron et al, Environmental Science and Pollution Research 2018). Biochem-Env est une plateforme scientifique et technique centrée sur le développement et la mesure d'indicateurs biochimiques dans l’environnement et les organismes des écosystèmes continentaux. Dans l'environnement (sols et sédiments), la plateforme permet la mesure d'indicateurs fonctionnels (activités enzymatiques impliquées dans les cycles biogéochimiques, métabolisme des macromolécules, activité métabolique globale, respiration…). Elle réalise également la mesure d'indicateurs biochimiques chez les invertébrés benthiques et terrestres (réserves énergétiques et macromolécules, stress oxydant, mécanismes de détoxication, exposition aux contaminants environnementaux...). Biochem-Env offrira à la communauté scientifique des jeux de données ouverts concernant les indicateurs biologiques liés à la biodiversité fonctionnelle des écosystèmes. Les informations et connaissances obtenues par la plateforme permettront le développement d'approches mathématiques et de modélisation pour évaluer et prévoir les impacts de perturbations de l'environnement sur la biodiversité fonctionnelle. Contact : Christian Mougin (christian.mougin@inra.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
October 28, 2019 9:43 AM
|
La Plateforme AnimEx de l’UMS IPSIT (Faculté de pharmacie, Université Paris-Sud) offre à la communauté scientifique des structures d’hébergement et d’élevage d’animaux et des équipements pour l’exploration fonctionnelle du petit animal. Elle est aussi en mesure de réaliser des protocoles expérimentaux à façon pour des laboratoires et collabore depuis 2016 avec l’Agence Française de Lutte contre le Dopage (AFLD – Châtenay-Malabry) afin de valider des méthodes de détection de substances interdites chez l’homme (nouvelles molécules/médicaments non autorisés ou en cours d’étude clinique) et d’évaluer leurs effets sur la performance. Dans ce cadre, un travail collaboratif a fait l’objet d’une récente publication (Marchand et al. 2019) et a porté sur des médicaments en cours d’essais cliniques qui stimulent la production de globules rouges, appelés stabilisateurs d’HIF (facteur inductible par l’hypoxie). Ce sont de petites molécules synthétisées chimiquement qui vont activer les voies de régulation de l’hypoxie. Le développement récent de ces médicaments offre une nouvelle stratégie pour traiter l’anémie mais en lien direct avec les effets prometteurs observés, l'abus pour dopage a déjà commencé, les sportifs malintentionnés pouvant se procurer ces nouvelles molécules par le marché noir sur internet. L'identification des biomarqueurs des stabilisateurs HIF aiderait donc à détecter leur utilisation par des athlètes. L’objectif du projet était double, d’une part déterminer si les hypoxamiRs, les microARNs associés à la réponse cellulaire à l'hypoxie, sont des biomarqueurs potentiels des stabilisateurs HIF dans le sang et d’autre part si la réponse au traitement avec un stabilisateur d’HIF diffère de la réponse à un environnement hypoxique. Pour ce faire des rats ont été traités pendant 6 jours avec un placebo ou 2 mg/kg de Molidustat, un stabilisateur d’HIF, ou ont été mis en hypoxie (10% d'O2) pour la même durée et des échantillons de plasma ont été collectés et analysés avant, pendant et 48 heures après les traitements. Résultats : Les hypoxamiRs miR-130a et miR-21 ont été spécifiquement et sensiblement augmentés pendant le traitement de Molidustat et miR-21 en particulier est resté surexprimé 48 heures après la fin du traitement. Bien que l'utilisation de ces microARN comme biomarqueurs semble peu probable en raison d'autres facteurs possibles de régulation des microARNs miR-130a et miR-21, cette étude a fourni la première identification d'un effet spécifique des stabilisateurs HIF sur des microARNs. D'autres études sont nécessaires pour mieux comprendre les conséquences possibles d'une telle régulation. La collaboration entre l’AFLD et la plateforme AnimEx continue pour aider à l’évolution future de la lutte contre le dopage.… La Plateforme AnimEx (Animalerie et Exploration fonctionnelle du petit animal) est située sur deux sites de l’Université Paris-Sud, la Faculté de Pharmacie de Châtenay-Malabry et le Centre INSERM de l’Hôpital Antoine-Béclère à Clamart. Sa mission principale est d’offrir à la communauté scientifique du secteur public ou privé, des structures d’hébergement et d’élevage d’animaux et des équipements pour l’exploration fonctionnelle du petit animal. AnimEx est membre de CAPSud (Consortium des Animalerie Paris-Sud), est rattaché au Comité d’éthique CEEA n°26 et utilise le logiciel Tick@Lab pour la gestion de ses animaux. Contact : valerie.domergue@u-psud.fr Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
October 12, 2019 6:24 PM
|
Démonstration ! De nombreux animaux reconnaissent les membres de leur famille et coopèrent plus facilement avec eux qu’avec d’autres individus, les exemples les plus connus étant les primates et les insectes sociaux comme les abeilles ou les fourmis. Une étude publiée récemment du laboratoire Ecologie, Systématique et Evolution impliquant la plateforme Serre de Biologie Végétale (Fortuna T. et al., Mol. Ecol. 2018) a montré que les champignons aussi sont capables de reconnaître leurs apparentés et de partager préférentiellement leurs ressources avec eux. Les champignons pathogènes Microbotryum castrent leurs plantes hôtes en produisant leurs propres spores à la place du pollen dans les anthères (voir illustration, fleur de la plante Saponaria officinalis parasitée par le champignon pathogène Microbotryum saponariae). Un génotypage des spores du champignon a montré que plusieurs individus du champignon coexistent dans la plante dans les populations naturelles, et doivent donc se partager les ressources de la plante, mais que les individus au sein d’une même plante sont apparentés. Des inoculations expérimentales en serre sur des lignées clonales de plantes ont confirmé que les individus du champignon qui colonisaient une même plante étaient apparentés. Des expériences de croissance du champignon in vitro suggèrent que ce phénomène est dû à une exclusion des souches non apparentées qui se produit via une croissance ralentie. Cette étude montre donc que les champignons sont capables de comportements visant à partager leurs ressources préférentiellement avec leurs apparentés, ce qui est en accord avec la théorie de la sélection de parentèle expliquant l’évolution de la coopération. D’autre part, ces résultats sont importants pour comprendre l’évolution des maladies des plantes. En effet, la coexistence de plusieurs individus de pathogène dans un hôte et leur degré d’apparentement sont connus pour impacter la sévérité des maladies. La Serre de Biologie Végétale de l'UFR Sciences est située dans le campus d'Orsay. Elle est équipée de compartiments de serre et de chambres climatiques pour les cultures expérimentales en niveau de confinement S1. Elle possède aussi quelques parcelles de pleine terre sous serre en S1. Les surfaces de cultures peuvent être louées pour la recherche publique ou privée, et pour l'enseignement. Contact : virginie.heraudet@u-psud.fr Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
September 29, 2019 10:41 AM
|
La Plateforme d’Imagerie Multiphotonique du Petit Animal (PIMPA) située sur le campus d’Orsay et rattachée au laboratoire « Imagerie et Modélisation en Neurobiologie et Cancérologie (IMNC) » permet notamment de détecter le signal de génération de second harmonique (SHG) sur échantillon vivant. Exemple choisi d’application : L’équipe « biophotonique » du Laboratoire Aimé Cotton (CNRS, Univ. Paris-Sud et ENS Paris-Saclay) animée par François Treussart (francois.treussart@ens-paris-saclay.fr) a mis au point sur la plateforme PIMPA, avec l’aide d’Isabelle Garcin (isabelle.garcin@u-psud.fr), une méthode originale de mesure du transport intraneuronal. En effet, cette équipe analyse le transport endosomal dans des cultures primaires de neurones de souris à l’aide de nanocristaux à réponse optique non-linaire internalisés spontanément dans les endosomes. Elle utilise le signal de génération de second harmonique (SHG) généré par les nanocristaux sous l’effet des impulsions laser brèves du laser de la plateforme, à une longueur d’onde du proche infrarouge. L’intensité élevée du signal SHG, combinée au balayage rapide du faisceau d’excitation assuré par le scanner galvanométrique résonant, ont permis de suivre fidèlement les déplacements rapides des endosomes à des vitesses de plusieurs µm/s. Leurs trajectoires sont ensuite reconstruites à partir des vidéos du signal SHG, puis analysées quantitativement pour évaluer l’impact fonctionnel de perturbations d’intérêt (e.g. facteurs de risque génétique de maladies, molécule potentiellement toxique…). La validation de cette méthode en culture monocouche permet maintenant de l’étendre aux cultures 3D et à des tranches de cerveaux de souris, pour lesquelles l’excitation dans la fenêtre de transparence optique NIR est indispensable pour s’affranchir de l’autofluorescence tissulaire (S. Haziza et al., 2018). La Plateforme d’Imagerie Multiphotonique du Petit Animal (PIMPA) a pour principal objectif de proposer à la communauté scientifique, académique ou privée un outil de pointe destiné à l'imagerie optique non linéaire chez le petit animal. La plateforme est implantée au bâtiment 440 du campus d'Orsay dans une zone de confinement P2 et à proximité de l'animalerie de l'IBAIC. Cette plateforme est équipée d'un dispositif instrumental offrant la possibilité de combiner les imageries confocale et biphotonique du petit animal et est également ouverte à l'analyse cellulaire et tissulaire. Ainsi configuré, le dispositif a la spécificité de rassembler différentes modalités d'imagerie (spatiale, temporelle et spectrale), multimodalité d'importance majeure pour les expériences in vivo. A cela s'ajoute l'acquisition et la reconstruction tridimensionnelles en temps réel ou différées de processus biologiques de l'échelle cellulaire à tissulaire. PIMPA est une plateforme du programme investissement d’avenir France Life Imaging et membre du Réseau d’Imagerie Cellulaire (RIC) de Paris-Saclay. Contact : darine.abihaidar@imnc.in2p3.fr Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
September 15, 2019 10:20 AM
|
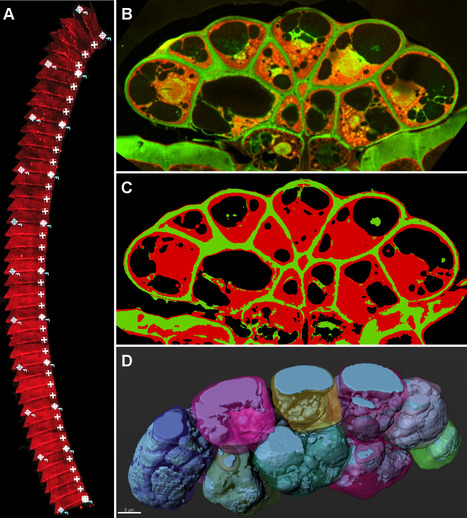
La Plateforme d’Imagerie Photonique (Institut de Biologie Intégrative de la Cellule – I2BC, Gif-sur-Yvette) donne accès à un parc de microscopes équipés pour répondre aux besoins d’imagerie de différents modèles cellulaires étudiés en biologie. Elle s’approprie régulièrement de nouvelles méthodes d’imagerie et met cette richesse d’expertise au service de la communauté scientifique de Paris-Saclay toute entière. Tout récemment, notre plateforme a développé un ensemble méthodologique permettant l’imagerie et la reconstruction 3D de gros volumes par microscopie confocale à balayage laser automatisée et l’analyse optimisée par une approche s’appuyant sur le « machine learning ». Contexte de ce développement et démonstration par l’image ! Les plantes carnivores subissent des remaniements majeurs de leur système endomembranaire lorsqu’elles entrent dans un processus de digestion, ce qui en fait un modèle d’étude idéal pour la morphologie des endomembranes. La microscopie électronique est alors la technique de choix pour les études morphologiques résolutives. Cependant elle s’avère longue et fastidieuse lorsqu’il s’agit d’imager de grands volumes comme des organes entiers. Dans notre cas, il s’agissait de glandes sécrétrices de Dionée, organes pour lesquelles typiquement des centaines de coupes sériées peuvent être réalisées (Figure A) après fixation chimique et enrobage en résine acrylique. Ces sections (250 nm d’épaisseur) peuvent être colorées puis imagées de manière automatique au microscope confocal à balayage laser (Figure B) permettant l’obtention d’images de très bonne résolution. Elles permettent également de séparer spectralement les composés fluorescents ou autofluorescents présents dans les différents compartiments cellulaires. Ces très grands volumes de données sont alors analysés par une approche de « machine learning » afin de détecter et segmenter automatiquement sur chaque image, les parois, le cytoplasme ainsi que les vacuoles (Figure C). Cette étape de segmentation automatique est d’autant plus facile que les marqueurs fluorescents sont discriminants spatialement et que les volumes de données sont importants pour « entrainer » le logiciel de machine learning. Enfin, ces données sont traitées par un logiciel de reconstruction des volumes (IMARIS) permettant d’extraire des objets et mesurer des volumes (Figure D). Ici, les données spectrales obtenues par microscopie confocale ont été essentielles pour permettre une reconnaissance automatique des compartiments cellulaires. Ce protocole d’analyse d’images a permis de mettre en évidence et de quantifier en 3 dimensions une forte fragmentation des vacuoles présentes dans le cytoplasme des cellules des glandes sécrétrices des plantes carnivores, lors du processus de digestion de proies. Ce travail a fait l’objet d’une communication par poster lors des 17èmes Journées du Réseau de Centres Communs de Microscopie (RCCM, aujourd’hui le RIME ou Réseau d’Imagerie en Microscopie Electronique) les 6, 7 et 8 juin 2018 à Clermont-Ferrand (Boulogne C et al., 3D analysis of secretory system in digestive glands of Venus flytrap). A noter : Ce travail a été mené en collaboration avec une autre plateforme d’Imagerie-Gif (I2BC) : la Plateforme de Microscopie Electronique (http://www.i2bc.paris-saclay.fr/spip.php?article282, Cynthia Gillet). Les différents systèmes d'imagerie de la Plateforme d’Imagerie Photonique permettent des approches multi-échelles allant du tissulaire (macroscopie), cellulaire (microscopie confocale) jusqu'au niveau subcellulaire (microscopie super résolue). La plateforme est hébergée au sein de l'Institut de Biologie Intégrative de la Cellule (I2BC), ce qui lui confère une forte expertise dans l'étude de différents modèles biologiques cellulaires : cellules animales, végétales, plantules, bactéries, levures… Afin de permettre aux utilisateurs d'exploiter au mieux les données obtenues sur les microscopes, ils sont également accompagnés par les ingénieur(e)s de la plateforme pour le traitement et l’analyse des images. Des stations de calcul puissantes équipées de logiciels dédiés sont mises à leur disposition. Certifiée ISO 9001 :2015/NFX 50-900 :2016, la plateforme accueille régulièrement des projets du secteur privé. Contact : romain.lebars@i2bc.paris-saclay.fr Plug In Labs Université Paris-Saclay : cliquer ici
|

|
Scooped by
Life Sciences UPSaclay
March 1, 2020 4:24 PM
|
Genopole et l’Université Paris-Saclay organisent le mardi 28 avril 2020 la deuxième édition de MeetMyPlatform | Sciences de la Vie, en partenariat avec le Crédit Agricole Consumer Finance qui nous accueillera dans ses locaux à Massy. MeetMyPlatform | Sciences de la Vie : Des temps forts scientifiques - une plénière, une table ronde industrielle, trois ateliers - des opportunités uniques de découverte et de partage avec les 113 plateformes académiques et 20 partenaires industriels présents, sans oublier des moments de convivialité et d’échange ... Et vous ? y serez-vous aussi ? Cliquer sur le lien ci-dessous pour découvrir le programme complet de cette journée, mais aussi vous inscrire (inscription gratuite mais obligatoire) ! PROGRAMME | INSCRIPTION EN LIGNE MeetMyPlatform | Sciences de la Vie est une journée de rencontres entre plateformes et entreprises, organisée par Genopole et le Département Sciences de la Vie de l’Université Paris-Saclay. Son objectif est double : rassembler et donner de la visibilité à la communauté des plateformes de l’Université Paris-Saclay et permettre aux acteurs de la recherche mais aussi aux entreprises présentes lors de cet évènement, de les découvrir ou de renforcer des liens déjà existants. Pour mémoire, la première édition de MeetMyPlatform | Sciences de la Vie s’était tenue le 31 janvier 2019 - un jour de neige ! – et avait su rassembler sur le plateau de Saclay près de 300 participants : ingénieurs et techniciens de plateformes, acteurs de la recherche académique mais aussi représentants de grands groupes, de PME/ETI et de Start-up/TPE. Ces derniers ont particulièrement apprécié la convivialité de l’événement et la facilité d’obtention d’informations pertinentes dans leurs domaines d’activités. Les membres des plates-formes ont, quant à eux, apprécié ce mode de communication et de présentation de leurs équipements, expertises et savoir-faire, ainsi que les moments d’échanges privilégiés (ateliers) qui leur étaient consacrés en deuxième partie de journée pour notamment renforcer ou structurer de nouveaux réseaux thématiques au niveau sud-francilien. Rendez-vous le mardi 28 avril 2020 pour une deuxième édition ! Vous souhaitez en savoir plus sur MeetMyPlatfom, cliquer ICI pour une présentation en 70 secondes chrono ! MeetMyPlatform | Sciences de la Vie s’inscrit naturellement dans la dynamique continue de mise en visibilité des compétences, expertises et équipements des plates-formes de l’Université Paris-Saclay et ceci notamment via son portail PlugInLabs Université Paris-Saclay. Comité d’organisation : Frédéric Dollé (frederic.dolle@cea.fr), Marion Le Devedec (marion.ledevedec@universite-paris-saclay.fr), Jessica Pericaud (jessica.pericaud@universite-paris-saclay.fr) et Julien Picot (julien.picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
February 16, 2020 12:55 PM
|
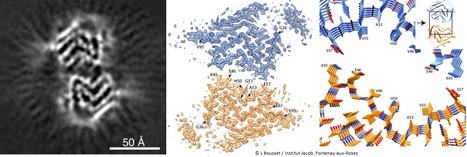
La cryo-microscopie électronique (cryoEM) consiste à congeler très rapidement des échantillons hydratés, afin de les figer dans une glace amorphe (vitrification). Elle n’utilise aucun additif, ce qui permet la préservation de l’état natif de l’échantillon et donc la détermination de sa structure interne. Les images enregistrées sont des projections de l’échantillon avec un contraste faible. Elles sont soumises à une analyse computationnelle, menant à la structure tridimensionnelle de l’objet d’intérêt. Ces dernières années, la cryoEM a révolutionné de façon spectaculaire, le domaine de la Biologie Structurale et permis des avancées majeures en Santé et Médecine. Ceci a été reconnu par le prix Nobel de chimie en 2017 attribué aux pionniers de ce domaine. La cryoEM permet des études à haute résolution spatiale sur des complexes réputés difficiles à étudier et notamment à cristalliser. Dans le meilleur des cas, l’étude à l’échelle atomique des états intermédiaires des machineries cellulaires est possible : la cryoEM permet en effet de classer les images en fonction de la conformation de l’objet à laquelle elles correspondent. Exemple choisi : En collaboration avec une équipe de l’Institut Jacob (L. Bousset, MIRCen, CEA, Laboratoire des Maladies Neurodégénératives, Fontenay-aux-Roses), la plateforme de cryo-microscopie électronique (Institut de Biologie Intégrative de la Cellule – I2BC, Gif-sur-Yvette) a contribué à la résolution de la structure atomique de deux polymorphes de l’α-synucléine. Ces structures résolues par cryoEM à 3.0 Å et 3.4 Å de résolution montrent des structures radicalement différentes des structures précédemment publiées. Ces structures permettent de proposer de nouvelles hypothèses sur le mécanismes d’assemblage in vitro des fibres amyloïdes et sur l’impact de mutations familiales associées à la maladie de Parkinson. Récemment publiés (Guerrero-Ferreira et al., eLife, 2019), ces travaux ont également fait l’objets de deux communiqués institutionnels, l’un par le CNRS, l’autre par le CEA (voir également notre info sur Scoop.it!). La plateforme de cryo-microscopie électronique de l’I2BC (CRYO-EM), fait partie des plateformes de Biologie Structurale de l’I2BC (Institut de Biologie Intégrative de la Cellule). Elle dispose de deux cryo-microscopes électroniques 200 kV et 120 kV (Tecnai FEI), équipés d’une caméra à détection directe (K2 Gatan) et d’un porte-objet refroidi (Gatan 626). L’équipement de la plateforme permet l’observation des objets biologiques (protéines, complexes multi-protéiques, virus, liposomes, assemblages multi-moléculaires, ...) après coloration négative, ou par cryo-microscopie électronique dans leur milieu aqueux naturel. La coloration négative permet de vérifier la qualité et l’homogénéité des échantillons, mais aussi d’avoir une idée de la forme des objets et éventuellement de déterminer la stœchiométrie de certains assemblages. Les images obtenues en cryo-microscopie électronique sur le FEI Tecnai G20, ainsi que les moyens de calcul disponibles sur la plateforme, permettent la détermination de structures 3D à moyenne résolution (8 Å). Ces premières structures sont nécessaires pour accéder aux microscopes plus puissants (eg. 300 kV Titan Krios de l’ESRF, Grenoble ou de l’IGBMC, Grenoble), menant aux résolutions atomiques. La plateforme permet aussi l’observation de complexes in situ (protéines à la surface d'organites purifiés ou de virus enveloppés, protéines membranaires reconstituées dans des liposomes, ...) par cryo-tomographie électronique. Cette plateforme fait partie d’un ensemble de plateformes de Biologie Structurale de l’I2BC, labélisées IBISA qui comprend: i) la plateforme de spectroscopie RMN (Résonance Magnétique Nucléaire). Cette technique est particulièrement bien adaptée pour analyser les états fonctionnels multiples, la flexibilité interne et les cinétiques de phosphorylation des protéines ; ii) la plateforme de cristallisation qui permet de réaliser le criblage, l’analyse et l’optimisation automatisés des conditions de cristallisation des macromolécules ; iii) la plateforme de mesures d’interactions de mesures macromoléculaires (protéine-ligand) qui offre un large éventail d’équipements pour réaliser des mesures d’interactions et de contrôles qualités avec notamment la microcalorimétrie, l’ultracentrifugation analytique, la MST, le SEC-MALS et le switchSense. Contact : Ana ARTENI (ana-andreea.arteni@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
February 1, 2020 12:48 PM
|
Exemple choisi. Equipée des technologies Illumina et Nanopore, la plateforme de transcriptomique de l’IPS2 (POPS) est impliquée dans un large projet d’analyses omiques avec les équipes du professeur Marc-André Selosse du Muséum National d’Histoire Naturelle de Paris et de l’Université de Gdansk (Pologne) visant à comprendre les relations complexes qu’entretiennent les orchidées européennes avec leur partenaires mycorhiziens. On observe en effet chez cette famille de plantes un large gradient de comportements trophiques, allant d’une symbiose classique où la plante et le champignon échangent des nutriments, au parasitisme où la plante reçoit tous ses nutriments du champignon. Dans ce dernier cas, l’orchidée a perdu sa capacité à faire de la photosynthèse. Etudier cette transition évolutive au laboratoire est impossible. Ainsi plusieurs centaines d’échantillons correspondant à 10 espèces d’orchidées différentes ont été prélevés aux printemps 2018 et 2019 sur des sites naturels polonais. En collaboration avec la plateforme Métabolisme-Métabolome de l’IPS2, le transcriptome et le métabolome de ces échantillons ont été analysés. Un des verrous majeurs à une analyse de transcriptome pertinente dans ces plantes est l’absence de génome séquencé disponible. Sans génome, il est impossible de connaitre quels sont les gènes qui sont activés dans une condition donnée. Sur la base des données de séquençage de transcriptome Illumina, il est possible d’assembler une liste de gènes exprimés mais cette liste reste très fragmentaire. Cela rend extrêmement complexe la comparaison des transcriptomes de ces 10 espèces. En les combinant avec des données de séquençage Nanopore, la plateforme POPS a réalisé des assemblages « hybrides » des transcriptomes qui s’avèrent de bien meilleure qualité que les assemblages basés uniquement sur les données Illumina. En associant dans un deuxième temps ces données transcriptomiques aux données métabolomiques, il devient alors possible de comparer la physiologie de ces 10 espèces d’orchidées. Bien que le gros des analyses soit encore en cours, les premiers résultats suggèrent que le cœur de cette transition évolutive se situe dans la plasticité métabolique des plantes et dans les capacités d’échanges entre le champignon et sa plante hôte (Lallemand et al. Plant J. 2019). Ce travail ouvre également de nouvelles perspectives pour l’analyse et la comparaison du transcriptome d’espèces d’intérêt mais qui ne sont pas des modèles de laboratoire. La plateforme de transcriptomique POPS est localisée à l’IPS2 à Gif-sur-Yvette (plateau du Moulon). Elle réalise des projets d’étude du transcriptome en collaboration avec des équipes de recherche nationales et internationales. Elle offre une expertise pour l’analyse de l’expression comparative des gènes par l’utilisation du séquençage à haut débit de l’ARN, ou RNA-seq. Cette offre est complétée par l’analyse bio-informatique et statistique des données brutes permettant l’analyse quantitative du transcriptome. POPS est certifiée ISO 9001 :2015, labélisée IBiSA et est membre des Réseaux GENOPS et SPS. Contacts : Ludivine Soubigou-Taconnat et Etienne Delannoy (pops.ips2@u-psud.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
January 19, 2020 12:12 PM
|
La Plateforme de biochimie environnementale (Biochem-Env) est centrée sur le développement et la mesure d'indicateurs biochimiques dans l’environnement et les organismes des écosystèmes continentaux. Dans l'environnement (sols et sédiments), la plateforme permet la mesure d'indicateurs fonctionnels (activités enzymatiques impliquées dans les cycles biogéochimiques, métabolisme des macromolécules, activité métabolique globale, respiration…). Elle réalise également la mesure d'indicateurs biochimiques chez les invertébrés benthiques et terrestres (réserves énergétiques et macromolécules, stress oxydant, mécanismes de détoxication, exposition aux contaminants environnementaux...). Après avoir robotisé certaines de ses méthodes de mesure au laboratoire, Biochem-Env poursuit le développement de son offre de service à destination de ses partenaires de recherche et de formation. A compter du printemps 2020, elle disposera d’un Lab-Mobile aménagé et équipé pour la collecte et la préparation d’échantillons environnementaux, et la mesure des indicateurs biochimiques. Cet équipement unique lui permettra de maintenir une excellente qualité de prestation lors de campagnes importantes de terrain, tout en offrant un confort de travail accru pour ses collaborateurs. Le Lab-Mobile sera également un outil de formation dans le cadre de projets ouverts à l’enseignement technique et supérieur. L’achat et l’équipement du Lab-Mobile ont été soutenus par l’Infrastructure de Recherche AnaEE-France, le GIS IBiSA et l’initiative Recotox. L’équipe Biochem-Env est heureuse de vous inviter à l’inauguration du Lab-Mobile le lundi 3 février 2020 à 13h30 sur le centre INRAE de Versailles, au niveau du bâtiment 6. Afin de faciliter l’organisation de cet événement, merci de vous inscrire via le lien ci-contre : Inauguration du Lab-mobile L’ensemble de ces équipements permettra à Biochem-Env d’offrir à la communauté scientifique des jeux de données ouverts concernant les indicateurs biologiques liés à la biodiversité fonctionnelle des écosystèmes. Les informations et connaissances obtenues par la plateforme permettront le développement d'approches mathématiques et de modélisation pour évaluer et prévoir les impacts de perturbations de l'environnement sur la biodiversité fonctionnelle. Contact : Christian Mougin (christian.mougin@inrae.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
January 3, 2020 5:58 PM
|
La plateforme Epigénomique et recherche translationnelle de l’IPS2 (EPITRANS, Gif-sur-Yvette) a pour but d’identifier les gènes et les régulations épigénomiques majeurs contrôlant les caractères agronomiques puis à créer des allèles qui permettent d’améliorer la performance des plantes dans les conditions de cultures. Elle se positionne comme pierre angulaire entre la recherche fondamentale et la recherche appliquée, sur le territoire Paris-Saclay, mais aussi au niveau national. Exemple choisi de développement : Au début des années 2000, l’équipe du Dr. A. Bendahmane (IPS2) se passionne sur l’étude des mécanismes moléculaires qui contrôlent le déterminisme du sexe chez les Cucurbitacées. En effet, depuis le travail pionnier de G. H. Shull sur la production des semences hybrides, les sélectionneurs n’ont pas cessé d’essayer de maîtriser les caractères génétiques contrôlant la fertilité et le sexe de la fleur chez les plantes cultivées. En parallèle, des outils de génomique fonctionnelle comme le TILLING (Targeting Induced Local Lesions in Genomes) sont développés afin de valider la fonction des gènes identifiés, donnant naissance à notre plateforme de recherche translationnelle. Soutenus par un ERC Advanced Grant en 2013 (SEXYPARTH), ces travaux de recherche aboutissent aux clonages de 5 gènes, brevetés ou en cours de brevetabilité, permettant d’induire chez n’importe quelle variété de cucurbitacées un morphe sexuel au choix : gynoïque, androïque, monoïque ou hermaphrodite (Boualem et al. Science 2008 ; Martin et al. Nature 2009). L’activité de la plateforme explose alors avec la production de plus de 50 000 lignées mutées chimiquement, rien que chez les Curcurbitacées (Dahmani-Mardas et al. PLoS One 2010) et le développement d’outils de plus en plus performants pour la création de nouveaux allèles (TILLING par NGS, mutagenèse saturante). Ainsi des plantes mutantes avec des morphes sexuels différents ont été produites pour diminuer le coût de production de graines hybrides F1 (nécessitant des croisements interlignées) ou pour améliorer les rendements. Fort de ce succès et de l’intérêt de l’industrie semencière pour ces morphotypes, un projet de maturation porté par la SATT Paris-Saclay a été sélectionné. Son objectif est de créer des prototypes de plantes, destinés à améliorer les rendements ou diminuer les couts de la production des semences hybrides, par une approche de génomique à haut-débit et non-OGM : le TILLING. La plateforme EPITRANS, contraction d'EPIgénomique et de recherche TRANSlationnelle, a pour missions de valoriser la recherche fondamentale en transformant les idées en produits et de faire le lien entre le chercheur et le secteur économique. Elle s'intéresse aux allèles et épi-allèles qui améliorent la performance des plantes dans un environnement de plus en plus contraint et plus particulièrement chez les espèces d'intérêt agronomique. Son activité consiste à développer et à mettre à disposition de la communauté scientifique des outils de génétique haut-débit, forward et reverse, chez les végétaux, notamment lorsque les études traditionnelles de génétiques ne peuvent s'appliquer (transformation génétique). Cinq outils sont ainsi proposés : le clonage positionnel par NGS, le TILLING et l'ECO-TILLING, l'Epigénomique et le CRISPR. Le premier permet de cloner des gènes d'intérêts agronomiques pour ensuite étudier leur(s) fonction(s) par TILLING, par recherche de modifications épigénomiques, ou encore par édition du génome via le système CRISPR (selon les espèces). Le TILLING s'appuie sur la production de larges collections (>5000 lignées) de plantes mutées ou de collections de germoplasmes (ECOTILLING) combinée à une identification rapide et systématique des mutations dans les séquences cibles. Ces allèles, non génétiquement modifiés, sont proposés aux sélectionneurs pour améliorer leurs lignées élites en leur offrant une alternative aux collections limitées de germoplasmes. C'est pourquoi, la plateforme a des liens étroits et durables avec le secteur industriel dans différents domaines d'applications (Limagrain, Syngenta, Rijk Zwaan, Symrise). EPITRANS est leader en Europe dans le domaine de la recherche translationnelle de par la richesse de ses collections de mutants (260 000 lignées) et par la diversité des espèces cultivées disponibles (13 au total). La plateforme est aussi reconnue Infrastructure Scientifique Collective (ISC) par l’INRA et possède le label IBISA (Infrastructures en Biologie Santé et Agronomie). Contact : Marion Dalmais (marion.dalmais@inra.fr); Abdel Bendahmane (abdelhafid.bendahmane@inra.fr) ; Fabien Marcel (fabien.marcel@inra.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
December 15, 2019 12:46 PM
|
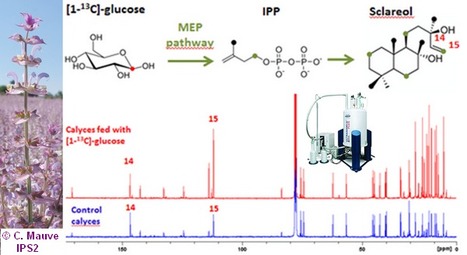
La Plateforme Métabolisme Métabolome de l’Institut des Sciences des Plantes Paris-Saclay (IPS2) est un plateau technique de pointe vous permettant de réaliser vos analyses avec un large panel de techniques complémentaires : GC/MS, LC/MS/MS, RMN, EA/IRMS et chromatographie liquide ou ionique. Des experts sont présents pour optimiser de nouveaux développements pour des problématiques liées au métabolisme primaire ou spécialisé, notamment dans le domaine du végétal. Elle joue un rôle important dans la transmission du savoir sur ces technologies de pointe par son intervention dans des formations initiales (du L3 au Master UPSaclay) et continue (CNRS). Rattachée à l’Ecole Universitaire de Recherche Saclay Plant Sciences – Graduate School of Research (EUR SPS-GSR), la plateforme s’intègre également dans une collaboration étroite inter-plateformes : Saclay Plant Omics (SPOmics) au sein de l’Institut des Sciences des Plantes Paris-Saclay (IPS2). Exemple choisi d’application : Vers une ingénierie métabolique des plantes à parfums : Le sclaréol, de la famille des diterpènes, est un métabolite spécialisé produit par la sauge sclarée (Salvia sclarea, Lamiacées) qui présente un fort intérêt pour l’industrie de la parfumerie mais un rendement de production faible. Nous avons développé une stratégie de marquage isotopique monitorée par de la résonnance magnétique nucléaire du carbone-13 (RMN 13C) afin d’identifier la voie de biosynthèse de ce métabolite d’intérêt. En effet, dans la littérature, deux voies de synthèse sont décrites : une voie cytosolique (MVA) et une voie plastidiale (MEP). L’attribution précise 13C et la quantification pour chaque carbone du sclaréol a permis de mettre en évidence l’origine des atomes de carbone dans la molécule et la voie métabolique utilisée. L’absorption d’une source de carbone marqué régiosélectivement ([1-13C] –glucose) par des tiges d’inflorescence de sauge sclarée conduit à la formation de sclaréol avec un profil isotopique propre à l’utilisation de la voie plastidiale. Ces résultats mettent en évidence des pistes prometteuses pour augmenter, par des techniques d'amélioration génétique ciblée, la production d'une molécule d’intérêt majeur pour la parfumerie, et ont fait l’objet d’une communication orale récente (C. Chalvin, Isotope labeling and mass spectrometry imaging to understand sclareol production in clary sage, Plant Metabolic Engineering (Lucca, Italy, june 2019)). La Plateforme Métabolisme Métabolome de l’Institut des Sciences des Plantes Paris-Saclay (IPS2) offre des services d'analyse métabolomique ou isotopique et sert de support aux recherches sur les métabolismes primaires (carboné, azoté et soufré) et secondaires des plantes et leurs réponses aux contraintes environnementales. Notre double spécialité permet de combiner la métabolomique et la fluxomique pour une meilleure compréhension des réseaux métaboliques au sein de la cellule végétale. Nos outils permettent d’effectuer le phénotypage métabolique rapide des lignées végétales afin d’interpréter les conséquences des mutations génétiques spécifiques, de déterminer l’efficacité d'utilisation du carbone, de l'azote et de l'eau ainsi que les flux métaboliques dans les organes végétaux. Ces recherches s’inscrivent dans un objectif d’optimisation du métabolisme végétal vers la production de matière première utilisable dans l’agroalimentaire avec un apport d’engrais minimum. Au-delà des collaborations locales, notre plateforme est ouverte aux laboratoires publics, privés, locaux, nationaux ou internationaux. Elle offre ses services dans le cadre de collaborations ou de prestations. Dans les deux cas, la plateforme offre une expertise allant du design expérimental aux analyses statistiques des données. Contacts : Bertrand Gakière (bertrand.gakiere@ips2.universite-paris-saclay.fr), Françoise Gilard (francoise.gilard@ips2.universite-paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
December 1, 2019 5:05 PM
|
Le réseau de Centres de Ressources Biologiques et de collections « ressources environnementales » BRC4Env (Biological Resource Centres for the Environment) a été créé en 2015 dans le cadre de l’Infrastructure de Recherche « Ressources Agronomiques pour la Recherche » (AgroBRC-RARe), dont il constitue le pilier « Environnement » (Mougin C. et al, Environmental Science and Pollution Research 2018). Rattaché à l’UMR ECOSYS, le pilier rassemble des ressources biologiques et/ou génomiques utiles aux recherches dans le champ de l’agroécologie, et les mobilise dans le cadre de programmes de recherche portant la santé des écosystèmes, des animaux et de l’homme. BRC4Env collecte, conserve, sécurise, caractérise et met à disposition les ressources biologiques issues de l'environnement (matériel biologique et données associées), de manière sécurisée et avec une traçabilité optimale. Ces ressources biologiques sont échantillonnées à partir de sols, de sédiments, d'eaux, d'écosystèmes agricoles, et conservées dans des collections organisées. Elles concernent les communautés microbiennes, ainsi que les ressources animales et végétales dont la gestion ou la propagation ne dépendent pas directement d'une intervention humaine. Ces ressources sont utilisées pour i) décrire, documenter et caractériser les différentes composantes de l'environnement, ii) développer des procédés à des fins de biocontrôle des ravageurs des cultures, et iii) comprendre les interactions entre différents types d'hôtes et de parasites. BRC4Env soutient la recherche fondamentale et appliquée et rend ses ressources disponibles pour les chercheurs du monde académique, mais aussi de l'industrie et de la société civile (ONG...). Les centres de ressources biologiques et collections de BRC4Env s'appuient sur des plateformes analytiques et des dispositifs d'accès aux écosystèmes. Ils mettent en œuvre le protocole de Nagoya. Par ailleurs, BRC4Env coordonne l’animation et le partage de connaissances, de compétences et de ressources entre ses différents Centres de Ressources Biologiques (CRBs) et collections. Il contribue également à l’animation scientifique et technique au sein de RARe en partenariat avec les piliers plantes, animaux, microorganismes et forêts. Les CRBs et collections de BRC4Env acquièrent progressivement différentes labellisations (Ibisa) ou certifications (ISO9001:2015), ce qui traduit leur haut niveau de prestation. Outre le partenariat de recherche avec le monde académique, les CRBs et collections de BRC4Env font preuve d’innovation dans le cadre de partenariats avec des entreprises privées. Ainsi, le laboratoire Diversité, Génomes & Interactions Microorganismes-Insectes (DGIMI) de l’INRA développe avec la société Nosopharm de nouvelles classes d’agents antimicrobiens pour le traitement des infections nosocomiales pharmaco-résistantes. Les campagnes de criblages ont porté sur des collections de bactéries symbiotiques des nématodes entomopathogènes uniques au monde, hébergées par l’INRA. Ce partenariat donnera suite à des dépôts de brevets et des publications scientifiques. Contact : Christian Mougin (christian.mougin@inra.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
November 16, 2019 5:30 PM
|
La Plateforme de Spectroscopies Vibrationnelles (CEA/DRF/JOLIOT, I2BC - UMR CEA/CNRS/UPSay) offre ses services à des équipes de recherche françaises et internationales. Les spectroscopies vibrationnelles sont basées sur l’interaction de la lumière sur la matière, et permettent d’obtenir des spectres (Raman, FTIR) qui sont le reflet des vibrations des liaisons présentes dans la molécule. Les spectres obtenus donnent des informations sur son identité chimique, sa structure moléculaire et ses interactions avec son environnement. Exemple choisi d’application : Grâce à leurs états électroniques excités, les chlorophylles sont responsables de l’absorption de l’énergie solaire, son transfert sous forme d’excitation vers les centres réactionnels, et sa traduction en potentiel chimique. Afin d’assurer le rendement quantique quasi-unitaire de ces opérations, les protéines photosynthétiques hôtes contrôlent les propriétés électroniques de ces cofacteurs chlorophylliens, ainsi que leur géométrie (distance et orientation mutuelles). Malgré de nombreuses études sur la fonction des protéines photosynthétiques, les paramètres susceptibles de moduler les propriétés électroniques des chlorophylles restent peu connus. Tout récemment, et en collaboration avec deux équipes internationales (R. Croce, VU University Amsterdam; C. Yang, University of Chinese Academy of Sciences, Beijing), la Plateforme de Spectroscopies Vibrationnelles a identifié la protéine antenne majeure chez les plantes vertes, appelée LHCII, comme candidat d’étude. En effet, les spectres d’absorption électronique de cette protéine dépendent du protocole utilisé pour sa préparation. Par des mesures d’absorption électronique à une température de 4,2 K, nous avons montré que ces différences correspondent au décalage vers le rouge des pics d’absorption de deux molécules de chlorophylles a au sein de la protéine. Ensuite, la spectroscopie de diffusion Raman de résonance nous a permis de lier ce décalage à la formation de ponts hydrogène de la protéine avec les groupements ketos des chlorophylles impliquées. Cette relation, entre formation de liaisons hydrogène et position des niveaux électroniques de la chlorophylle, qui a été confirmée par comparaison avec d’autres protéines antennes, révèle un mécanisme moléculaire majeur pour l’optimisation des transferts d’énergie dans la photosynthèse (Llansola-Portoles MJ et al., BBA 2019). La Plateforme de Spectroscopies Vibrationnelles fait partie des "Plateformes de Biophysique de Saclay". Cette plateforme propose des spectroscopies avancées comme la "Spectroscopie Raman de résonance" et la "Spectroscopie infra-rouge à transformée de Fourier" (FTIR). Les équipements « Raman » comprennent 7 spectromètres, avec plusieurs accessoires, et opèrent sur de larges gammes de température (thermostats 273-320 K, cryostats 4-250 K). Le laboratoire est spécialisé dans l’étude des propriétés physico-chimiques des cofacteurs pigmentés en biologie (caroténoïdes, chlorophylles, hèmes, flavines…), et inclut des études in vivo de réactions biochimiques photo-induites et régulatrices. Les analyses sont réalisables sur des échantillons de toutes formes physiques (liquide, poudre, gel, solide…), la seule limitation étant l’intensité du signal mesuré (la présence de molécules pigmentées est nécessaire pour des mesures en milieux complexes) [Exemples récents : processus régulateurs dans les membranes photosynthétiques in vivo (feuilles entières et micro-organismes), structure moléculaire des caroténoïdes et opsines dans la rétine humaine ex vivo]. Les équipements « FTIR » comprennent 4 spectromètres, avec cellule à transmission, accessoires d’ATR, cellule liquide thermostabilisée, et cryostats. Le laboratoire est spécialisé sur la spectroscopie FTIR différentielle, résolue en temps, et à basse température, avec une compétence pour l’étude des réactions biochimiques et photo-induites. Les analyses sont réalisables sur des échantillons de plusieurs formes physiques (liquides, poudres, solutions, protéines) avec la possibilité d’étudier des liaisons métal-métal et métal-protéine. La plateforme « FTIR » permet de suivre des mécanismes de réactions chimiques (Exemples récents : études des réactions biochimiques dans des membranes bioénergétiques in vivo). Cette plateforme fait partie d’un ensemble de plateformes de Biophysique de l’I2BC, labélisées IBISA qui comprend i) la résonance paramagnétique électronique (RPE et RPE haut-champ) [I2BC / Plateforme de résonance paramagnétique électronique (RPE)] pour étudier les radicaux organiques et métaux dans des mécanismes réactionnels ou dans des études structurales ; ii) la microscopie de fluorescence à super-résolution [I2BC / Plateforme de microscopie de fluorescence a super-résolution], qui permet d’obtenir des images de fluorescence avec une résolution de 50 nm en cellules entières ; iii) la spectroscopie électronique dans le proche UV, Vis et proche IR [I2BC / Plateforme de spectroscopies électroniques] avec une sensibilité et une résolution temporelle sans précédent et iv) la spectroscopie vibrationnelle [I2BC / Plateforme de spectroscopies vibrationnelles] qui permet notamment l’étude des propriétés physico-chimiques des cofacteurs pigmentés en biologie. Contact : Andrew Pascal (andrew.pascal@i2bc.paris-saclay.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
November 4, 2019 11:25 AM
|
FOCUS PLATEFORME : Meet My Platform | Sciences de la Vie #2 : Mardi 28 Avril 2020 : SAVE THE DATE !
Genopole et l’Université Paris-Saclay organisent le mardi 28 avril 2020, la deuxième édition de MeetMyPlatform | Sciences de la Vie, en partenariat avec le Crédit Agricole Consumer Finance, qui nous accueillera dans ses locaux à Massy. Nous vous invitons d’ores et déjà à bloquer cette date dans vos agendas ! SAVE THE DATE ! MeetMyPlatform | Sciences de la Vie est une journée de rencontres entre plates-formes et entreprises, organisée par Genopole et le Département Sciences de la Vie de l’Université Paris-Saclay. Son objectif est double : rassembler et donner de la visibilité à la communauté des plates-formes de l’Université Paris-Saclay et permettre aux acteurs de la recherche mais aussi entreprises présentes lors de cet évènement, de les découvrir ou de renforcer des liens déjà existants. Pour mémoire, la première édition de MeetMyPlatform | Sciences de la Vie s’était tenue le 31 janvier 2019 - un jour de neige ! – et avait su rassembler sur le plateau de Saclay près de 300 participants : ingénieurs et techniciens plates-formes, acteurs de la recherche académique mais aussi représentants de grands groupes, de PME/ETI et de Start-up/TPE. Ces derniers ont particulièrement apprécié la convivialité de l’événement et la facilité d’obtention d’informations pertinentes dans leurs domaines d’activités. Les membres des plateformes ont, quant à eux, apprécié ce mode de communication et de présentation de leurs équipements, expertises et savoir-faire, ainsi que les moments d’échanges privilégiés (ateliers) qui leur étaient consacrés en deuxième partie de journée pour notamment renforcer ou structurer de nouveaux réseaux thématiques au niveau sud-francilien. RV le mardi 28 avril 2020 pour une deuxième édition ! Vous souhaitez en savoir plus sur MeetMyPlatfom, cliquer ICI pour une présentation en 70 secondes chrono ! MeetMyPlatform | Sciences de la Vie s’inscrit naturellement dans la dynamique continue de mise en visibilité des compétences, expertises et équipements des plates-formes de l’Université Paris-Saclay et ceci notamment via son portail PlugInLabs Université Paris-Saclay. Contacts : Frédéric Dollé (frederic.dolle@cea.fr), Marion Le Devedec (marion.ledevedec@universite-paris-saclay.fr), Jessica Pericaud (jessica.pericaud@universite-paris-saclay.fr) et Julien Picot (julien.picot@genopole.fr)

|
Scooped by
Life Sciences UPSaclay
October 20, 2019 5:44 PM
|
L’identification de petites molécules modulatrices de phénomènes biologiques (aussi nommées sondes biologiques) peut se faire par une approche de type criblage sans a priori. Le principe est simple : Il s’agit de tester un grand nombre de molécules vis à vis d’un système biologique et d’étudier la réponse à ces molécules. Le système biologique peut être simple (un récepteur, une enzyme, un transporteur) ou complexe (une cellule) ; la cible de ces sondes peut ne pas être connue dans ce cas ! A titre de « success story », notre plateforme de chimie combinatoire et criblage à haut débit (CCCHD, Service de Chimie Bioorganique et de Marquage du CEA, Institut Joliot, CEA/Saclay, Gif-sur-Yvette) a, en collaboration avec une équipe de notre institut (Service d’Ingénierie Moléculaire des Protéines, équipe Daniel Gillet) mis en place, il y a quelques années, un tel criblage dans un contexte bien particulier, le bioterrorisme. L’objectif de ce criblage était de découvrir des molécules capables d’inhiber l’action délétère de la ricine sur des cellules humaines, et surprise, ce sont tout d’abord trois composés (hits) ayant une action protectrice qui ont pu être identifiés à partir d’une collection de 20.000 molécules (Stechmann et al, Cell 2010) L’histoire ne s’arrête pas là puisque chacun de ces trois composés a ensuite été optimisé par des approches de chimie médicinale permettant d’obtenir des activités inhibitrices améliorées d’environ mille fois (leads). Le mode d’action de ces composés a également été élucidé, permettant d’élargir le spectre de leur activité à un grand nombre de pathogènes (virus, bactéries….), ouvrant ainsi l’accès vers des candidats médicaments à large spectre (Gupta et al, Chemico-Biological Interactions 2017). Plus récemment, l’une de ces molécules a été à l’origine de l’identification de la cible cellulaire, permettant d’expliquer son action protectrice et son mode d’action inédit (collaboration Institut Curie, équipe Ludger Johannes). La connaissance de cette cible va permettre d’aller vers de nouvelles familles chimiques encore plus efficaces. NB : ces travaux ont bénéficié du soutien du programme interministériel de R&D contre les risques NRBCE. La plateforme de chimie combinatoire et criblage à haut débit combine les équipements et expertises nécessaires pour la réalisation de criblages biologiques à haut débit ainsi que la préparation de chimiothèques ciblées. Elle assure également l’optimisation des composés actifs les plus prometteurs (« touches » ou « hits »). La plateforme est aujourd’hui partie intégrante de C@PS (pour criblage sur le plateau de Saclay), labélisée par IBISA depuis fin 2018. C@PS agrège également les compétences des plateformes i) CIBI (ICSN, CNRS, Gif-sur-Yvette) pour les mesures de cytotoxicité, de criblage d’interactions protéines/ligands et criblage in ovo, ii) CIBLOT (Faculté de Pharmacie, Université Paris sud, Châtenay-Malabry) pour les mesures d’interactions protéines/ligands par technologie alpha-screen et la quantification de paramètres cellulaires, et iii) CTPF (ICSN, CNRS, Gif-sur-Yvette) pour les mesures d’interactions protéines/ligands par thermal shift assay et d’analyses de transcriptomes par PCR quantitative. Contact : Jean-Christophe Cintrat (jean-christophe.cintrat@cea.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
October 6, 2019 12:45 PM
|
L'exploration multi-échelles du vivant : un défi relevé depuis plus de 10 ans par la Plateforme Imagerie et Cytométrie (PFIC) de l'UMS 23/3655. Observer la dynamique des réponses moléculaires et des interactions cellulaires est l'un des enjeux de l'imagerie moderne pour améliorer la compréhension des processus pathologiques ou de réparation. Les systèmes modernes d'imagerie photonique permettent d'aborder ces questions ex-vivo, mais l'approche in-vivo dans un contexte physiologique réel se heurte à 2 obstacles majeurs : l'accès aux tissus cibles à haute résolution et le suivi dans le temps de l'évolution des systèmes biologiques sur un même tissu. Une solution simple et efficace appelée « 'chambre dorsale » permet d’accéder à l'information intra-vitale multi-échelles de la molécule à l'organe. Un modèle optimisé a été développé par la PFIC avec le soutien de la plate-forme d'évaluation préclinique de UMS 23/3655. Ce dispositif implanté – véritable fenêtre d’observation du vivant – est parfaitement toléré par les spécimens vivants sur plusieurs mois et permet de réaliser une imagerie dynamique in vivo à l'échelle subcellulaire et sur le long terme. La stabilité de la chambre facilite l'imagerie 3D sur des champs très larges et permet de combiner les échelles d'observation, de la cellule isolée à l'organe et son environnement. Compatible avec tous les microscopes (Vidéo, confocal, spinning disk, multiphoton) cette chambre permet également la photomanipulation (FRET, FRAP, Photoconversion) in-vivo, pour l'étude dynamique des interactions moléculaires et cellulaires ou encore la thermomanipulation pour la création de modèles ischémiques, par exemple. Ces chambres également conçues pour l'imagerie multi-modalités sur modèle murin sont compatibles avec les imageries Ultrason, X-Ray et IRM. Conçues initialement pour l'imagerie des tumeurs et l’évaluation préclinique de traitements, ces chambres qui ont fait l’objet d’un dépôt de brevet (WO2015135992A1), aujourd’hui commercialement disponibles (CREOBS, info@creobs.com), sont utilisées aujourd'hui localement, par des équipes du campus Paris-Saclay (UMR U1179 (INSERM/UVSQ), UMR-S 1197 (INSERM-UPSud)) mais aussi par de nombreuses équipes au niveau national pour explorer des domaines très différents comme l'ischémie, l'inflammation ou encore la réparation musculaire. Pour ces applications également, cet outil unique a ouvert de nouvelles perspectives pour la caractérisation d’évènements dynamiques moléculaires et cellulaires à haute résolution dans un contexte d’organes ou de tissus vivants. Pour optimiser l'implantation de la technique dans les laboratoires intéressés, la plateforme met en place en 2020, une formation nationale de la chirurgie des chambres à l'imagerie. Vous souhaitez en savoir plus ? cliquer ici La Plateforme Imagerie et Cytométrie (PFIC, UMS AMMICA) est une plateforme de service et de développement à l’interface des programmes de recherche fondamentale, translationnelle et clinique. Son objectif est de mettre à disposition des équipes de recherche internes à Gustave Roussy, des équipes académiques externes, ainsi qu’aux industriels, un pôle d’expertise en cytométrie en flux, tri cellulaire, imagerie photonique, imagerie multiphotonique moléculaire et cellulaire, imagerie tissulaire et imagerie photonique du petit animal. Contact : Corinne Laplace-Builhé (Corinne.LAPLACE@gustaveroussy.fr) Plug In Labs Université Paris-Saclay : cliquer ici

|
Scooped by
Life Sciences UPSaclay
September 22, 2019 10:11 AM
|
L’aimant du projet Iseult, en installation à Neurospin (CEA/DRF/JOLIOT, plateforme NEUROSPIN / Imagerie in vivo chez l’homme, ex vivo et in vitro, IRM 11,7T) a atteint son champ nominal de 11,7 teslas (T) le 18 juillet 2019. Il s’agit d’un record mondial pour un aimant IRM humain corps entier, qui vient couronner des années de R&D, à la pointe de l’innovation dans le domaine des aimants supraconducteur. En cours d’installation depuis juillet 2017, le colosse magnétique est désormais pleinement opérationnel. Les équipes ont dû pour cela relever tous les défis que représente la mise en œuvre d’un tel équipement : i) connecter l’aimant aux équipements auxiliaires déjà installés et testés (usine cryogénique, alimentations électriques, système de contrôle-commande) ; ii) atteindre une température homogène de 1,8 K : l’aimant doit être refroidi à 1,8 K (-271°C), température à laquelle l’hélium est dans un état physique particulier, dit « superfluide ». À ce niveau de température, le conducteur qui compose l’aimant (alliage niobium-titane) n’oppose aucune résistance au courant électrique, évitant ainsi toute dissipation du courant et tout échauffement : il est à l’état supraconducteur ; et enfin iii) monter par palier pour atteindre 11,7 T : une fois le conducteur refroidi à sa température nominale, il a fallu ensuite injecter progressivement le courant dans l’aimant. Cette « montée en puissance » s’est réalisée en plusieurs étapes, avec de nombreux essais électriques et magnétiques, ainsi que des tests des procédures d’arrêt d’urgence. En tout, 1300 procédures prévues pour détecter l’apparition de défauts potentiels ont été testées. A noter que pour atteindre son champ magnétique nominal, l’aimant est alimenté par un courant de 1500 ampères, et les bobines de conducteur sont en permanence refroidies par 7000 litres d’hélium à l’état superfluide. La fabrication de ce prototype de 132 tonnes, 5 m de longueur, 5 m de diamètre extérieur et 90 cm de diamètre intérieur, aura demandé six ans dans les usines de Alstom – devenu GE – à Belfort et près de deux ans de travaux d’installation et de tests (Le Bihan and Schild, Supercond. Sci. Technol. 2017). Au cours des prochains mois, les équipements nécessaires pour réaliser les images cérébrales seront installés autour de l’aimant ainsi que dans son tunnel central, pour en faire un scanner IRM humain capable de sonder le cerveau à des précisions jamais atteintes, au bénéfice de la recherche fondamentale, des sciences cognitives et du diagnostic des maladies neurodégénératives. La plateforme NEUROSPIN / Imagerie in vivo chez l’homme, ex vivo et in vitro, IRM 11,7T a pour objectifs scientifique de i) visualiser le cerveau humain à une résolution spatiale inégalée jusqu'ici : échelle « mésoscopique » (~100 microns) ; ii) découvrir de nouveaux contrastes, et iii) explorer les possibilités de l'imagerie moléculaire à très haute sensibilité. Ceci sera rendu possible grâce aux caractéristiques exceptionnelles du scanner IRM 11.7 T, résumées ci-contre : (1) aimant opérant en mode non-persistant pour une meilleure stabilité temporelle ; (2) bobine principale composée de bobines individuelles de conception «en double galettes » pour une meilleure homogénéité spatiale ; (3) deux bobines de blindage actif ; (4) champ magnétique : 11.75 T ; (5) énergie stockée : 338 MJ ; (6) courant : 1483 A ; (7) inductance : 308 H ; (8) longueur Cryostat : 5 m ; (9) diamètre extérieur : 5.2 m ; (11) diamètre interne du tunnel de l'aimant : 90 cm ; et (12) poids : 132 tonnes. Contact : cecile.lerman@cea.fr ou nicolas.boulant@cea.fr PluginLabs Université Paris-Saclay : cliquer ici
|



 Your new post is loading...
Your new post is loading...