Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
July 11, 5:43 AM
|
Depuis sa création, le Genoscope – Centre National de Séquençage participe à des projets collaboratifs à fort impact scientifique en fournissant à la communauté scientifique toute l’expertise et les capacités de production et d’analyse de données indispensables à ces projets. Depuis sa contribution majeure, il y a près de 20 ans, au projet Human Genome en publiant la séquence complète du chromosome 14 (Heilig et al., Nature 2003), la plateforme de séquençage située au cœur du biocluster Genopole installé à Évry-Courcouronnes a réalisé le séquençage de nombreuses plantes (vigne, riz, blé…), d’animaux (tétraodon, anophèle…), de champignons (truffe, champignons pathogènes du colza et de la vigne) et de plus d’une centaine de procaryotes. Grâce à sa collaboration avec le Très Grand Centre de Calculs du CEA, situé lui-aussi en Essonne, le Genoscope - CNS a permis de traiter de très grands ensembles de données en utilisant l’informatique haute performance de pointe. Ainsi, depuis sa création, le Genoscope-CNS a contribué à plus de 750 publications et ceci grâce à sa plateforme de séquençage. Très récemment, le Genoscope - CNS a choisi de s’aligner sur la stratégie et les engagements de l'Union Européenne en matière de biodiversité pour 2030 tels que la lutte contre le déclin des pollinisateurs, l'amélioration de l'état de conservation des espèces et des habitats clés, la promotion de la restauration écologique des systèmes forestiers et des rivières, et la minimisation des impacts des espèces invasives. Accompagné d’autres partenaires européens, le Genoscope – CNS participe activement à la recherche en génomique de la biodiversité en Europe visant à étayer la conservation de celle-ci et sa biosurveillance basée sur l'ADN par la génération de génomes de référence de haute qualité provenant de toutes les formes de vie afin d’établir un Atlas européen des génomes de référence (ERGA). L’initiative ERGA est une réponse scientifique paneuropéenne aux menaces actuelles qui pèsent sur la biodiversité. Les génomes de référence fournissent l’aperçu le plus complet des bases génétiques qui constituent chaque espèce et représentent une ressource puissante pour comprendre le fonctionnement de la biodiversité. Alors qu’environ un cinquième des quelques 200 000 espèces européennes sont menacées d’extinction, nous devons agir rapidement et ensemble pour générer à grande échelle des ressources génomiques complètes de haute qualité. Parallèlement à cet important programme européen, le Genoscope - CNS a récemment lancé ATLASea, l’un des projets et équipements prioritaires de recherche (PEPR) exploratoires sélectionnés dans le cadre de France 2030. Copiloté par le CNRS et le CEA et financé à hauteur de 41,3 millions d’euros sur 8 ans, le programme national ATLASea a pour objectif de séquencer le génome de 4500 espèces marines contenant, par conséquent, des millions de gènes encore inconnus à ce jour en visant spécifiquement l’analyse des génomes d'un large éventail d'espèces marines dans tous les groupes eucaryotes présents au sein du littoral français, plus précisément dans sa zone économique exclusive qui s’étend sur plus de 11 millions de Km2, et qui en fait la plus grande du monde, devant celle des Etats-Unis et de l’Australie. Autre actualité importante de la plateforme de séquençage : l’arrivée, courant 2023, d’un séquenceur Revio (PacBio) (en photo) dédié au séquençage des longs fragments et capable de séquencer 1300 génomes humains par an en profondeur 30X, venu renforcer le parc de séquenceurs de la plateforme déjà composé d’un NovaSeq 6000 (Illumina) acquis en 2018, deux MiSeq (Illumina), un PromethION (Oxford Nanopore Technologies), un GridION (Oxford Nanopore Technologies), un MinION Mk1C (Oxford Nanopore Technologies) et un DNBSEQ-G400 (MGI Tech). Pour obtenir des renseignements sur les modalités d’accès de la plateforme de séquençage, n’hésitez pas à contacter Pedro Oliveira ! Aussi, le 24 mai 2021 (déjà !), la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? Contact : Pedro OLIVEIRA (pcoutool@genoscope.cns.fr) Plug In Labs Université Paris-Saclay : cliquer ICI GENOSCOPE / Plateforme de Séquençage - Genopole. Le Genoscope s’est orienté vers l’étude de la biodiversité, à travers le séquençage de novo de grands génomes complets, de métagénomes complexes et plus généralement de collections d’ADN nécessitant une approche de séquençage à très grande échelle. L’équipe du laboratoire de séquençage s’implique dans la prise en main des projets à partir de la réception des échantillons jusqu’à la validation des séquences brutes. Le séquençage est assuré par des séquenceurs très haut débit de deuxième et troisième génération. Ces équipements high-techs permettent d’atteindre un débit autorisant le séquençage de génomes entiers dans un intervalle de temps limité. A propos de Genopole. Premier biocluster français dédié à la recherche en génomique et aux biotechnologies appliquées à la santé et à l’environnement, Genopole rassemble 66 entreprises de biotechnologies, 17 laboratoires de recherche, 24 plateformes technologiques et plateaux techniques mutualisés, ainsi que des formations universitaires (université d’Évry, Paris-Saclay). Son objectif : soutenir les politiques nationales de réindustrialisation et de souveraineté sanitaire, créer et soutenir les entreprises de biotechnologies et le transfert de technologies vers le secteur industriel (notamment pour décarboner la production), favoriser le développement de la recherche dans les sciences de la vie, développer des enseignements de haut niveau dans ces domaines. Situé à Évry-Courcouronnes, Genopole est principalement soutenu par l’État, la Région Ile-de-France, le Département de l’Essonne, l’agglomération Grand Paris Sud, la Ville d’Évry-Courcouronnes et l’AFM-Téléthon. Pour obtenir plus de renseignements sur les plateformes labellisées par Genopole, ainsi que sur les équipements mutualisés accessibles à la communauté scientifique francilienne, vous pouvez aussi contacter Julien Picot (Julien.Picot@genopole.fr).

|
Scooped by
Life Sciences UPSaclay
July 9, 5:33 AM
|
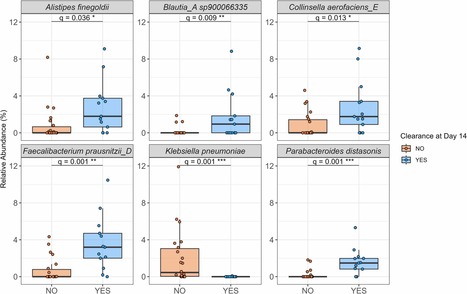
Dans un contexte d’augmentation continu de la résistance aux antibiotiques, un travail coordonné à l’hôpital Raymond Poincaré (AP-HP/UVSQ/UPSaclay, Garches), en collaboration avec Vedanta BioSciences (Boston, USA) et la banque de fèces de Leiden (Pays-Bas), a cherché à étudier l’intérêt de la transplantation de microbiote fécale (TMF) dans la lutte contre l’antibiorésistance. En effet, la TMF ayant déjà démontré son efficacité dans le traitement des infections digestives à Clostrioides difficile, l’objectif principal était la décolonisation digestive de patients porteurs d’Entérobactéries Productrices de Carbapénémases (EPC) à 14 jours de greffe fécale. L’objectif secondaire était l’étude de son efficacité à 1 mois, 3 mois et analyse métagénomique de l’impact d’un donneur sélectionné sur l’efficacité de la TMF. Ce travail publié dans Journal of Infection repose sur une étude multicentrique conduite de 2015 à 2019 en France ayant inclus 20 patients âgés de plus de 18 ans ayant bénéficié d’une TMF dans cette indication. Les patients étaient principalement porteurs de K. pneumoniae de type OXA-48 (75%), dont 15% avaient des antécédents d’infection à bactérie multi-résistante. Après greffe fécale, 20% des patients étaient décolonisés à J15 et jusqu’à 40% à M3. Comparativement au groupe contrôle (n=82) surveillé cliniquement et n’ayant pas bénéficié de greffe fécale, l’analyse des courbes de Kaplan-Meier n’a pas révélé de différence significative en termes de décolonisation (p=0.9). A noter que 2/2 patients colonisés et qui présentant des infections urinaires à répétitions à Entérobactéries de type BLSE ont été considérés indemnes de symptômes à plus de 12 mois. De plus, l’étude métagénomique a mis en évidence que parmi les individus décolonisés après greffe fécale, la réponse n’était pas liée à un donneur particulier. De même les patients ayant répondu favorablement à la TMF avaient une quantité moins importante d’EPC à la base (effet inoculum), et présentaient une richesse et un score de diversité alpha significativement plus élevé à J14 (p<0.05). Enfin, cette étude a mis en évidence une signature bactérienne associée au succès de la greffe, à savoir les patients présentant en plus grande quantité à postériori 5 taxons : Faecalibacterium prausnitzii, Parabacteroides distasonis, Collinsella aerofaciens, Alistipes finegoldii et Blautia_A sp900066335 (q<0.01). Bien que cette étude ne démontre pas d’effet significatif de la TMF en population « tout venante » colonisée à EPC, le transfert de bactéries sélectionnées au sein d’un consortium visant à traiter des patients non seulement porteurs mais infectés à des bactéries multi-résistantes reste une piste sérieuse à étudier. Légende Figure: taxons bactériens associés à la décolonisation. Contact : benjamin.davido@aphp.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 6:11 AM
|
Dans une étude publiée dans Molecular Medicine, coordonnée par l’équipe du Pr Frédéric Charbonnier (UMR-S 1124, Université Paris Cité) et associant le Laboratoire de biologie de l’exercice pour la performance et la santé (LBEPS) (UPSaclay/UEVE/IRBA, Evry), les scientifiques ont étudié l’importance de l’individualisation de la prise en charge physiothérapeutique dans le cadre du diabète de type 2 (DT2). En effet, si l'activité physique est recommandée pour traiter le DT2, ses effets peuvent varier considérablement d’un individu à l’autre sans en connaître les raisons. Les auteurs ont posé comme postulat que la réponse à l’exercice dépend du statut métabolique musculaire initial, statut très peu étudié dans le cadre des DT2 et de leur pluralité. Pour tester cette hypothèse, la signature métabolique du muscle quadriceps a été déterminée, en utilisant une combinaison de spectroscopie en RMN et de RT-qPCR, dans plusieurs modèles de souris DT2 suivant le sexe, le fond génétique (129S1/SvImJ, C57Bl/6J), et l’étiologie (alimentaire, toxique ou génétique). Trois modèles de souris présentant des signatures métaboliques musculaires uniques ont été identifiés puis soumis à trois programmes différents d’exercice de nage, conçus pour répondre à chaque spécificité métabolique. De manière intéressante, ce sont bien les exercices de nage dédiés qui induisent des effets optimaux sur la réduction de la résistance à l'insuline, l’amélioration de l'homéostasie du glucose et les capacités sensorimotrices. Ces travaux montrent que le métabolisme musculaire constitue un élément clef pour la conception d'exercices de précision, avec des implications cliniques certaines. Transférer ces résultats chez l'humain pose le défi de l'analyse individualisée et non invasive du métabolisme musculaire des patients : spectroscopie RMN in vivo ? » Contact : frederic.charbonnier@u-paris.fr ou olivier.biondi@univ-evry.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 8:41 AM
|
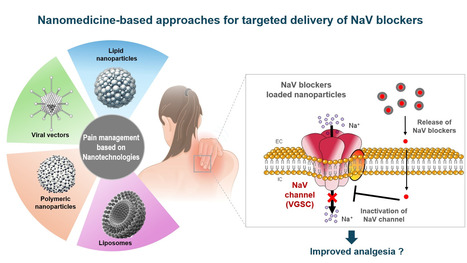
La douleur, une condition complexe et invalidante touchant des millions de personnes dans le monde, représente une préoccupation majeure, particulièrement dans le contexte des douleurs chroniques et la récupération post-opératoire. Dans une revue publiée dans Drug Delivery and Translational Research, l'équipe Nanomédicaments pour le traitement des maladies graves de l'Institut Galien Paris Saclay (UMR 8612 CNRS/UPSaclay, Orsay), présente les approches innovantes, en se focalisant particulièrement sur la nanomédecine, pour gérer la douleur en agissant sur les canaux sodiques voltage-dépendants (VGSC ou canaux NaV). Cette revue décrit tout d’abord la complexité de la douleur et son impact global, puis les différentes cibles de celle-ci en mettant l'accent sur les canaux NaV comme une avenue prometteuse pour la gestion de la douleur dans le but de réduire le recours aux opioïdes. Elle examine le rôle des différents isoformes de ces canaux sodiques, en particulier ceux impliqués dans la nociception. Ensuite, elle expose les différents traitements disponibles utilisant des bloqueurs sélectifs de ces isoformes de canaux NaV, en détaillant leur efficacité dans la gestion de la douleur. Elle aborde également les défis associés à leur distribution systémique et aux effets secondaires potentiels. Enfin, cette revue met en lumière le potentiel de la nanomédecine pour améliorer la biodisponibilité et la délivrance de ces médicaments pour une libération ciblée et prolongée au site de la douleur. Cette avancée ouvre de nouvelles perspectives pour développer des stratégies de soulagement de la douleur plus efficaces et mieux adaptées aux patients, améliorant ainsi leur qualité de vie. Contact : sinda.lepetre@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 9:01 AM
|
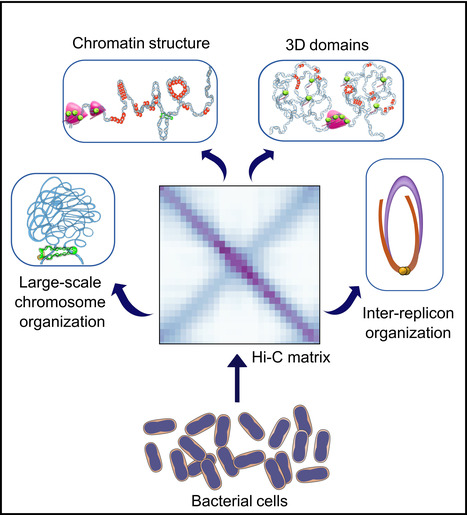
Dans une revue publiée dans Molecular Microbiology, Virginia S. Lioy et ses collaborateurs de l’équipe Conformation and segregation of the bacterial chromosome à l’I2BC (CNRS/CEA/UPsaclay, Gif-sur-Yvette) ont revu les dernières découvertes du Hi-C et discuté des principes de repliement des chromosomes bactériens qui peuvent en être déduits. L'utilisation de Hi-C a non seulement révélé la diversité des architectures 3D des génomes de multiples espèces bactériennes à travers l'arbre phylogénétique, mais aussi les différentes échelles d'organisation des génomes et les mécanismes d'action des facteurs et processus structuraux clés. Les auteurs revisitent ici les travaux réalisés sur ces bactéries et discutent des règles de repliement du génome qui peuvent en être déduites. Contact : virginia.lioy@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
July 11, 4:43 AM
|
Dans une étude publiée dans Pediatric Research, des chercheurs travaillant sur les données de la cohorte française Constances (UMS 011 INSERM/UVSQ/UPSaclay) ont étudié la relation entre une naissance prématurée (avant 37 semaine d’aménorrhée) ou à terme mais avec un petit poids de naissance (i.e. <10ème percentile) et l’état de santé à l’âge adulte. Réalisée sur 30 295 participants, l’étude a confirmé que ces deux facteurs étaient associés à un risque de complications métaboliques plus élevé (+ 35% de syndrome métabolique en cas de naissance prématurée, + 30% de risque de diabète en cas de naissance à terme avec un petit poids de naissance). Néanmoins, l’étude apporte de nouveaux éléments. En effet, les investigateurs ont identifié un excès de risque d’obésité en cas de naissance prématurée (+ 25%) alors que ce risque est réduit en cas de naissance à terme avec un petit poids de naissance (- 17%). Bien qu’elles partagent certaines étiologies, ces résultats suggèrent que la prématurité et la restriction de croissance présentent des mécanismes métaboliques adaptatifs complexes et vraisemblablement différents impliquant pour la première un excès de masse grasse et pour la deuxième des mécanismes d’insulinorésistance plus complexes. Contact : coralie.amadou@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
July 11, 5:19 AM
|
The conference will take place on November 13-15, 2024 at the Institute of Neuroscience Paris Saclay (NeuroPSI), located ~30km south of Paris. Registration opens on the August 1st and closes on October 15th. Abstracts We welcome abstracts submissions from the entire community from trainees to PIs.
There is the possibility for abstracts to be choses for a talk, and, in any case, all presenters will be given ample time for poster presentations and a preview datablitz talk. Minischool As usual, ahead of the conference, on November 12th (afternoon) and 13th (morning), there will be an optional and free Minischool at NeuroPSI. Bear in mind that there will be a limitation to 150 participants to the conference and places will be assigned on a « first come, first served » basis. Registration fees: - Student/Postdoc: 30€
- Permanent researcher/engineer: 60€
- Industry (without booth, /pers): 150€
- Industry (with boot, /pers): 500€ (+80€/suppl. pers.).
See the website for the program and registration The organizing committee:
Anindita Das, Andrew Davison, Thomas Deneux, Mathilde Do Paco, Jean-Marc Edeline, Valérie Ego-Stengel, Isabelle Férézou, Aline Ginois, Nicolas Giret, Chloé Huetz, Tihana Jovanic, Daniel Shulz, Virginie van Wassenhove.

|
Scooped by
Life Sciences UPSaclay
July 10, 5:37 AM
|
L’objectif de cette matinée, coorganisée avec les GS Chimie et HeaDS, sera de vous présenter les différentes formes de partenariats entres les laboratoires et les entreprises, ainsi que les services d’accompagnement proposés par la Cellule des partenariats de l'Université Paris-Saclay. Programme : - 09h00 : Présentation de la politique de partenariat de l’université: cadre administratif-légal
- 09h30 : Rôle de la cellule des partenariats de l'Université et services proposés aux laboratoires
- 10h00 : Revue des différentes formes de partenariats, avec une prise en compte des spécificités de chaque établissement
- 10h30 : Pause-café
- 11h00 : Témoignages issus de trois Graduate Schools GS Chimie ; GS HeaDS ; GS LSH
- 12h30 : Déjeuner & échanges
Le lien de connexion pour la visio sera communiqué prochainement pour celles et ceux qui s'inscrivent. Vous pouvez vous inscrire à l'événement ICI

|
Scooped by
Life Sciences UPSaclay
July 8, 5:05 AM
|
► Donnez votre avis aujourd'hui pour améliorer les partenariats entre les entreprises et le Pôle Universitaire d’Innovation de l’Université Paris-Saclay ! ► Vous êtes intéressé par un partenariat avec le Pôle Universitaire d’Innovation de l’Université Paris-Saclay et ses partenaires ? N’hésitez pas à répondre à cette enquête ! Le Pôle Universitaire d’Innovation (PUI) de l’Université Paris-Saclay regroupe treize membres fondateurs : - l’Université Paris-Saclay ;
- les quatre grandes écoles membres de l’Université que sont CentraleSupélec, AgroParisTech, ENS Paris-Saclay et l’Institut d’Optique Graduate School ;
- les deux universités membres-associés – Université d’Évry et Université de Versailles Saint-Quentin-en-Yvelines ;
- quatre organismes nationaux de recherche – CNRS, INRAE, Inria et Inserm – et
- deux acteurs de l’innovation sur le territoire que sont IncubAlliance et la SATT Paris-Saclay, ainsi que quinze partenaires1.
Ses membres fondateurs souhaitent établir un état des lieux de la qualité de leurs relations avec les entreprises ainsi que leurs besoins afin d’améliorer leurs offres et leurs pratiques envers les entreprises. Nous vous remercions de bien vouloir nous faire connaître votre appréciation ou besoin en répondant à ce questionnaire à titre individuel et non au niveau global de votre entreprise. Compléter le questionnaire Le questionnaire vous prendra environ 15 minutes à compléter
J’ai un projet biotech innovant, Shaker m’apporte les outils pour développer mon projet Pour réussir ma POC - Les clés d’un laboratoire tout équipé
- Une enveloppe pour l’achat de consommables / réactifs
- Accès à des technologies de pointe
Pour créer ma startup - Un coaching par des experts de la bio-innovation
- Un programme ajusté à mes besoins
- Un accompagnement spécifique aux biotechnologies
+ Je rejoins Genopole, 1er biocluster créé en France Postulez à la 15e édition du dispositif Shaker ! Date limite de candidatures : 28 juillet 2024 Je postule ! Shaker est entièrement financé par Genopole (sans prise de propriété intellectuelle ni de part au capital)
Via Life Sciences UPSaclay

|
Scooped by
Life Sciences UPSaclay
July 10, 5:52 AM
|
L’Objet Interdisciplinaire New Frontiers in Cancer lance deux AAPs pour soutenir des projets de recherche interdisciplinaires par le financement de gratifications de stage M2 et/ou allocations doctorales. Pour en savoir plus
La GS LSH propose plusieurs appels à projet au fil de l’eau, auxquels vous pouvez répondre sans contrainte de dates : - « Actions pédagogiques » – soutenir des actions pédagogiques transformantes pour les formations.
Pour accéder au formulaire : ICI - « Mobilité sortante pour les doctorants » – Participation à des Écoles d’été et formations équivalentes (congrès, workshops, etc.)
Pour accéder au formulaire : ICI - « Manifestations scientifiques » – Soutien à l’organisation de manifestations scientifiques (conférences, colloques, webinaire, etc.)
Pour accéder au formulaire : ICI - « Labélisation cours pratiques innovants GS LSH » - Valoriser et soutenir financièrement des unités d’enseignements de M2 proposant une formation fortement adossée à une forte expertise de Recherche.
Pour accéder au formulaire : ICI - AMI « Innovations technologiques en santé » GS LSH & Hub PASREL
Pour en savoir plus : ICI
Via Life Sciences UPSaclay

|
Scooped by
Life Sciences UPSaclay
July 10, 4:47 AM
|
La Société Française d’Écologie et d’Évolution (SFE²), Humanité et Biodiversité (H&B) et la Fondation pour la Recherche sur la Biodiversité (FRB) s’associent afin de promouvoir « l’écologie impliquée » en offrant une bourse de 5000 euros. Cette bourse en hommage à Robert Barbault et Jacques Weber peut être utilisée pour financer une partie spécifique d’un projet plus vaste faisant appel à d’autres sources de financement. Cette bourse récompense des projets d’écologie impliquée définie ici comme un champ de l’écologie qui cherche à résoudre des questions de société autour d’enjeux environnementaux par des projets de recherches associant des personnes du monde académique et des personnes du monde non-académique. Pour plus de précisions sur les bourses SFE²-H&B-FRB, consultez les objectifs, conditions d’éligibilité et critères d’évaluation.
|

|
Scooped by
Life Sciences UPSaclay
July 9, 5:07 AM
|
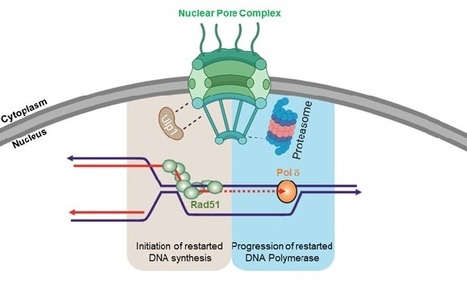
Dans une étude publiée dans Nucleic Acids Research, les scientifiques du laboratoire « Recombinaison, Réplication et Stabilité des génomes » de l’unité « Intégrité du génome, RNA et Cancer » (UMR3348 CNRS/Institut Curie/UPSaclay, Orsay) ont mis en évidence un rôle majeur des pores nucléaires dans la réactivation des fourches de réplication par la recombinaison homologue. De nombreuses études ont démontré que les sites souffrant d’accidents de la réplication de l’ADN sont délocalisés vers la périphérie du noyau, notamment pour s’associer avec les pores nucléaires, des structures macromoléculaires agissant comme des portes pour assurer la communication moléculaire entre le noyau et le cytoplasme. En combinant des approches génétique et génomique chez la levure Schizosaccharomyces pombe, et en collaboration avec des équipes du CRCM de Marseille, de l’université de Wroclaw en Pologne, et de l’université du Sussex en Angleterre, les auteurs démontrent que deux activités enzymatiques, particulièrement enrichies dans l’environnement des pores nucléaires, sont nécessaires à la réactivation des fourches de réplication : la SUMO protéase Ulp1 qui facilite l’activation de la synthèse d’ADN réparatrice et le protéasome qui facilite la progression de cette synthèse d’ADN réparatrice. De plus, ces activités ne se compensent pas et sont contrôlées par des composants différents des pores nucléaires. Ces travaux révèlent l’importance de mobiliser les accidents de la réplication d’un compartiment nucléaire vers l’environnement des pores nucléaires pour faciliter leur réparation, environnement cellulaire plus favorable à la duplication du génome en condition de stress. Contact : sarah.lambert@curie.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 5:56 AM
|
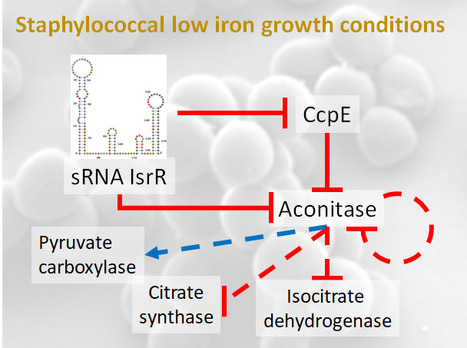
Staphylococcus aureus, responsable d'infections nosocomiales et de septicémies, est la première cause de mortalité bactérienne dans la plupart des pays du monde. Sa pathogénicité repose sur son adaptation à une large gamme de biotopes. La biodisponibilité du fer étant parfois un facteur limitant pour la croissance, les bactéries ont développé des stratégies pour récupérer le fer et réduire son utilisation dans des conditions de croissance appauvries en fer. L'une de ces stratégies implique la modulation des protéines utilisatrices de fer par l'action d'ARN régulateurs non codants (sRNA). Les recherches antérieures de l’équipe de Philippe Bouloc à l’I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) ont révélé l'importance d'IsrR, un sRNA staphylococcique nécessaire à une croissance optimale dans des milieux appauvris en fer et au maintien d'une virulence efficace (Coronel-Tellez et al., 2022). Induit par une carence en fer, IsrR coordonne la répression le l’expression de gènes codant pour les enzymes contenant des noyaux [Fe-S], dont l'aconitase. L'aconitase convertit le citrate en isocitrate. Son gène est régulé positivement par CcpE, un régulateur transcriptionnel activé par le citrate. Dans une nouvelle étude publiée dans Nucleic Acids Research, en collaboration avec Claire Toffano-Nioche de l’équipe RNA Sequence, Structure & Function, l’équipe montre que IsrR inhibe non seulement la traduction de l'ARNm de l'aconitase, mais cible également l'ARNm de ccpE, établissant ainsi une boucle de régulation cohérente (feed-forward). Ce double mécanisme de répression assure un contrôle efficace de la production d'aconitase par l'IsrR. L'aconitase est une protéine connue pour avoir une seconde (moonlighting) activité en l'absence de son cluster [Fe-S] ; elle devient une protéine de liaison à l'ARN (RBP) dotée d'une capacité de régulation. Cette caractéristique est conservée des procaryotes aux eucaryotes. Les auteurs montrent ici que l'activité RBP de l’aconitase de S. aureus permet une diminution de la quantité aconitase et influence l'expression des gènes codant pour des enzymes métaboliques lors de carences en fer. Cette étude révèle le réseau complexe de régulations post-transcriptionnelles gouvernant la production d'aconitase qui permet au Staphylocoque doré de proliférer dans des conditions de carence en fer. A noter que ces conditions sont rencontrées par les bactéries dans l'hôte du fait d'une réaction de défense connu sous le nom d'immunité nutritionnelle, dans laquelle le fer et d'autres nutriments essentiels sont séquestrés pour prévenir les infections. Contact : philippe.bouloc@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 8:33 AM
|
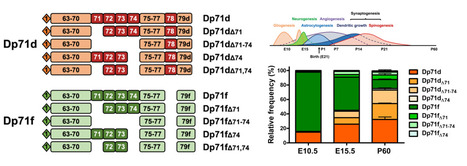
La Dp71 est le plus petit produit du gène DMD (71kDa) mais le plus largement exprimé dans le cerveau. Son absence a été associée à la genèse de déficience intellectuelle dans un sous-groupe de patients souffrant de dystrophie musculaire de Duchenne (DMD). Exprimée dans différents types cellulaires, notamment dans les pieds des astrocytes périvasculaires et des sous-types de synapses, la Dp71 joue des rôles multiples dans le cycle cellulaire, l’homéostasie et la plasticité cérébrale. La Dp71 subit de nombreux épissages alternatifs, principalement dans la partie C-terminale. Une dizaine d’isoformes de la Dp71 ont ainsi été identifiées dans le cerveau, mais leur rôle fonctionnel est encore inconnu. Dans un article publié dans Molecular Neurobiology, l’équipe de Cyrille Vaillend à l’Institut des Neurosciences Paris-Saclay – NeuroPSI (CNRS/UPsaclay, Gif-sur-Yvette), en collaboration avec une équipe Mexicaine (CINVESTAV, Mexico city), a étudié l’expression de ces isoformes de Dp71 au cours du développement du cerveau de la souris et du rat par clonage et RT-PCR, puis par séquençage nanopore en collaboration avec l’Institut de Biologie Intégrative de la Cellule - I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) et Jérome Roger (NeuroPSI). Les résultats obtenus par ces méthodes convergent et démontrent qu’une isoforme dépourvue de l’exon 78 est initialement majoritaire dans le cerveau fœtal, puis cinq autres isoformes apparaissent graduellement et montrent des variations d’expression au cours du développement postnatal. Cette étude suggère que des mécanismes d’épissages régulent finement l’expression de ces isoformes de Dp71 à des moments clés de la genèse et de la maturation de différents types de cellules dans le cerveau. Contact : cyrille.vaillend@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 8:51 AM
|
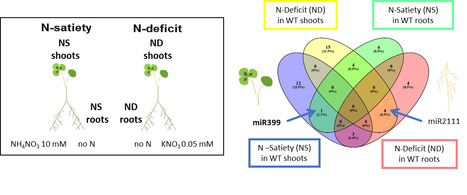
Les plantes utilisent une combinaison de voies locales et systémiques sophistiquées pour optimiser leur croissance en fonction de la disponibilité hétérogène des nutriments dans le sol. Les légumineuses peuvent acquérir de l’azote minéral (N) soit par leurs racines, soit via une interaction symbiotique avec des bactéries rhizobia fixatrices d’azote hébergées dans des nodosités racinaires. Dans un article récemment publié dans Journal of Experimental Botany, en collaboration avec Marc Lepetit (Montpellier) and Wouter Kohlen (Wageningen, The Netherlands), l'équipe de Florian Frugier s'est intéressée à identifier les signaux systémiques agissant des parties aériennes vers les racines chez les plantes de Medicago truncatula en réponse à un déficit ou à une satiété en N. Dans ce but, les plantes ont été cultivées dans un système expérimental de « split-root », dans lequel une quantité élevée ou faible de N est fournie à la moitié du système racinaire, permettant ainsi l'analyse des voies systémiques indépendamment de la réponse locale en N. Parmi les familles d’hormones végétales analysées, la cytokinine trans-Zéatine s’accumule dans les plantes en satiété en N. L'application localisée de cytokinine via un pétiole conduit à une inhibition à la fois de la croissance des racines et de la nodulation. De plus, une analyse exhaustive des miARNs a révélé que miR2111 s'accumule de manière systémique sous déficit en N à la fois dans les parties aériennes et dans les racines distantes non-traitées, alors qu'un miARN lié à l'acquisition de phosphate inorganique (Pi), le miR399, s’accumule dans les plantes cultivées en satiété en N. Ces deux profils d'accumulation dépendent de CRA2 (« Compact Root Architecture 2 »), un récepteur requis pour la signalisation des peptides CEP (« C-terminally Encoded Peptide »). L'expression ectopique constitutive du miR399 réduit le nombre de nodosités et la biomasse des racines en fonction de la disponibilité du Pi, ce qui suggère que le module de régulation de l'acquisition du Pi dépendant du miR399, contrôlé par la disponibilité en N, affecte le développement de l'ensemble du système racinaire des légumineuses. Contact : florian.frugier@universite-paris-scalay.fr

|
Scooped by
Life Sciences UPSaclay
July 9, 9:34 AM
|
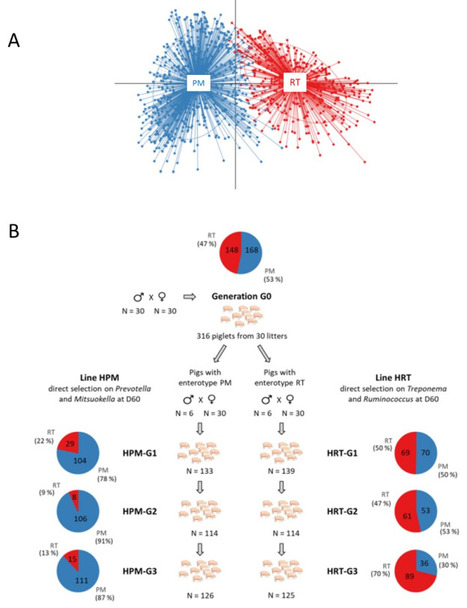
Le microbiote intestinal se construit progressivement après la naissance chez les animaux et les humains. On considère souvent que l’environnement, notamment maternel, et l’alimentation sont les principaux facteurs influençant la composition du microbiote chez les jeunes. L’importance de la génétique de l’hôte sur son microbiote est encore débattue car mal évaluée. En collaboration avec l’unité GenPhySE de Toulouse, des scientifiques INRAE des unités de Génétique Animale et Biologie Intégrative – GABI (INRAE/AgroParisTech/UPSaclay, équipe Génétique Microbiote Santé et plateforme @BRIDGe) et MetaGenoPolis de Jouy-en-Josas publient une étude dans Microbiome, qui prouve expérimentalement, chez le porc, que la formation du microbiote intestinal est en partie héritable. Ils ont mis en évidence que des porcelets âgés de 60 jours se répartissent en deux groupes selon leur type de microbiotes appelé entérotype, et produit deux lignées de porcs, chacune sélectionnée pour un entérotype. Les résultats montrent que, sur trois générations et pour un total d’environ 1000 porcs, la fréquence de chaque entérotype augmente de génération en génération dans chaque lignée. Les porcs issus des deux lignées diffèrent par leur vitesse de croissance mesurée jusqu’à 70 jours d’âge. Une analyse complémentaire a révélé que les deux entérotypes n’ont pas la même diversité microbienne et contiennent des bactéries ayant un potentiel métabolique différent. Les travaux se poursuivent pour étudier la stabilité des entérotypes au cours de la vie et leur influence sur la production, la santé et le comportement des porcs. Ils confirment l’intérêt de prendre en compte les liens entre génétique de l’hôte et microbiote intestinal, en élevage comme en médecine. Légende Figure : Répartition des porcs selon deux entérotypes appelés PM ou RT (A) et réponse à la sélection sur trois générations (B). Contact : jordi.estelle@inrae.fr ou claire.rogel-gaillard@inrae.fr

|
Scooped by
Life Sciences UPSaclay
July 8, 5:38 PM
|
Portrait Jeune Chercheuse – Hélène Quelquejay, Chercheuse en imagerie biomédicale et théranostique
Hélène Quelquejay a intégré le groupe ONCO de l’équipe « Imagerie en Oncologie » du laboratoire d’Imagerie Biomédicale Multimodale Paris Saclay (UMR BioMaps CEA/CNRS UMR9011/Inserm UMR1281/UPSaclay) au Service Hospitalier Frédéric Joliot (CEA/DRF/Joliot) à Orsay en novembre 2023, en tant qu’ingénieur-chercheur au CEA. Hélène a effectué sa thèse en biologie au Centre de Recherche Cardiovasculaire de Paris (Inserm U970) de 2015 à 2019, en collaboration avec l’Institut de Recherches Servier. Ses recherches ont porté sur le rôle vasculaire du gène L-WNK1, connu pour réguler la pression artérielle au niveau rénal, mais dont le rôle dans les cellules musculaires lisses (CML) vasculaires restait à explorer. Elle a démontré que la délétion spécifique de L-WNK1 dans les CML favorise un changement phénotypique de ces cellules, passant d’un état contractile à un état inflammatoire, ce qui provoque une aortite sévère chez la souris. Après sa thèse, Hélène a appliqué ses connaissances sur l’inflammation dans les maladies vasculaires au domaine de la radioprotection. En 2020, elle a rejoint l’Institut de Radioprotection et de Sûreté Nucléaire pour étudier l'impact du stress sur la réponse aux faibles doses de rayonnements ionisants (RI) sur le système vasculaire. Son post-doctorat de trois ans a été réalisé au sein du laboratoire de radiotoxicologie et radiobiologie expérimentale. Dans un premier temps, Hélène a développé et validé un modèle murin de stress induit par l’odeur de prédateur, puis elle a mené une étude dosimétrique après une contamination chronique à de faibles doses de césium 137 (Cs137), identifiant un dimorphisme sexuel dans l’accumulation du Cs137. Ensuite, elle a montré une altération de la capacité olfactive des souris contaminées, empêchant l’induction du stress par l’odeur de prédateur. Elle a donc étudié uniquement les effets d’une contamination chronique à de faibles doses de Cs137 sur une pathologie vasculaire, l’athérosclérose, découvrant une dose seuil à partir de laquelle les effets des RI passent de protecteurs à délétères. Son intérêt croissant pour l'interaction entre le rayonnement et le vivant, développé au fil de son cursus, l'a conduite à rencontrer des experts en radiothérapie interne vectorisée pour orienter ses recherches vers la radiobiologie. Fin 2023, elle a rejoint le groupe ONCO de Charles Truillet au laboratoire BioMaps, spécialisé dans l’imagerie du système immunitaire et des thérapies ciblées pour le cancer. Les recherches actuelles d’Hélène se concentrent sur le développement de nouveaux biomarqueurs prédictifs, notamment en radiothérapie interne ciblant le cancer du poumon, et sur la lymphopénie radio-induite. En outre, elle participe à divers projets portant sur les approches théranostiques et la radioimmunothérapie interne. « Tout seul on va plus vite, ensemble, on va plus loin ». Proverbe africain Contact : helene.quelquejay@cea.fr

|
Scooped by
Life Sciences UPSaclay
July 10, 6:20 AM
|
Les Lundis de l'IPSIT - Lundi 16 septembre 2024 : "Expérimentation animale : procédures expérimentales de la conception à l'analyse rétrospective"
Dans le cadre de l'article 5 de l'arrêté du 1er février 2013 relatif à la validation des compétences des personnels des établissements, utilisateurs d'animaux à des fins scientifiques, la SFR IPSIT organise un séminaire exceptionnel sur : Expérimentation animale : "procédures expérimentales de la conception à l'analyse rétrospective" le lundi 16 septembre 2024, de 09h15 à 12h15 Université Paris-Saclay, Bâtiment Henri Moissan 17, avenue des Sciences 91400 Orsay (AMPHI Hervé Daniel) Organisateurs : Valérie Domergue (Directrice de l'UMS IPSIT, US31, UAR3679 et Directrice technique de la plateforme AnimEx, Orsay), Grégory Merlen (Chercheur – UMR-S 1193, Physiopathogénèse et Traitement des Maladies du Foie, INSERM/UPSaclay, Orsay). Ce séminaire, ouvert à tous, est validant pour 1/2 journée dans le cadre des obligations réglementaires de formation continue à l'expérimentation animale. Pour être validée, la présence à l'entièreté du séminaire est requise. L'inscription au séminaire est GRATUITE mais OBLIGATOIRE, dans la limite des places disponibles et après validation des organisateurs, au plus tard le 9 septembre 2024, à l'adresse suivante : nadine.belzic@inserm.fr Intervenants : - Sophie PICAVET (Vétérinaire désignée Dipl. LAS, Servier, Formatrice Sévérité FELASA, réseau Cometh IdF et SBEA IdF) : Evaluation du degré de gravité des procédures (prospective et rétrospective)
- Susana GOMEZ (Vétérinaire, Inserm, responsable de la formation au FC3R) : Anesthésie et analgésie des rongeurs et lagomorphes
- Patrick GONIN (Vétérinaire, Responsable de la Plateforme Evaluation Préclinique de Gustave Roussy, président du CEEA26, Coordinateur réseau CAPSud) : Appréciation Rétrospective

|
Scooped by
Life Sciences UPSaclay
July 10, 6:04 AM
|
Nous avons le plaisir de vous convier à une rencontre pour découvrir le projet de ville nouvelle contre le cancer, Campus Grand Parc. Campus Grand Parc : Le cluster d’innovation dédié à la lutte contre le cancer Date : 8 octobre 2024
Heure : 14h00 - 17h30
Lieu : Campus Grand Parc, Villejuif Pourquoi participer ? - Découvrez le quartier Campus Grand Parc à Villejuif, et la ville nouvelle contre le cancer.
- Participez à des tables rondes inspirantes avec des acteurs clés de l'innovation en oncologie.
- Exprimez vos besoins et attentes pour rejoindre le cluster de l’innovation.
- Renforcez votre réseau lors du cocktail et des visites guidées des bâtiments Perelis, Byos et Kadans.
Programme : - 14h00 - 14h20 : Introduction par les acteurs du territoire et du cluster
- 14h20 - 15h00 : Table Ronde 1 - Vivre à Campus Grand Parc : hébergement, commerces, qualité de vie
- 15h00 - 15h45 : Table Ronde 2 – Développer des partenariats de proximité pour inventer l’oncologie de demain
- 16h15 - 17h00 : Table Ronde 3 – Faciliter l’accès au support technologique et aux infrastructures
- 17h00 - 17h15 : Conclusion
- 17h30 - 18h30 : Cocktail et visites des bâtiments
Inscription
Le PEPR « Systèmes alimentaires, microbiomes et santé (SAMS) » lance un appel à quatre chaires juniors. Site de l’appel Objectif Cet appel vise à donner les moyens à de jeunes scientifiques d’ouvrir et de diriger une équipe de recherche au sein d’un laboratoire établi en France. Les chaires devront avoir l’ambition de développer une recherche d’excellence dans le domaine du microbiome en prévention et santé humaine, et d’enrichir l’environnement et les infrastructures de recherche nationales en cours de construction dans ce domaine. Durée et financement : 48 à 54 mois Aide allouée : maximum 500 000 euros (hors frais de gestion) Déroulement : - Première phase : sélective et obligatoire. Remise d'un dossier dont l'objectif est de démontrer l'intérêt à intégrer l'environnement de recherche français (identification du laboratoire d'accueil non obligatoire).
Date limite de dépôt : 24 septembre 2024, 11 h CET - Deuxième phase : remise d’un projet de recherche en collaboration avec le laboratoire d'accueil choisi.
Date limite prévisionnelle de dépôt : février 2025. Pour toutes questions, contacter : equipe@pepr-sams.fr Pour toutes informations sur le PEPR SAMS, consulter le site : https://pepr-sams.fr/

|
Scooped by
Life Sciences UPSaclay
July 10, 6:08 AM
|
La période de soumission pour l'appel à projet de la Fondation Cœur & Recherche a débuté le 08 juillet 2024. Cette année, les quatre thèmes des projets sont : - Insuffisance Cardiaque (100 000€ de dotation)
- Mort subite (100 000€ de dotation)
- Cœur des femmes (100 000€ de dotation)
- Cœur et Obésité (100 000€ de dotation)
Notre volonté est de soutenir vos projets de recherche, améliorer la compétitivité des équipes françaises de recherche et de participer activement aux enjeux de santé publique. Pour nous soumettre votre projet, connectez-vous sur notre site Internet (soumission en ligne uniquement) : https://www.coeur-recherche.fr/
RAPPEL ! Appel à Projets Émergence 2024 de l'OI HEALTHI - Nouvelles dates
l’Objet Interdisciplinaire Health and Therapeutic Innovation (HEALTHI) de l’Université Paris-Saclay centrée sur la santé humaine et le traitement et la prévention des maladies, et rassemblant les compétences transdisciplinaires de ses 4 Graduate Schools piliers : Health and Drug Sciences (HeaDS), Life Sciences and Health (LSH), Chimie et Santé Publique lance l'Appel à Projet Émergence 2024 pour financer des projets de recherche interdisciplinaire. Ces projets débuteront partir de octobre 2024. La finalité de cet appel à projet est de promouvoir l’interdisciplinarité et la collaboration entre les équipes du périmètre de HEALTHI sur des thématiques complémentaires. Ces projets seront financés par l'OI HEALTHI pour un montant maximum de 50 k€/projet pour une période de 12 mois. Vous trouverez ci-joint le texte de cadrage et calendrier de l'appel avec ses annexes: la listes des équipes partenaires, la description des pôles et le formulaire pour le dépôt des projets de recherche. Calendrier - Le projet de recherche devra être soumis via email à l’adresse suivante : healthi@universite-paris-saclay.fr avant le 16 septembre 2024 à 17h.
- Du 17 septembre au 07 octobre 2024 : Evaluation des projets
- Mi-octobre 2024 : Publication de la liste des lauréats
- Novembre 2024 : Début des projets
Contact et informations complémentaires : anne-sophie.rossler@universite-paris-saclay.fr
Via Life Sciences UPSaclay

|
Scooped by
Life Sciences UPSaclay
July 10, 4:51 AM
|
La Société Française de Nutrition (SFN) et Synadiet (Syndicat national des compléments alimentaires) s’associent pour récompenser un travail de Thèse, fondamental, clinique ou en santé publique, dans le domaine des compléments alimentaires en décernant un Prix de Thèse d’un montant de 5 000 €. Le but de ce Prix de Thèse est d’encourager et récompenser la prise en compte des problématiques relatives aux compléments alimentaires dans les travaux de recherche en Nutrition-Santé. - Appel à Projet à télécharger : Prix de Thèse – SFN-SYNADIET – 2024
- Dossier de Candidature à adresser à : nutrition.org@gmail.com
- Deadline : 15 Juillet 2024
|



 Your new post is loading...
Your new post is loading...